- 配置文件
- 节点流
- 配置文件 2s-agcn_ntucs_joint_fsd.yaml
- MODEL 字段
- DATASET 字段
- PIPELINE 和 INFERENCE 字段
- OPTIMIZER 字段
- agcn2s.py
- graph
- 输入通道数
- 骨骼流
- Dataset 和 Pipeline
- 配置文件
- DATASET
- PIPELINE
- 源码
- skeleton.py
- skeleton_pipeline.py
- AutoPadding
- SkeletonNorm
- Iden
- SketeonModalityTransform
- 解决维度不匹配问题
- 结果融合
- ensemble.py
- test.py
前面部分内容省略,参考博文:基于 PaddleVideo 的骨骼行为识别模型 AGCN(一)
Github PaddleVideo :https://github.com/PaddlePaddle/PaddleVideo/tree/develop/paddlevideo
直接从训练脚本开始。
配置文件
注意,2s-AGCN 是双流框架,分为节点流 joint 和骨骼流 bone,所以配置文件也有两个分别用于 joint 和 bone。
训练 AGCN 模型时的配置文件是 agcn_fsd.yaml,
详解参见博文:PaddleVideo 中 agcn_fsd.yaml 配置文件代码详解
# 使用 GPU 版本
!python3.7 main.py -c configs/recognition/agcn/agcn_fsd.yaml
现在,PaddleVideo 更新了,新加入了2s-AGCN 和 CTR-GCN,但是需要自己调整配置文件,因为没有和花滑比赛数据集 fsd-10花样滑冰数据集 完全匹配的配置。
节点流
PaddleVideo/configs/recognition/agcn2s/ 文件夹下的配置文件有:
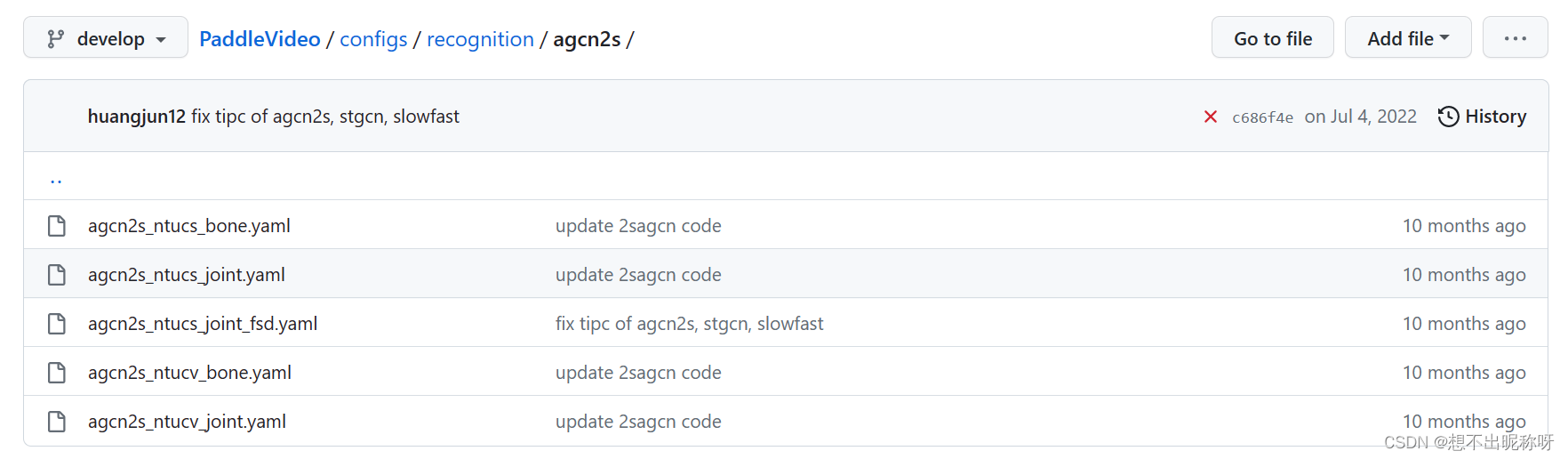
使用 2s-AGCN 进行训练,可以尝试使用 2s-agcn_ntucs_joint_fsd.yaml 配置文件作为节点流的配置。
该配置文件是用于 NTU-CS 数据集和 FSD 数据集的联合训练的,适用于多模态数据的分类任务,与使用的原始 AGCN 配置文件 agcn_fsd.yaml 类似。
将命令行中的配置文件路径改为:
configs/recognition/2s-agcn/2s-agcn_ntucs_joint_fsd.yaml
然后对 2s-agcn_ntucs_joint_fsd.yaml 稍加修改,使其用于 FSD 数据集,(.yaml 文件名也可以修改如 agcn2s_fsd_joint.yaml)即可开始 2s-AGCN 的节点流的训练。
配置文件 2s-agcn_ntucs_joint_fsd.yaml
MODEL: #MODEL field
framework: "RecognizerGCN" #Mandatory, indicate the type of network, associate to the 'paddlevideo/modeling/framework/' .
backbone: #Mandatory, indicate the type of backbone, associate to the 'paddlevideo/modeling/backbones/' .
name: "AGCN2s" #Mandatory, The name of backbone.
num_point: 25
num_person: 1
graph: "ntu_rgb_d"
graph_args:
labeling_mode: "spatial"
in_channels: 2
head:
name: "AGCN2sHead" #Mandatory, indicate the type of head, associate to the 'paddlevideo/modeling/heads'
num_classes: 60 #Optional, the number of classes to be classified.
in_channels: 64 #output the number of classes.
M: 1 #number of people.
DATASET: #DATASET field
batch_size: 64 #Mandatory, bacth size
num_workers: 4 #Mandatory, the number of subprocess on each GPU.
test_batch_size: 64
test_num_workers: 0
train:
format: "SkeletonDataset" #Mandatory, indicate the type of dataset, associate to the 'paddlevidel/loader/dateset'
file_path: "data/fsd10/FSD_train_data.npy" #Mandatory, train data index file path
label_path: "data/fsd10/FSD_train_label.npy"
test:
format: "SkeletonDataset" #Mandatory, indicate the type of dataset, associate to the 'paddlevidel/loader/dateset'
file_path: "data/fsd10/test_A_data.npy" #Mandatory, valid data index file path
test_mode: True
PIPELINE: #PIPELINE field
train: #Mandotary, indicate the pipeline to deal with the training data, associate to the 'paddlevideo/loader/pipelines/'
sample:
name: "AutoPadding"
window_size: 300
transform: #Mandotary, image transfrom operator
- SkeletonNorm:
test: #Mandotary, indicate the pipeline to deal with the training data, associate to the 'paddlevideo/loader/pipelines/'
sample:
name: "AutoPadding"
window_size: 300
transform: #Mandotary, image transfrom operator
- SkeletonNorm:
OPTIMIZER: #OPTIMIZER field
name: 'Momentum'
momentum: 0.9
learning_rate:
iter_step: True
name: 'CustomWarmupAdjustDecay'
step_base_lr: 0.1
warmup_epochs: 5
lr_decay_rate: 0.1
boundaries: [ 30, 40 ]
weight_decay:
name: 'L2'
value: 1e-4
use_nesterov: True
METRIC:
name: 'SkeletonMetric'
out_file: 'submission.csv'
INFERENCE:
name: 'STGCN_Inference_helper'
num_channels: 2
window_size: 350
vertex_nums: 25
person_nums: 1
model_name: "AGCN2s"
log_interval: 10 #Optional, the interal of logger, default:10
epochs: 50 #Mandatory, total epoch
save_interval: 10
针对 FSD 数据集,需要进行如下修改,才能与该配置文件相适配:
MODEL 字段
在 MODEL 字段中,将 num_classes 修改为 30,对应 FSD 数据集中的 30 个类别。
head:
name: "AGCN2sHead"
num_classes: 30
in_channels: 64
M: 1
这段代码定义了 AGCN2s 模型的头部,也就是最后一层网络结构,用于将经过编码器和解码器的中间表示转化为分类结果。其中:
name: "AGCN2sHead"表示使用 AGCN2s 模型的头部结构。num_classes:30表示 FSD 数据集中总共有 30 个标签类别需要分类,因此网络的最后一层输出大小为 30。in_channels: 64表示每个时间序列的输入向量的维度为 64,这个值应该根据数据集的特点进行设置,以便网络能够更好地感知时序上的不同特征。M: 1表示每个时间序列中只包含一个人物的骨架数据,因为 FSD 数据集中每个样本只包含一个人物的动作数据。
其中,in_channels: 64 是一个可以调整的参数,具体取值应该根据数据集的特点和模型的架构来进行设置。
在 AGCN2s 模型中,输入的时序数据首先会经过空间时序图卷积网络(STGCN)的多层卷积、池化和归一化操作,将 25 个节点上的特征转化为一个时序上的特征表示。然后,这个时序上的特征表示会被 送到 AGCN 模块中进行更加精细的建模操作,最终输出到头部网络中进行分类。
因此,
in_channels的取值应该考虑 STGCN 和 AGCN 模块的设计,以及数据集中时序数据的性质。例如,在使用 FSD 数据集时,可以根据实验结果确定一个相对合适的
in_channels值,一般在 64 到 256 之间选择。
- 当
in_channels取值较小时,模型可能难以捕捉复杂的时序特征。- 当
in_channels取值较大时,模型可能容易过拟合,训练时间也会增加。因此,需要在实验中尝试不同的
in_channels值,并根据实验结果来确定最佳的取值。
DATASET 字段
在 DATASET 字段中,将 train 和 test 中的 file_path 分别修改为 FSD 数据集中的训练集和测试集的路径。
train:
format: "SkeletonDataset"
file_path: "/home/aistudio/data/data104925/train_data.npy" #训练数据集路径
label_path: "/home/aistudio/data/data104925/train_label.npy" #训练数据集路径
test:
format: "SkeletonDataset"
file_path: "/home/aistudio/data/data104924/test_A_data.npy" #测试数据集路径
test_mode: True
PIPELINE 和 INFERENCE 字段
window_size: 300 是一个可以调整的参数。
window_size 用于指定自动填充补零的窗口大小(单位为帧数),由于不同视频序列的长度可能不同,因此在训练过程中需要将视频序列填充到相同的长度,以便于模型处理。
OPTIMIZER 字段
OPTIMIZER: #OPTIMIZER field
name: 'Momentum'
momentum: 0.9
learning_rate:
iter_step: True
name: 'CustomWarmupAdjustDecay'
step_base_lr: 0.1
warmup_epochs: 5
lr_decay_rate: 0.1
boundaries: [ 30, 40 ]
weight_decay:
name: 'L2'
value: 1e-4
use_nesterov: True
这段代码定义了一个优化器(optimizer),用于在训练 AGCN2s 模型时更新网络参数。
具体来说:
name: 'Momentum'表示使用动量(momentum)优化器,即 SGD with Momentum。momentum: 0.9表示设置动量系数为 0.9,这个系数表示在更新梯度时引入前一次梯度的影响程度,主要用于加速优化过程。learning_rate定义了学习率(learning rate)的调整策略,包括学习率衰减和学习率热身两个过程:name: 'CustomWarmupAdjustDecay'表示使用一个自定义的学习率调整策略,即先进行学习率热身,然后根据预定的步骤调整学习率大小,并在每个阶段结束时进行学习率衰减。step_base_lr: 0.1表示初始学习率为 0.1。warmup_epochs: 5表示设置学习率热身的轮数为 5 轮,即在前 5 轮迭代中,学习率会从很小的值逐步增加到设定的初始值。lr_decay_rate: 0.1表示设置学习率的衰减率为 0.1,即在预定的迭代轮数结束时将学习率缩小到原来的 0.1 倍。boundaries: [ 30, 40 ]表示在第 30 轮和第 40 轮结束时进行学习率调整。具体来说,将学习率按照一定比例进行缩小,并在后续训练中保持调整后的大小。
weight_decay定义了权重衰减(weight decay)的方式,即在优化过程中对参数进行正则化以避免过拟合:name: 'L2'表示使用 L2 正则化方式对网络参数进行约束。value: 1e-4表示设置 L2 正则化系数为 0.0001。
use_nesterov: True表示同时采用 Nesterov 动量(Nesterov Momentum)来加速优化过程。Nesterov 动量相比于普通动量算法,可以更好地处理优化问题中的高曲率区域,从而提升优化效果。
agcn2s.py
文件路径:PaddleVideo/paddlevideo/modeling/backbones/agcn2s.py
参见博文:2s-AGCN 代码理解
import paddle
import paddle.nn as nn
import numpy as np
from ..registry import BACKBONES
def import_class(name):
components = name.split('.')
mod = __import__(components[0])
for comp in components[1:]:
mod = getattr(mod, comp)
return mod
class UnitTCN(nn.Layer):
def __init__(self, in_channels, out_channels, kernel_size=9, stride=1):
super(UnitTCN, self).__init__()
pad = int((kernel_size - 1) / 2)
self.conv = nn.Conv2D(in_channels,
out_channels,
kernel_size=(kernel_size, 1),
padding=(pad, 0),
stride=(stride, 1))
self.bn = nn.BatchNorm2D(out_channels)
self.relu = nn.ReLU()
def forward(self, x):
" input size : (N*M, C, T, V)"
x = self.bn(self.conv(x))
return x
class UnitGCN(nn.Layer):
def __init__(self,
in_channels,
out_channels,
A,
coff_embedding=4,
num_subset=3):
super(UnitGCN, self).__init__()
inter_channels = out_channels // coff_embedding
self.inter_c = inter_channels
PA = self.create_parameter(shape=A.shape, dtype='float32')
self.PA = PA
self.A = paddle.to_tensor(A.astype(np.float32))
self.num_subset = num_subset
self.conv_a = nn.LayerList()
self.conv_b = nn.LayerList()
self.conv_d = nn.LayerList()
for i in range(self.num_subset):
self.conv_a.append(nn.Conv2D(in_channels, inter_channels, 1))
self.conv_b.append(nn.Conv2D(in_channels, inter_channels, 1))
self.conv_d.append(nn.Conv2D(in_channels, out_channels, 1))
if in_channels != out_channels:
self.down = nn.Sequential(nn.Conv2D(in_channels, out_channels, 1),
nn.BatchNorm2D(out_channels))
else:
self.down = lambda x: x
self.bn = nn.BatchNorm2D(out_channels)
self.soft = nn.Softmax(-2)
self.relu = nn.ReLU()
def forward(self, x):
N, C, T, V = x.shape
A = self.A + self.PA
y = None
for i in range(self.num_subset):
A1 = paddle.transpose(self.conv_a[i](x),
perm=[0, 3, 1,
2]).reshape([N, V, self.inter_c * T])
A2 = self.conv_b[i](x).reshape([N, self.inter_c * T, V])
A1 = self.soft(paddle.matmul(A1, A2) / A1.shape[-1])
A1 = A1 + A[i]
A2 = x.reshape([N, C * T, V])
z = self.conv_d[i](paddle.matmul(A2, A1).reshape([N, C, T, V]))
y = z + y if y is not None else z
y = self.bn(y)
y += self.down(x)
return self.relu(y)
class Block(nn.Layer):
def __init__(self, in_channels, out_channels, A, stride=1, residual=True):
super(Block, self).__init__()
self.gcn1 = UnitGCN(in_channels, out_channels, A)
self.tcn1 = UnitTCN(out_channels, out_channels, stride=stride)
self.relu = nn.ReLU()
if not residual:
self.residual = lambda x: 0
elif (in_channels == out_channels) and (stride == 1):
self.residual = lambda x: x
else:
self.residual = UnitTCN(in_channels,
out_channels,
kernel_size=1,
stride=stride)
def forward(self, x):
x = self.tcn1(self.gcn1(x)) + self.residual(x)
return self.relu(x)
# This Graph structure is for the NTURGB+D dataset. If you use a custom dataset, modify num_node and the corresponding graph adjacency structure.
class Graph:
def __init__(self, labeling_mode='spatial'):
num_node = 25
self_link = [(i, i) for i in range(num_node)]
inward_ori_index = [(1, 2), (2, 21), (3, 21), (4, 3), (5, 21), (6, 5),
(7, 6), (8, 7), (9, 21), (10, 9), (11, 10),
(12, 11), (13, 1), (14, 13), (15, 14), (16, 15),
(17, 1), (18, 17), (19, 18), (20, 19), (22, 23),
(23, 8), (24, 25), (25, 12)]
inward = [(i - 1, j - 1) for (i, j) in inward_ori_index]
outward = [(j, i) for (i, j) in inward]
neighbor = inward + outward
self.num_node = num_node
self.self_link = self_link
self.inward = inward
self.outward = outward
self.neighbor = neighbor
self.A = self.get_adjacency_matrix(labeling_mode)
def edge2mat(self, link, num_node):
A = np.zeros((num_node, num_node))
for i, j in link:
A[j, i] = 1
return A
def normalize_digraph(self, A):
Dl = np.sum(A, 0)
h, w = A.shape
Dn = np.zeros((w, w))
for i in range(w):
if Dl[i] > 0:
Dn[i, i] = Dl[i]**(-1)
AD = np.dot(A, Dn)
return AD
def get_spatial_graph(self, num_node, self_link, inward, outward):
I = self.edge2mat(self_link, num_node)
In = self.normalize_digraph(self.edge2mat(inward, num_node))
Out = self.normalize_digraph(self.edge2mat(outward, num_node))
A = np.stack((I, In, Out))
return A
def get_adjacency_matrix(self, labeling_mode=None):
if labeling_mode is None:
return self.A
if labeling_mode == 'spatial':
A = self.get_spatial_graph(self.num_node, self.self_link,
self.inward, self.outward)
else:
raise ValueError()
return A
@BACKBONES.register()
class AGCN2s(nn.Layer):
def __init__(self,
num_point=25,
num_person=2,
graph='ntu_rgb_d',
graph_args=dict(),
in_channels=3):
super(AGCN2s, self).__init__()
if graph == 'ntu_rgb_d':
self.graph = Graph(**graph_args)
else:
raise ValueError()
A = self.graph.A
self.data_bn = nn.BatchNorm1D(num_person * in_channels * num_point)
self.l1 = Block(in_channels, 64, A, residual=False)
self.l2 = Block(64, 64, A)
self.l3 = Block(64, 64, A)
self.l4 = Block(64, 64, A)
self.l5 = Block(64, 128, A, stride=2)
self.l6 = Block(128, 128, A)
self.l7 = Block(128, 128, A)
self.l8 = Block(128, 256, A, stride=2)
self.l9 = Block(256, 256, A)
self.l10 = Block(256, 256, A)
def forward(self, x):
N, C, T, V, M = x.shape
x = x.transpose([0, 4, 3, 1, 2]).reshape_([N, M * V * C, T])
x = self.data_bn(x)
x = x.reshape_([N, M, V, C,
T]).transpose([0, 1, 3, 4,
2]).reshape_([N * M, C, T, V])
x = self.l1(x)
x = self.l2(x)
x = self.l3(x)
x = self.l4(x)
x = self.l5(x)
x = self.l6(x)
x = self.l7(x)
x = self.l8(x)
x = self.l9(x)
x = self.l10(x)
return x
graph
注意 class AGCN2s 中,邻接矩阵
A
A
A 的构造是根据 graph == 'ntu_rgb_d',
num_node = 25
self_link = [(i, i) for i in range(num_node)]
inward_ori_index = [(1, 2), (2, 21), (3, 21), (4, 3), (5, 21), (6, 5), (7, 6),
(8, 7), (9, 21), (10, 9), (11, 10), (12, 11), (13, 1),
(14, 13), (15, 14), (16, 15), (17, 1), (18, 17), (19, 18),
(20, 19), (22, 23), (23, 8), (24, 25), (25, 12)]
inward = [(i - 1, j - 1) for (i, j) in inward_ori_index]
outward = [(j, i) for (i, j) in inward]
neighbor = inward + outward
其中,
- 变量
num_node表示节点数量, self_link是自环的节点列表,inward_ori_index和inward是表示从其他节点指向该节点的边的列表,outward是表示从该节点指向其他节点的边的列表,neighbor是所有边的列表。
邻接矩阵是由 self_link, inward 和 outward 构成的一个三维数组。
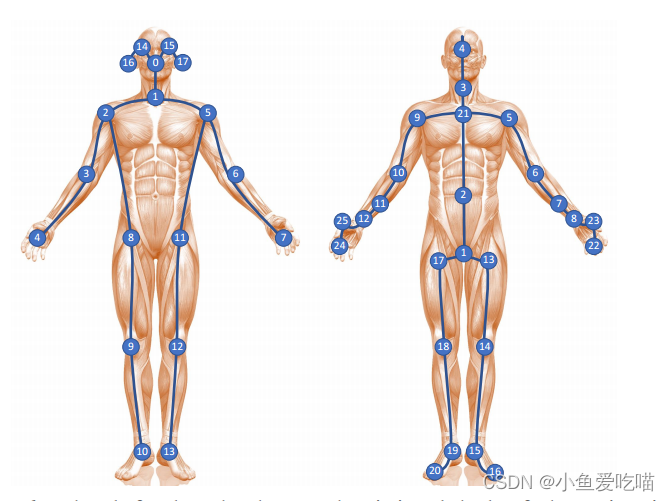
i n w a r d _ o r i _ i n d e x inward\_ori\_index inward_ori_index 是一个变量,用于存储 每个节点与向心节点的连接对。向心节点是指 从其他节点指向该节点的节点。例如, ( 1 , 2 ) (1, 2) (1,2) 表示节点1与节点2相连,且节点2是向心节点。这个变量是用于创建 NTU RGB-D 数据集对应的图结构的,其中每个节点代表一个人体关节,每条边代表一个人体骨骼。
i n w a r d _ o r i _ i n d e x inward\_ori\_index inward_ori_index 是根据下图定义的,左图显示了 Kinetics-Skeleton 数据集的关节标签,右图显示了 NTU-RGBD 数据集的关节标签(21是中心关节)。
NTU-RGBD 数据集的关节标签(21是中心关节),但是 fsd-10花样滑冰数据集中,8号索引关键点为人体中心。要做如下修改:
self.num_node = 25
self_link = [(i, i) for i in range(self.num_node)]
inward_ori_index = [(1, 8), (0, 1), (15, 0), (17, 15), (16, 0),
(18, 16), (5, 1), (6, 5), (7, 6), (2, 1), (3, 2),
(4, 3), (9, 8), (10, 9), (11, 10), (24, 11),
(22, 11), (23, 22), (12, 8), (13, 12), (14, 13),
(21, 14), (19, 14), (20, 19)]
修改如上后,可以把 2s-agcn_ntucs_joint_fsd.yaml 另存为 2s-agcn_fsd_joint.yaml 表示用于 fsd 的节点流配置文件。
输入通道数
self.data_bn = nn.BatchNorm1D(num_person * in_channels * num_point)
原来的 NTU 数据集是 25 num_point * 3 in_channels * 1 num_person = 75 输入通道数,但是现在 FSD 数据集是 25 num_point * 2 in_channels * 1 num_person = 50 输入通道数,注意在配置文件中修改 num_person、in_channels 和 num_point 即可。
骨骼流
刚开始只改了配置文件中的 num_person、in_channels 和 num_point 参数,但是运行时候遇到了问题:
File "/home/aistudio/work/FigureSkating/paddlevideo/modeling/backbones/agcn2s.py", line 218, in forward
x = self.data_bn(x)
ValueError: (InvalidArgument) ShapeError: the shape of scale must equal to [75]But received: the shape of scale is [50]
[Hint: Expected scale_dim[0] == C, but received scale_dim[0]:50 != C:75.] (at /paddle/paddle/phi/infermeta/multiary.cc:593)
问题出在这一行代码 x = self.data_bn(x),报错提示预期的维度是 [75],但是我的输入通道数是 [50](同上节点流中的 self.data_bn = nn.BatchNorm1D(num_person * in_channels * num_point))。
那我就好奇了,为什么节点流的可以运行,到了骨骼流就不行了,于是我打印了相关数据,
def forward(self, x):
N, C, T, V, M = x.shape
print(N,C,T,V,M) # 打印
x = x.transpose([0, 4, 3, 1, 2]).reshape_([N, M * V * C, T])
x = self.data_bn(x)
...
发现,节点流的输出是:64 2 350 25 1,骨骼流的输出是:64 3 2500 25 1。
- 64 很容易理解,因为
batch_size: 64所以每个批次都是 64 个样本。 - 节点流的从
3 2500变成2 350,猜测是因为在 Dataset 和 Pipeline 部分做了某些变换:去除了(x, y, conf)中的conf置信度,所以只保留了节点坐标(x, y)两个维度。2500帧只保留350帧,这和window_size: 350保持一致。
那么,为什么骨骼流没有发生变换呢,那得去 Dataset 和 Pipeline 源码看看。
Dataset 和 Pipeline
配置文件
DATASET
DATASET: #DATASET field
batch_size: 64 #Mandatory, bacth size
num_workers: 4 #Mandatory, the number of subprocess on each GPU.
test_batch_size: 1
test_num_workers: 0
train:
format: "SkeletonDataset"
file_path: "/home/aistudio/work/dataset/train_data.npy"
label_path: "/home/aistudio/work/dataset/train_label.npy"
...
这是节点流的 DATASET 配置,骨骼流的 DATASET 配置也一模一样。
batch_size: 64,这和输出中的N的大小是 64 一致,说明每个批次确实是 64 个样本。format: "SkeletonDataset"说明训练数据集的格式是SkeletonDataset。
在训练模型时,通常需要 从数据集中读取数据进行训练,不同的数据集可能有不同的格式。
PIPELINE
PIPELINE: #PIPELINE field
train:
sample:
name: "AutoPadding"
window_size: 350
transform: #Mandotary, image transfrom operator
- SkeletonNorm:
...
这是节点流的 PIPELINE 配置,采用了 AutoPadding 样本处理器和 SkeletonNorm 数据变换。
PIPELINE: #PIPELINE field
train:
sample:
- Iden:
transform: #Mandotary, image transfrom operator
- SketeonModalityTransform:
joint: False
bone: True
motion: False
graph: 'fsd'
...
这是骨骼流的 PIPELINE 配置,采用了 Iden 样本处理器和 SketeonModalityTransform 数据变换。
源码
skeleton.py
文件路径:/paddlevideo/loader/dataset/skeleton.py
import os.path as osp
import copy
import random
import numpy as np
import pickle
from ..registry import DATASETS
from .base import BaseDataset
from ...utils import get_logger
logger = get_logger("paddlevideo")
@DATASETS.register()
class SkeletonDataset(BaseDataset):
"""
Skeleton dataset for action recognition.
The dataset loads skeleton feature, and apply norm operatations.
Args:
file_path (str): Path to the index file.
pipeline(obj): Define the pipeline of data preprocessing.
data_prefix (str): directory path of the data. Default: None.
test_mode (bool): Whether to bulid the test dataset. Default: False.
"""
def __init__(self, file_path, pipeline, label_path=None, test_mode=False):
self.label_path = label_path
super().__init__(file_path, pipeline, test_mode=test_mode)
def load_file(self):
"""Load feature file to get skeleton information."""
logger.info("Loading data, it will take some moment...")
self.data = np.load(self.file_path)
if self.label_path:
if self.label_path.endswith('npy'):
self.label = np.load(self.label_path)
elif self.label_path.endswith('pkl'):
with open(self.label_path, 'rb') as f:
sample_name, self.label = pickle.load(f)
else:
logger.info(
"Label path not provided when test_mode={}, here just output predictions."
.format(self.test_mode))
logger.info("Data Loaded!")
return self.data # used for __len__
def prepare_train(self, idx):
"""Prepare the feature for training/valid given index. """
results = dict()
results['data'] = copy.deepcopy(self.data[idx])
results['label'] = copy.deepcopy(self.label[idx])
results = self.pipeline(results)
return results['data'], results['label']
def prepare_test(self, idx):
"""Prepare the feature for test given index. """
results = dict()
results['data'] = copy.deepcopy(self.data[idx])
if self.label_path:
results['label'] = copy.deepcopy(self.label[idx])
results = self.pipeline(results)
return results['data'], results['label']
else:
results = self.pipeline(results)
return [results['data']]
这段代码定义了一个名为 SkeletonDataset 的类,它继承了 BaseDataset 这个基类。这个类的作用是为了 实现骨架数据集的动作识别,即从3D骨架关节数据序列中识别人类的动作。
这个类有以下几个参数:
file_path (str):数据文件的路径。pipeline(obj):定义 数据预处理的流程。label_path (str):标签文件的路径。默认为 None。test_mode (bool):是否构建测试数据集。默认为 False。
这个类有以下几个方法:
__init__(self, file_path, pipeline, label_path=None, test_mode=False):这是类的 构造函数,用于 初始化类的属性和调用基类的构造函数。load_file(self):这是一个 加载数据文件 的方法,用于 获取骨架信息。
- 它会打印一条日志信息
"Data Loaded!",然后从file_path中 加载数据 到self.data中。- 如果提供了
label_path,它会根据文件后缀名是npy还是pkl来 加载标签 到self.label中。- 如果没有提供
label_path,它会打印一条日志信息,表示只输出预测结果。- 最后它会返回
self.data作为数据集的长度。
prepare_train(self, idx):这是一个 准备训练/验证数据 的方法。
- 给定索引
idx,它会创建一个字典results,然后将self.data[idx]和self.label[idx]分别复制到results['data']和results['label']中。- 然后它会调用
pipeline对results进行预处理,并返回results['data']和results['label']作为训练/验证数据。
prepare_test(self, idx):这是一个 准备测试数据 的方法。
- 给定索引
idx,它会创建一个字典results,然后将self.data[idx]复制到results['data']中。- 如果提供了
label_path,它会将self.label[idx]复制到results['label']中。- 然后它会调用
pipeline对results进行预处理,并返回results['data']和results['label']作为测试数据。- 如果没有提供
label_path,它会只返回[results['data']]作为测试数据。
总结:
DATASET 中加载了文件中的数据到 self.data 和 self.label 变量中,然后根据 idx 取出训练集和验证集数据,并对其进行 results = self.pipeline(results) 操作后,作为模型训练的输入。
skeleton_pipeline.py
文件路径:/paddlevideo/loader/pipelines/skeleton_pipeline.py
AutoPadding
import os
import numpy as np
import paddle.nn.functional as F
import random
import paddle
from ..registry import PIPELINES
@PIPELINES.register()
class AutoPadding(object):
"""
Sample or Padding frame skeleton feature.
Args:
window_size: int, temporal size of skeleton feature.
random_pad: bool, whether do random padding when frame length < window size. Default: False.
"""
def __init__(self, window_size, random_pad=False):
self.window_size = window_size
self.random_pad = random_pad
def get_frame_num(self, data):
C, T, V, M = data.shape
for i in range(T - 1, -1, -1):
tmp = np.sum(data[:, i, :, :])
if tmp > 0:
T = i + 1
break
return T
def __call__(self, results):
data = results['data']
C, T, V, M = data.shape
T = self.get_frame_num(data)
if T == self.window_size:
data_pad = data[:, :self.window_size, :, :]
elif T < self.window_size:
begin = random.randint(
0, self.window_size - T) if self.random_pad else 0
data_pad = np.zeros((C, self.window_size, V, M))
data_pad[:, begin:begin + T, :, :] = data[:, :T, :, :]
else:
if self.random_pad:
index = np.random.choice(
T, self.window_size, replace=False).astype('int64')
else:
index = np.linspace(0, T, self.window_size).astype("int64")
data_pad = data[:, index, :, :]
results['data'] = data_pad
return results
这段代码定义了一个名为 AutoPadding 的类,它继承了 object 这个基类。这个类的作用是为了 对骨架特征进行采样或填充,使其具有相同的时间长度。
类的参数:
window_size: int,骨架特征的帧数。random_pad: bool,当帧长度小于window_size时,是否进行随机填充。默认为 False。
类的方法:
__init__(self, window_size, random_pad=False):这是类的构造函数,用于初始化类的属性。get_frame_num(self, data):这是一个 获取有效帧数 的方法,给定数据data。它会 从后往前遍历数据的时间维度,找到 第一个非零帧,然后返回其 索引加一 作为 有效帧数T。__call__(self, results):这是一个 对数据进行采样或填充 的方法。给定字典results,它会从results中获取数据data,并获取其有效帧数T。
- 如果
T等于window_size,它会直接返回数据的前window_size帧作为data_pad。- 如果
T小于window_size,它会创建一个全零数组data_pad,并根据random_pad参数决定在哪个位置开始将数据的前T帧复制到data_pad中。- 如果
T大于window_size,它会根据random_pad参数决定从数据中随机或均匀地选择window_size个帧作为data_pad。- 最后它会将
data_pad赋值给results['data']并返回results。
所以节点流的数据 results['data'] = data_pad 经过这个处理后,都是 350 帧。
SkeletonNorm
@PIPELINES.register()
class SkeletonNorm(object):
"""
Normalize skeleton feature.
Args:
aixs: dimensions of vertex coordinate. 2 for (x,y), 3 for (x,y,z). Default: 2.
"""
def __init__(self, axis=2, squeeze=False):
self.axis = axis
self.squeeze = squeeze
def __call__(self, results):
data = results['data']
# Centralization
data = data - data[:, :, 8:9, :]
data = data[:self.axis, :, :, :] # get (x,y) from (x,y, acc)
C, T, V, M = data.shape
if self.squeeze:
data = data.reshape((C, T, V)) # M = 1
results['data'] = data.astype('float32')
if 'label' in results:
label = results['label']
results['label'] = np.expand_dims(label, 0).astype('int64')
return results
这段代码定义了一个名为 SkeletonNorm 的类,它继承了 object 这个基类。这个类的作用是为了 对骨架特征进行归一化处理。
类的参数:
axis: int,顶点坐标的维度。2表示(x,y),3表示(x,y,z)。默认为2。squeeze: bool,是否将数据的最后一个维度压缩。默认为 False。
类的方法:
__call__(self, results):这是 对数据进行归一化 的方法。
- 给定字典
results,它会从results中获取数据data,并对其进行 中心化处理,即减去第9个顶点(鼻子)的坐标。- 然后它会根据
axis参数选择前两个或三个维度作为顶点坐标,忽略加速度信息。- 接着它会获取数据的形状
C, T, V, M,并根据squeeze参数决定是否将数据的最后一个维度压缩(当M=1时)。- 最后它会将数据转换为 float32 类型并赋值给
results['data']并返回results。- 如果
results中有'label'键,它还会将标签扩展一个维度并转换为int64类型并赋值给results['label']。
这一句代码 data = data[:self.axis, :, :, :] # get (x,y) from (x,y, acc) 就是只取 (x, y) 坐标而 去除了置信度。
Iden
@PIPELINES.register()
class Iden(object):
"""
Wrapper Pipeline
"""
def __init__(self, label_expand=True):
self.label_expand = label_expand
def __call__(self, results):
data = results['data']
results['data'] = data.astype('float32')
if 'label' in results and self.label_expand:
label = results['label']
results['label'] = np.expand_dims(label, 0).astype('int64')
return results
这段代码定义了一个名为 Iden 的类,它继承了 object 这个基类。这个类的作用是为了 包装流水线处理。
类的参数:
label_expand: bool,是否 对标签进行扩展维度。默认为 True。
类的方法:
__call__(self, results):这是一个 对数据进行包装 的方法。
给定字典
results,它会从results中获取数据data,并将其转换为float32类型并赋值给results['data']。
- 如果
results中有'label'键,并且label_expand参数为 True,它还会 将标签扩展一个维度 并转换为 int64 类型并赋值给results['label']。- 最后返回
results。
对标签进行扩展维度 的目的是为了 使标签的形状与数据的形状一致,方便后续的处理。
例如,如果数据的形状是
(C, T, V),而标签的形状是(1),那么对标签进行扩展维度后,标签的形状就变成了(1, 1, 1)。这样就可以将数据和标签拼接在一起,形成一个(C+1, T, V)的数组。
对标签进行扩展维度的方法是使用 numpy 的 expand_dims 函数,指定要扩展的轴。例如,如果要在第0轴扩展一个维度,可以写成 np.expand_dims(label, 0)。
SketeonModalityTransform
@PIPELINES.register()
class SketeonModalityTransform(object):
"""
Sketeon Crop Sampler.
Args:
crop_model: str, crop model, support: ['center'].
p_interval: list, crop len
window_size: int, sample windows size.
"""
def __init__(self, bone, motion, joint=True, graph='fsd'): # 改为 fsd
self.joint = joint
self.bone = bone
self.motion = motion
self.graph = graph
if self.graph == "fsd":
self.bone_pairs = ((1, 8), (0, 1), (15, 0), (17, 15), (16, 0),
(18, 16), (5, 1), (6, 5), (7, 6), (2, 1), (3, 2),
(4, 3), (9, 8), (10, 9), (11, 10), (24, 11),
(22, 11), (23, 22), (12, 8), (13, 12), (14, 13),
(21, 14), (19, 14), (20, 19))
else:
raise NotImplementedError
def __call__(self, results):
if self.joint:
return results
data_numpy = results['data']
if self.bone:
bone_data_numpy = np.zeros_like(data_numpy)
for v1, v2 in self.bone_pairs:
bone_data_numpy[:, :, v1 -
1] = data_numpy[:, :, v1 -
1] - data_numpy[:, :, v2 - 1]
data_numpy = bone_data_numpy
if self.motion:
data_numpy[:, :-1] = data_numpy[:, 1:] - data_numpy[:, :-1]
data_numpy[:, -1] = 0
results['data'] = data_numpy
return results
这段代码定义了一个名为 SketeonModalityTransform 的类,它继承了 object 这个基类。这个类的作用是为了 对骨架特征进行不同的变换,如骨架、运动和图结构。
类的参数:
bone: bool,是否对骨架特征进行 骨架变换,即 将每个顶点的坐标减去其连接的另一个顶点的坐标。默认为 False。motion: bool,是否对骨架特征进行 运动变换,即 将每个时间步的坐标减去前一个时间步的坐标。默认为 False。joint: bool,是否 保持原始的骨架特征不变。默认为 True。graph: str,选择使用的 图结构,默认为 ‘fsd’。
类的方法:
__call__(self, results):这是一个 对数据进行变换 的方法。
- 给定字典
results,它会从results中获取数据data_numpy,并根据bone参数决定是否进行 骨架变换。
- 如果进行骨架变换,它会创建一个全零数组
bone_data_numpy,并根据self.bone_pairs中定义的 骨架连接关系,计算每个顶点与其连接顶点的差值,并赋值给bone_data_numpy。- 然后它会将
bone_data_numpy赋值给data_numpy。
- 接着它会根据
motion参数决定是否进行 运动变换。
- 如果进行运动变换,它会将
data_numpy中 除了最后一帧之外的每一帧减去前一帧,并将最后一帧置零。
- 最后它会将
data_numpy赋值给results['data']并返回results。
所以这里没有进行 data = data[:self.axis, :, :, :] # get (x,y) from (x,y, acc) 操作。
那这个是给 NTU-RGB 数据集适配的,所以就要去看看 NTU 数据集的节点坐标是怎样的。NTU 数据集是(x, y, z)三维的坐标,那就不能只是简单地把配置文件中的 in_channels: 3 改成 in_channels: 2,还要在 SketeonModalityTransform 类中加上 data = data[:self.axis, :, :, :] # get (x,y) from (x,y, acc),去掉置信度后的输入才是真正的二维。
解决维度不匹配问题
至此,找到问题所在了,把上面的 SketeonModalityTransform 类按如下修改。
data_numpy = results['data']
# print(data_numpy.shape) # (3, 2500, 25, 1)
data_numpy = data_numpy[:2, :, :, :] # get (x,y) from (x,y, acc)
# print(data_numpy.shape) # (2, 2500, 25, 1)
if self.bone:
bone_data_numpy = np.zeros_like(data_numpy)
for v1, v2 in self.bone_pairs:
bone_data_numpy[:, :, v1 -
1] = data_numpy[:, :, v1 -
1] - data_numpy[:, :, v2 - 1]
data_numpy = bone_data_numpy
报错内存溢出:
ResourceExhaustedError:
Out of memory error on GPU 0.
Cannot allocate 976.562500MB memory on GPU 0,
31.607422GB memory has been allocated and available memory is only 144.500000MB.
我把 batch_size: 64 改成 batch_size: 32 还是溢出,不过需要的内存更少了,说明是有效的:
ResourceExhaustedError:
Out of memory error on GPU 0.
Cannot allocate 488.281250MB memory on GPU 0,
31.472656GB memory has been allocated and available memory is only 282.500000MB.
改成 batch_size: 16 后终于开始训练了:
16 2 2500 25 1
[05/23 18:24:50] epoch:[ 1/90 ] train step:0 loss: 6.33603 lr: 0.020000 top1: 0.06250 top5: 0.06250 batch_cost: 3.76629 sec, reader_cost: 0.36598 sec, ips: 4.24822 instance/sec, eta: 13:44:45
总之,经历了这样的过程:
N, C, T, V, M = x.shape
print(N,C,T,V,M)
# 64 3 2500 25 1 (维度不匹配) -> 64 2 2500 25 1 (内存溢出)-> 16 2 2500 25 1(成了)
但是,运行完第一个 epoch 后,新 bug 又出现了。
File "/home/aistudio/work/FigureSkating/paddlevideo/solver/custom_lr.py", line 322, in step
self.last_epoch += 1 / self.num_iters # update step with iters
TypeError: unsupported operand type(s) for /: 'int' and 'NoneType'
问题定位,
class CustomWarmupAdjustDecay(LRScheduler):
r"""
We combine warmup and stepwise-cosine which is used in slowfast model.
Args:
step_base_lr (float): start learning rate used in warmup stage.
warmup_epochs (int): the number epochs of warmup.
lr_decay_rate (float|int, optional): base learning rate decay rate.
step (int): step in change learning rate.
last_epoch (int, optional): The index of last epoch. Can be set to restart training. Default: -1, means initial learning rate.
verbose (bool, optional): If ``True``, prints a message to stdout for each update. Default: ``False`` .
Returns:
``CosineAnnealingDecay`` instance to schedule learning rate.
"""
def __init__(self,
step_base_lr,
warmup_epochs,
lr_decay_rate,
boundaries,
num_iters=None,
last_epoch=-1,
verbose=False):
self.step_base_lr = step_base_lr
self.warmup_epochs = warmup_epochs
self.lr_decay_rate = lr_decay_rate
self.boundaries = boundaries
self.num_iters = num_iters
#call step() in base class, last_lr/last_epoch/base_lr will be update
super(CustomWarmupAdjustDecay, self).__init__(last_epoch=last_epoch,
verbose=verbose)
def step(self, epoch=None):
"""
``step`` should be called after ``optimizer.step`` . It will update the learning rate in optimizer according to current ``epoch`` .
The new learning rate will take effect on next ``optimizer.step`` .
Args:
epoch (int, None): specify current epoch. Default: None. Auto-increment from last_epoch=-1.
Returns:
None
"""
if epoch is None:
if self.last_epoch == -1:
self.last_epoch += 1
else:
self.last_epoch += 1 / self.num_iters # update step with iters
else:
self.last_epoch = epoch
self.last_lr = self.get_lr()
if self.verbose:
print('Epoch {}: {} set learning rate to {}.'.format(
self.last_epoch, self.__class__.__name__, self.last_lr))
上述代码中,self.num_iters 初始化为 None,且后面没有赋其他值就用了 self.last_epoch += 1 / self.num_iters,才导致了上面的报错。
但是 joint 流的配置是可以跑通的,于是我仔细对比了两个流的 OPTIMIZER 的配置。
OPTIMIZER: #OPTIMIZER field
name: 'Momentum'
momentum: 0.9
learning_rate:
iter_step: True
name: 'CustomWarmupAdjustDecay'
step_base_lr: 0.1
warmup_epochs: 5
lr_decay_rate: 0.1
boundaries: [ 30, 40 ]
weight_decay:
name: 'L2'
value: 1e-4
use_nesterov: True
发现,原来 bone 的配置中少了 iter_step: True,加上后继续跑了。
iter_step: True 是一个配置选项,用于指定 是否在每个迭代步骤中更新学习率。
至此,骨骼流也跑通了。
结果融合
PaddleVideo 没有提供双流的结果融合,需要自行添加 ensemble.py,然后执行 !python3.7 ensemble.py 命令。
ensemble.py
import os
import re
import numpy as np
import csv
def softmax(X):
m = np.max(X, axis=1, keepdims=True)
exp_X = np.exp(X - m)
exp_X = np.exp(X)
prob = exp_X / np.sum(exp_X, axis=1, keepdims=True)
return prob
output_prob = None
folder = './logits'
for logits_file in os.listdir(folder):
logits = np.load(os.path.join(folder, logits_file))
prob = softmax(logits)
if output_prob is None:
output_prob = prob
else:
output_prob = output_prob + prob
pred = np.argmax(output_prob, axis=1)
with open('./submission_ensemble.csv', 'w') as f:
writer = csv.writer(f)
writer.writerow(('sample_index', 'predict_category'))
for i, p in enumerate(pred):
writer.writerow((i, p))
这段代码是从一个文件夹中读取多个 logits 文件,对每个 logits 文件应用 softmax 函数,得到一个概率矩阵,然后将所有概率矩阵相加,得到一个输出概率矩阵。最后,对输出概率矩阵按行取最大值的索引,作为预测类别,写入一个 csv 文件中。
具体来说:
softmax函数,它接受一个二维数组 X X X 作为输入,沿着第二个维度(即每一行)计算每个元素的指数值,然后除以每一行的指数和,得到一个归一化的概率矩阵。- 接着初始化了一个空的输出概率矩阵
output_prob。 - 接着指定了一个文件夹的路径,假设该文件夹中存放了多个
logits文件。 - 然后遍历该文件夹中的每个
logits文件,使用numpy.load函数读取文件内容,得到一个二维数组logits,然后调用softmax函数对其进行处理,得到一个概率矩阵prob。如果output_prob为空,则将prob赋值给output_prob;否则将prob与output_prob相加,并更新output_prob。 - 接着对输出概率矩阵按行取最大值的索引,得到一个一维数组
pred,表示预测类别。 - 最后使用
csv模块创建一个csv文件,并写入表头和数据。表头包含两列:sample_index和predict_category。数据包含每个样本的索引和预测类别。
logits文件是一种存储了 模型输出的未归一化概率 的文件。通常是由某种机器学习算法或框架生成的,可以用于计算softmax函数或交叉熵损失等操作,或者用于评估模型的性能。
test.py
文件路径:/paddlevideo/tasks/test.py。
想要得到上面的 logits 文件,还要相应修改 test.py 中的代码,让模型在测试的过程中生成 logits 文件。
import paddle
from paddlevideo.utils import get_logger
from ..loader.builder import build_dataloader, build_dataset
from ..metrics import build_metric
from ..modeling.builder import build_model
from paddlevideo.utils import load
import numpy as np
import os
import paddle.nn.functional as F
logger = get_logger("paddlevideo")
@paddle.no_grad()
def test_model(cfg, weights, parallel=True):
"""Test model entry
Args:
cfg (dict): configuration.
weights (str): weights path to load.
parallel (bool): Whether to do multi-cards testing. Default: True.
"""
# 1. Construct model.
if cfg.MODEL.backbone.get('pretrained'):
cfg.MODEL.backbone.pretrained = '' # disable pretrain model init
model = build_model(cfg.MODEL)
if parallel:
model = paddle.DataParallel(model)
# 2. Construct dataset and dataloader.
cfg.DATASET.test.test_mode = True
dataset = build_dataset((cfg.DATASET.test, cfg.PIPELINE.test))
batch_size = cfg.DATASET.get("test_batch_size", 8)
places = paddle.set_device('gpu')
# default num worker: 0, which means no subprocess will be created
num_workers = cfg.DATASET.get('num_workers', 0)
num_workers = cfg.DATASET.get('test_num_workers', num_workers)
dataloader_setting = dict(batch_size=batch_size,
num_workers=num_workers,
places=places,
drop_last=False,
shuffle=False)
data_loader = build_dataloader(dataset, **dataloader_setting)
model.eval()
state_dicts = load(weights)
model.set_state_dict(state_dicts)
# add params to metrics
cfg.METRIC.data_size = len(dataset)
cfg.METRIC.batch_size = batch_size
print('{} inference start!!!'.format(cfg.model_name))
Metric = build_metric(cfg.METRIC)
ans = np.zeros((len(data_loader), 30))
for batch_id, data in enumerate(data_loader):
outputs = model(data, mode='test')
ans[batch_id, :] = outputs
Metric.update(batch_id, data, outputs)
os.makedirs('logits', exist_ok=True)
with open('logits/{}.npy'.format(cfg.model_name), 'wb') as f:
np.save(f, ans)
print('{} inference finished!!!'.format(cfg.model_name))
Metric.accumulate()
这段代码的目的是 测试一个模型在一个数据集上的性能,具体来说:
test函数三个参数:cfg是一个配置字典,包含了模型、数据集、处理流程和评估指标的相关设置;weights是一个字符串,表示 要加载的模型权重的路径(通常是训练出的best模型权重);parallel是一个布尔值,表示是否使用多卡进行测试,默认为 True。- 根据配置字典中的
MODEL部分,构建一个模型对象,并根据parallel参数决定是否使用paddle.DataParallel进行多卡同步。 - 根据配置字典中的
DATASET和PIPELINE部分,构建一个测试数据集和一个数据加载器。数据加载器的一些参数,如batch_size、num_workers、places等,也可以从配置字典中获取或设置默认值。 - 将模型 设置为评估模式,不进行梯度更新。
- 使用
load函数从weights路径 加载模型权重,并使用model.set_state_dict方法 将权重赋值给模型。 - 根据配置字典中的
METRIC部分,构建一个评估指标对象,并将数据集的大小和批次大小作为参数传入。 - 初始化一个零矩阵
ans,用于 存储模型输出的logits。 - 遍历数据加载器中的 每个批次的数据,将数据输入模型,得到输出
logits,并将其存入ans矩阵中。同时,调用评估指标对象的update方法,更新评估结果。 - 创建一个
logits文件夹,并将ans矩阵保存为一个npy文件,文件名为模型名称。 - 打印一条信息,表示测试完成。
- 调用评估指标对象的
accumulate方法,计算并打印最终的评估结果。



![[SpringBoot]Knife4j框架Knife4j的显示内容的配置](https://img-blog.csdnimg.cn/dfabe1d4d96b41b0b32d3c62dc99f02d.png)