rWCVP入门
- 介绍
- 1. 访问到WCVP
- 1.1 方法一
- 1.2 方法二(谨慎)
- 2. WCVP数据筛选
- 2.1 关于按分类单元筛选的说明
- 2.2 关于按分布区域筛选的说明
- 笔者实践
介绍
世界维管植物名录(WCVP)是维管植物物种的全球共识。它提供了科学已知的> 340,000 种维管植物物种的名称、同义词、分类学和分布。
作者们开发了 rWCVP,使一些使用 WCVP 的常见任务更容易。其中包括根据WCVP标准化分类单元名称列表,获取和绘制物种分布图,以及创建在特定区域发现的分类单元清单。
1. 访问到WCVP
要使 rWCVP 中的函数正常工作,您需要有权访问 WCVP 的副本。
1.1 方法一
加载 WCVP 的一种方法是安装关联的数据包 rWCVPdata:
if (!require(rWCVPdata)) {
install.packages("rWCVPdata",
repos = c(
"https://matildabrown.github.io/drat",
"https://cloud.r-project.org"
)
)
}
==等待下载安装完成后,==可以看到rWCVPdata的类群和分布数据:
library(rWCVPdata)
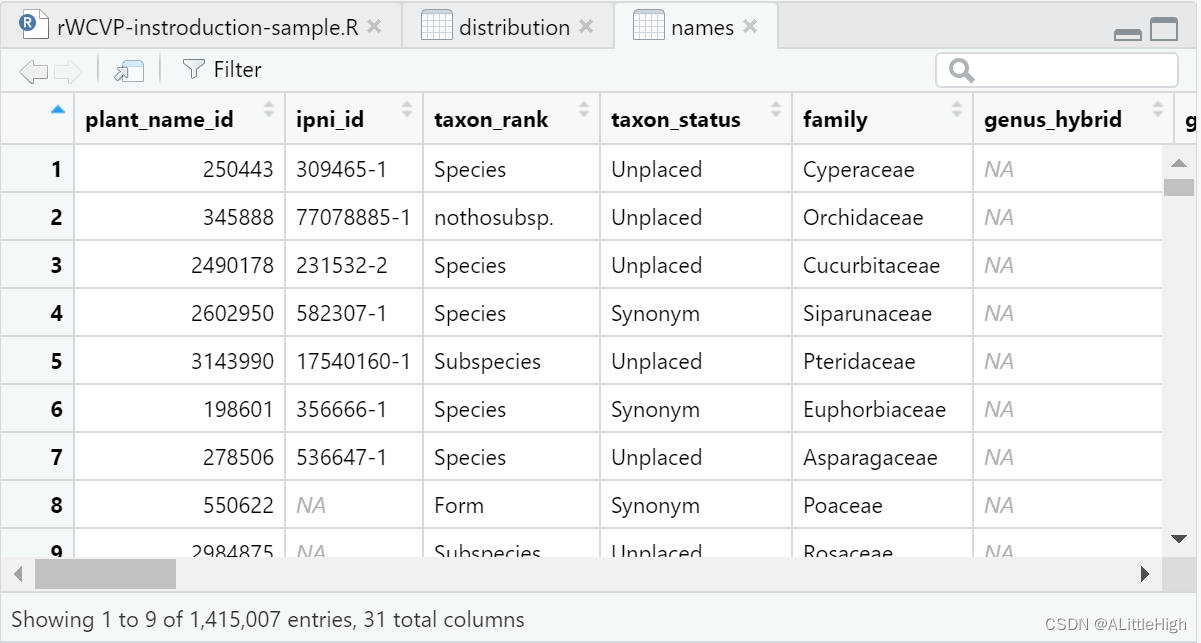
names = rWCVPdata::wcvp_names
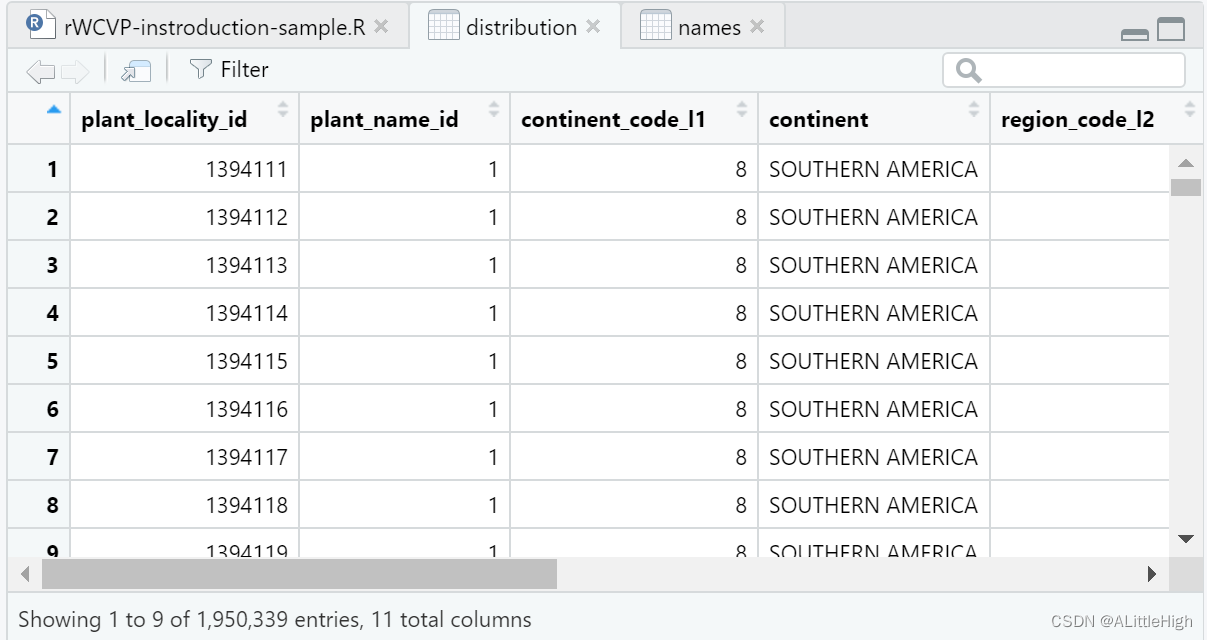
distribution = rWCVPdata::wcvp_distributions
rWCVPdata含有超过140万条物种信息:
rWCVPdata含有超过190万条物种分布信息:
1.2 方法二(谨慎)
如果数据包不可用,或者您希望使用不同版本的 WCVP,则可以向此包中的主函数提供数据的本地副本。例如,要生成清单:
names <- read_csv("/path/to/wcvp_names.csv")
distributions <- read_csv("/path/to/wcvp_distributions.csv")
checklist <- wcvp_checklist("Acacia",
taxon_rank = "genus", area_codes = "CPP",
wcvp_names = names, wcvp_distributions = distributions
)
如果您使用的是自己的WCVP版本,请小心!WCVP 表的结构有时会因版本而异。应将 rWCVP 设置为与最新版本的 WCVP 以及共享相同结构的任何先前版本一起使用。
2. WCVP数据筛选
一些rWCVP功能涉及WCVP数据筛选,以生成特定区域中维管植物物种的列表或摘要。
这些函数接受两个用于筛选 WCVP 的参数:
- taxon:具有物种或更高物种分类等级的有效分类单元的名称(例如物种“Myrcia almasensis”,属“Myrcia”或“Myrtaceae”科)。
- area:要关注的区域的 WGSRPD 3 级代码向量。
这些参数可以在wcvp_checklist、wcvp_occ_mat和wcvp_summary中组合使用,以产生所需区域中焦点分类群的输出。例如,巴西的桃金娘科:
# filter Myrtaceae species in Brazil
checklist <- wcvp_checklist("Myrtaceae",
taxon_rank = "family",
area_codes = c("BZC", "BZN", "BZS", "BZE", "BZL")
)

2.1 关于按分类单元筛选的说明
按分类筛选时,需要使用 taxon.rank 参数告诉函数您提供的名称的分类等级
例如,生成早熟禾属的摘要表:
# summary table for Poa
wcvp_summary("Poa", taxon_rank = "genus")
ℹ No area specified. Generating global summary.
$Taxon
[1] "Poa"
$Area
[1] "the world"
$Grouping_variable
[1] "area_code_l3"
$Total_number_of_species
[1] 573
$Number_of_regionally_endemic_species
[1] 573
$Summary
# A tibble: 280 × 6
area_code_l3 Native Endemic Introduced Extinct Total
<chr> <int> <int> <int> <int> <int>
1 ABT 21 0 5 0 26
2 AFG 23 1 0 0 23
3 AGE 13 2 5 0 18
4 AGS 24 0 10 0 34
5 AGW 34 8 3 0 37
6 ALA 6 0 3 0 9
7 ALB 17 0 0 0 17
8 ALG 8 0 1 0 11
9 ALT 30 3 1 0 31
10 ALU 7 0 3 0 10
# ℹ 270 more rows
# ℹ Use `print(n = ...)` to see more rows
您可以提供等级的分类单元名称 种、属、科、目或更高。 WCVP仅提供科级以下的分类信息。我们包括了一个名为taxonomic_mapping的表格,用于根据APG IV将家族映射到目和更高的分类法。
# APG Ⅳ
head(taxonomic_mapping)
higher order family
1 Angiosperms Acorales Acoraceae
2 Angiosperms Alismatales Alismataceae
3 Angiosperms Alismatales Aponogetonaceae
4 Angiosperms Alismatales Araceae
5 Angiosperms Alismatales Butomaceae
6 Angiosperms Alismatales Cymodoceaceae
我们在后台使用此表,以便使用这些更高的分类排名进行筛选。例如,对所有禾本目进行汇总:
# summary all Poales:
wcvp_summary("Poales", taxon_rank = "order")
ℹ No area specified. Generating global summary.
$Taxon
[1] "Poales"
$Area
[1] "the world"
$Grouping_variable
[1] "area_code_l3"
$Total_number_of_species
[1] 23770
$Number_of_regionally_endemic_species
[1] 23770
$Summary
# A tibble: 368 × 6
area_code_l3 Native Endemic Introduced Extinct Total
<chr> <int> <int> <int> <int> <int>
1 ABT 359 0 70 0 429
2 AFG 485 16 15 0 504
3 AGE 908 31 166 0 1074
4 AGS 389 20 88 0 477
5 AGW 890 130 105 0 995
6 ALA 711 2 153 0 864
7 ALB 363 4 14 0 387
8 ALD 44 7 13 0 57
9 ALG 445 11 41 0 497
10 ALT 383 9 6 0 389
# ℹ 358 more rows
# ℹ Use `print(n = ...)` to see more rows
2.2 关于按分布区域筛选的说明
WCVP使用世界记录植物分布地理方案(WGSRPD)在第3级列出分类单元分布。这一水平对应于“植物国家”,它们大多遵循国家的边界,除非大国被分割或省略边远地区。rWCVP 中的函数期望area作为 WGSRPD 3 级代码的向量提供。
查找您感兴趣的整个区域可能会很烦人。例如,要按巴西的物种过滤,您需要提供 5 个代码的向量。为了方便起见,rWCVP 具有将区域名称转换为 WGSRPD 3 级代码向量的功能。
# convert region to codes
get_wgsrpd3_codes("Brazil")
ℹ Matches to input geography found at Country (Gallagher) and Region (Level 2)
[1] "BZC" "BZE" "BZL" "BZN" "BZS"
这可以直接输入到按区域过滤 WCVP 的函数中。
wcvp_summary("Poa", taxon_rank = "genus", area = get_wgsrpd3_codes("Southern Hemisphere"))
ℹ Matches to input geography found at Hemisphere level
ℹ Including WGSRPD areas that span the equator. To turn this off, use `include_equatorial = FALSE`
$Taxon
[1] "Poa"
$Area
[1] "Southern Hemisphere (incl. equatorial Level 3 areas)"
$Grouping_variable
[1] "area_code_l3"
$Total_number_of_species
[1] 264
$Number_of_regionally_endemic_species
[1] 237
$Summary
# A tibble: 74 × 6
area_code_l3 Native Endemic Introduced Extinct Total
<chr> <int> <int> <int> <int> <int>
1 AGE 13 2 5 0 18
2 AGS 24 0 10 0 34
3 AGW 34 8 3 0 37
4 ANT 0 0 1 0 1
5 ASC 0 0 1 0 1
6 ASP 2 1 3 0 5
7 ATP 8 1 3 0 11
8 BOL 31 2 3 0 34
9 BOR 2 1 0 0 2
10 BUR 3 0 0 0 3
# ℹ 64 more rows
# ℹ Use `print(n = ...)` to see more rows
笔者实践
m = wcvp_checklist(taxon = "Mahonia", taxon_rank = "genus")
b = wcvp_checklist(taxon = "Berberis", taxon_rank = "genus")

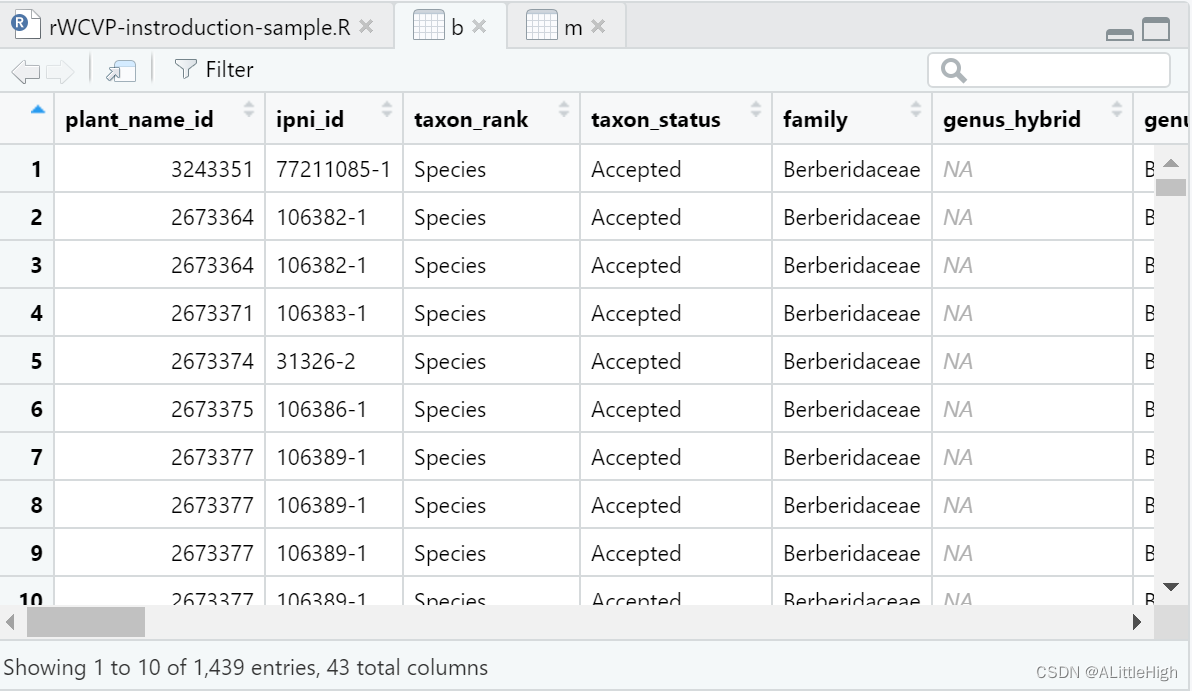
m表示的是mahonia,b表示berberis。在WCVP中并不承认mahonia的独立属地位,将其并入berberis 中。或许在rWCVP中筛选得到berberis,再从U.Taxonstand中尝试能否将Mahonia挑选出来。