欢迎关注我的CSDN:https://spike.blog.csdn.net/
本文地址:https://blog.csdn.net/caroline_wendy/article/details/130519945

DIT:https://zhanggroup.org/D-I-TASSER/
D-I-TASSER (Deep-learning based Iterative Threading ASSEmbly Refinement) is a new method extended from I-TASSER for high-accuracy protein structure and function predictions.
D-I-TASSER (Deep-learning based Iterative Threading ASSEmbly Refinement,即基于深度学习的迭代、穿线、组装、精调) ,从 I-TASSER 扩展的新方法,用于高精度的蛋白结构和功能预测。
Starting from a query sequence, D-I-TASSER first generates inter-residue contact and distance maps and hydrogen-bond (HB) networks using multiple deep neural-network predictors, including AttentionPotential (self-attention network built on MSA transformer) and DeepPotential.
从一个查询序列开始,D-I-TASSER 首先使用多个深度神经网络预测器,生成,残基间接触 (inter-residue contact) 、距离图 (distance maps) 以及 氢键 (HB) 网络,包括 AttentionPotential (基于 MSA Transformer 的自注意力网络) 和 DeepPotential 。
Meanwhile, it identifies structural templates by the meta-threading LOMETS3 approach, which includes the models built from the state-of-the-art AlphaFold2 program.
同时,通过 元穿线 (meta-threading) LOMETS3 方法,识别结构模板,其中包括,由最先进的AlphaFold2程序构建的模型。
The full-length atomic models are finally assembled by iterative fragment assembly Monte Carlo simultions under the guidance of I-TASSER force field and deep-learning contact/distance/HB restraints, where biological functions of the query protein are derived from the structure models by COFACTOR.
最终,全长原子模型,在 I-TASSER 力场和深度学习 接触(contact) / 距离(distance) / HB 约束的指导下,通过迭代的 片段组装蒙特卡罗模拟 (fragment assembly Monte Carlo simultions) 进行组装,其中查询蛋白的生物学功能,由 COFACTOR 从结构模型中推导出来。
The D-I-TASSER pipeline (as ‘UM-TBM’ and ‘Zheng’) was ranked as the No. 1 server/predictor in all categories of protein structure prediction in the most recent CASP15 experiment, including Multi-domain Targets, Single-domain Targets, and Multi-chain Targets.
D-I-TASSER 流程 (作为“UM-TBM”和“Zheng”),在最近的 CASP15 实验中,被评为所有类别蛋白质结构预测的第一名,在服务器组和预测器组中,包括多域目标、单域目标和多链目标。

DeepMSA2:https://zhanggroup.org/DeepMSA/
DeepMSA (version 2) is a hierarchical approach to create high-quality multiple sequence alignments (MSAs) for monomer and multimer proteins.
DeepMSA (version 2) 是层次方法,用于创建单体和多体蛋白质的高质量多序列比对 (MSAs)。
The method is built on iterative sequence database searching followed by fold-based MSA ranking and selection.
该方法基于迭代的序列数据库搜索,然后,根据折叠为基础的 MSA 排名和选择。
For protein monomers, MSAs are produced with three iterative MSA searching pipelines (dMSA, qMSA and mMSA) through whole-genome (Uniclust30 and UniRef90) and metagenome (Metaclust, BFD, Mgnify, TaraDB, MetaSourceDB and JGIclust) sequence databases.
对于蛋白质单体,通过全基因组 (Uniclust30 和 UniRef90) 和宏基因组 (Metaclust、BFD、Mgnify、TaraDB、MetaSourceDB 和 JGIclust) 序列数据库,使用三种迭代的 MSA 搜索流程 (dMSA、qMSA 和 mMSA) 生成 MSAs。
For protein multimers, a number of hybrid MSAs are created by pairing the sequences from monomer MSAs of the component chains, with the optimal multimer MSAs selected based on a combined score of MSA depth and folding score of the monomer chains.
对于蛋白质多体,通过将来自组分链的单体 MSAs 的序列配对,创建了一些混合 MSAs,根据组合得分,包括 MSA 深度和单体链的折叠得分,选择最佳的多体 MSAs。
Large-scale benchmark data show significant advantage of DeepMSA2 in generating accurate MSAs with balanced depth and alignment coverage which are most suitable for deep-learning based protein and protein complex stucture and function predictions.
大规模的基准数据显示,DeepMSA2 在生成准确的 MSAs 方面具有显著的优势,这些 MSAs 具有平衡的深度和比对覆盖度,最适合基于深度学习的蛋白质以及蛋白质复合物结构和功能预测。
DeepMSA2 整体架构:
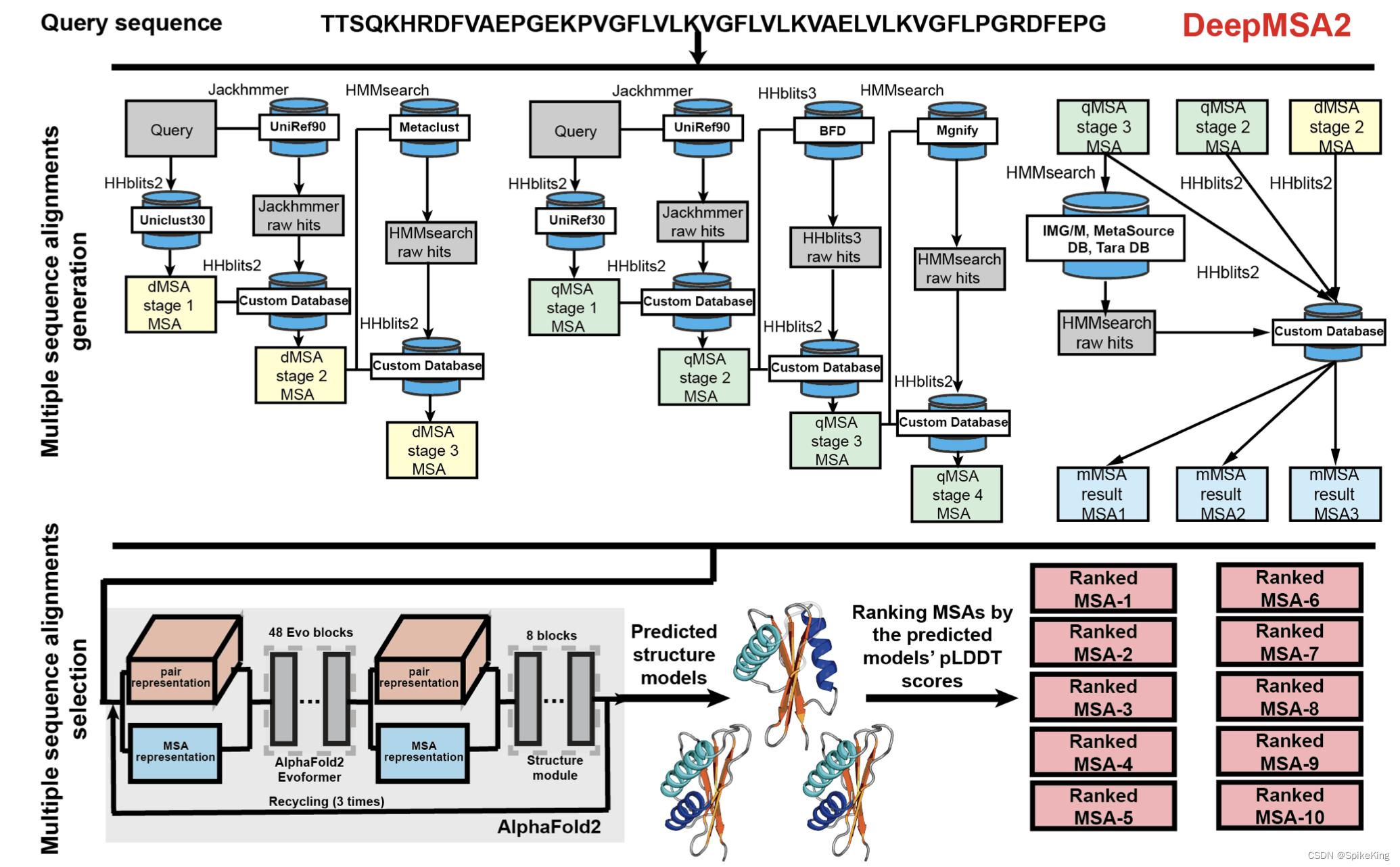
1. DeepMSA2 (no IMG)
核心逻辑位于DeepMSA2_noIMG.pl:
dq_time = time.time()
subprocess.run([f"contact/DeepMSA2/scripts/DeepMSA2_noIMG.pl",
self.seqname, self.datadir, self.pkgdir, self.libdir, python2])
print(f"[Info] dMSA和qMSA步运行时间: {time_elapsed(dq_time, time.time())}")
(1) 运行脚本qMSA2.py,即qMSA:
$python2 $HHLIB/scripts/qMSA2.py \\
-hhblitsdb=$qhhblitsdb \\
-jackhmmerdb=$qjackhmmerdb \\
-hhblits3db=$qhhblits3db \\
-hmmsearchdb=$qhmmsearchdb \\
-tmpdir=/tmp/$ENV{USER}/$tag \\
-ncpu=$cpu \\
$datadir/MSA/qMSA.fasta
(2) 运行脚本build_MSA.py,即dMSA,DeepMSA:
$python2 $HHLIB/scripts/build_MSA.py \\
-hhblitsdb=$dhhblitsdb \\
-jackhmmerdb=$djackhmmerdb \\
-hmmsearchdb=$dhmmsearchdb \\
-tmpdir=/tmp/$ENV{USER}/$tag \\
-ncpu=$cpu \\
$datadir/MSA/DeepMSA.fasta
1.1 qMSA
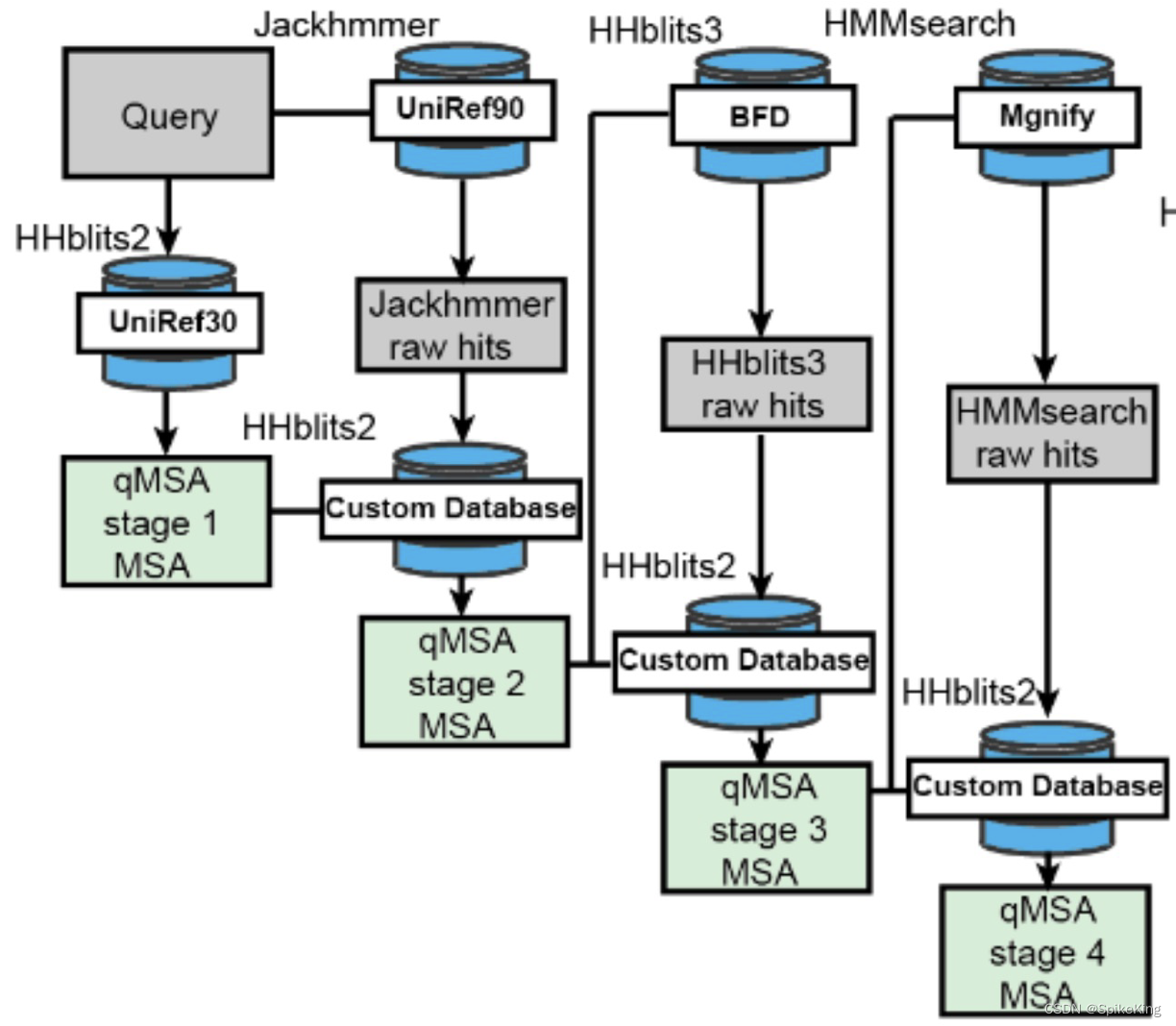
源码位于qMSA2.py,包括4个部分,即Uniclust30、UniRef90、BFD、MGnify。使用级联搜索,优先以Neff值高的MSA作为索引,最终,选择Neff值最高的作为qMSA。
第1部分:
- 搜索算法:hhblits
- 搜库:Uniclust30 (
uniclust30_2017_04),约64G,时间2017.04,最新是2022.02 - 输出:qMSA.hhba3m.gz、qMSA.hhbaln.gz
- 时间:19.1695s (117)
源码如下:
print("[Info] " + "+" * 100)
q1_time = time.time()
print("[Info] #### run hhblits ####")
hhblits_prefix=os.path.join(tmpdir,"hhblits")
if db_dict["hhblitsdb"]:
c1 = overwrite_dict["hhblits"]
c2 = not os.path.isfile(prefix+".hhbaln.gz")
c3 = not os.path.isfile(prefix+".hhba3m.gz")
print("[Info] 是否运行: {}, 具体条件: {} or {} or {}".format(c1 or c2 or c3, c1, c2, c3))
if c1 or c2 or c3:
# generates hhblits_prefix.a3m hhblits_prefix.aln
# hhblits_prefix.60.a3m hhblits_prefix.60.aln
sys.stdout = open(os.devnull, 'w') # 关闭日志
hhb_nf = run_hhblits(query_fasta, db_dict["hhblitsdb"], ncpu, hhblits_prefix)
plain2gz(hhblits_prefix + ".aln", prefix + ".hhbaln.gz")
plain2gz(hhblits_prefix + ".a3m", prefix + ".hhba3m.gz")
sys.stdout = sys.__stdout__ # 打开日志
else:
gz2plain(prefix + ".hhbaln.gz", hhblits_prefix + ".aln")
gz2plain(prefix + ".hhba3m.gz", hhblits_prefix + ".a3m")
hhb_nf = getNf(hhblits_prefix)
sys.stdout.write("%s and %s exists, skip hhblitsdb\n"%(prefix+".hhbaln",prefix+".hhba3m"))
nf = hhb_nf
print("[Info] hhb_nf = {}, target_nf = {}, passed: {}".format(nf, target_nf, hhb_nf >= target_nf))
if hhb_nf >= target_nf: # 满足条件直接停止
shutil.copyfile(hhblits_prefix+".aln", prefix+".aln")
shutil.copyfile(hhblits_prefix+".a3m", prefix+".a3m")
sys.stdout.write("Final MSA by hhblits with Nf >=%.1f\n" % nf)
if os.environ.get("DITADDUP", "N") == "N":
return nf
print("[Info] #### FINISH run hhblits ####")
print("[Info] 数据库: {}".format(db_dict["hhblitsdb"]))
print("[Info] qMSA 第1步耗时: {} 输出: {}".format((time.time() - q1_time), prefix + ".hhbaln.gz(hhba3m.gz)"))
print("[Info] " + "+" * 100)
Uniclust30:https://wwwuser.gwdg.de/~compbiol/uniclust/2022_02/
- 最新版本是2022.02
第2部分:
- 搜索算法:jackblits
- 搜库:UniRef90 (
uniref90.fasta),约74G - 额外输入:hhblits.a3m,搜子库 jackblits-mydb (4.9M)
- 输出:qMSA.jacaln.gz、qMSA.jaca3m.gz
- 时间:230.8850s (117)
源码如下:
print("[Info] " + "+" * 100)
q2_time = time.time()
print("[Info] #### run jack_hhblits ####")
jackblits_prefix=os.path.join(tmpdir,"jackblits")
if db_dict["jackhmmerdb"]:
c1 = overwrite_dict["jackhmmer"]
c2 = not os.path.isfile(prefix+".jacaln.gz")
c3 = not os.path.isfile(prefix+".jaca3m.gz")
print("[Info] 是否运行: {}, 具体条件: {} or {} or {}".format(c1 or c2 or c3, c1, c2, c3))
if c1 or c2 or c3:
# generates jackblits_prefix.a3m jackblits_prefix.aln
# jackblits_prefix.60.a3m jackblits_prefix.60.aln
sys.stdout = open(os.devnull, 'w') # 关闭日志
jack_nf=run_jackblits(query_fasta, db_dict["jackhmmerdb"].split(':'), ncpu, hhblits_prefix, jackblits_prefix)
plain2gz(jackblits_prefix+".aln",prefix+".jacaln.gz")
plain2gz(jackblits_prefix+".a3m",prefix+".jaca3m.gz")
sys.stdout = sys.__stdout__ # 打开日志
else:
gz2plain(prefix+".jacaln.gz",jackblits_prefix+".aln")
gz2plain(prefix+".jaca3m.gz",jackblits_prefix+".a3m")
jack_nf=getNf(jackblits_prefix)
sys.stdout.write("%s and %s exists, skip jackhmmerdb\n"%(prefix+".jacaln.gz",prefix+".jaca3m.gz"))
nf = max([jack_nf, hhb_nf])
print("[Info] jack_nf = {}, max nf = {}, target_nf = {}, passed: {}"
.format(jack_nf, nf, target_nf, nf >= target_nf))
if jack_nf >= target_nf:
shutil.copyfile(jackblits_prefix+".aln",prefix+".aln")
shutil.copyfile(jackblits_prefix+".a3m",prefix+".a3m")
sys.stdout.write("Final MSA by jackhmmer with Nf >=%.1f\n"%nf)
if os.environ.get("DITADDUP", "N") == "N":
return nf
print("[Info] #### FINISH run jack_hhblits ####")
print("[Info] 数据库: {}".format(db_dict["jackhmmerdb"]))
print("[Info] 是否使用 qMSA 第1步的输出: {}".format(os.path.isfile(hhblits_prefix + ".a3m")))
print("[Info] qMSA 第2步耗时: {} 输出: {}".format((time.time() - q2_time), prefix + ".jacaln.gz(jaca3m.gz)"))
print("[Info] " + "+" * 100)
第3部分:
- 搜索算法:hhblits3
- 搜库:BFD(
bfd_metaclust_clu_complete_id30_c90_final_seq.sorted_opt),1.8T - 额外输入:优先使用第2步的输出 (jackblits.a3m),其次第1步 (hhblits.a3m),搜子库hhblits3-mydb (1.5M)。
- 输出:qMSA.hh3aln.gz、qMSA.hh3a3m.gz
- 时间:276.7781s (117)
源码如下:
print("[Info] " + "+" * 100)
q3_time = time.time()
print("[Info] #### run hhblits3 ####") # , flush=True)
hhblits3_prefix=os.path.join(tmpdir, "hhblits3")
if db_dict["hhblits3db"]:
c1 = overwrite_dict["hhblits3"]
c2 = not os.path.isfile(prefix+".hh3aln.gz")
c3 = not os.path.isfile(prefix+".hh3a3m.gz")
print("[Info] 是否运行: {}, 具体条件: {} or {} or {}".format(c1 or c2 or c3, c1, c2, c3))
if c1 or c2 or c3:
sys.stdout = open(os.devnull, 'w') # 关闭日志
hh3_nf=run_hhblits3(query_fasta, db_dict["hhblits3db"].split(':'),
ncpu, hhblits_prefix, jackblits_prefix, hhblits3_prefix)
plain2gz(hhblits3_prefix+".aln", prefix+".hh3aln.gz")
plain2gz(hhblits3_prefix+".a3m", prefix+".hh3a3m.gz")
sys.stdout = sys.__stdout__ # 打开日志
else:
gz2plain(prefix+".hh3aln.gz",hhblits3_prefix+".aln")
gz2plain(prefix+".hh3a3m.gz",hhblits3_prefix+".a3m")
hh3_nf=getNf(hhblits3_prefix)
sys.stdout.write("%s and %s exists, skip hhblits3db\n"%(prefix+".hh3aln.gz",prefix+".hh3a3m.gz"))
nf = max([hh3_nf, jack_nf, hhb_nf])
print("[Info] hh3_nf = {}, max nf = {}, target_nf = {}, passed: {}"
.format(hh3_nf, nf, target_nf, hh3_nf >= target_nf))
if hh3_nf >= target_nf:
shutil.copyfile(hhblits3_prefix+".aln",prefix+".aln")
shutil.copyfile(hhblits3_prefix+".a3m",prefix+".a3m")
sys.stdout.write("Final MSA by hhblits3 with Nf >=%.1f\n"%nf)
if os.environ.get("DITADDUP", "N") == "N":
return nf
print("[Info] 数据库: {}".format(db_dict["hhblits3db"]))
print("[Info] 是否使用 qMSA 第1-2步的输出: {}, {}".format(
os.path.isfile(jackblits_prefix+".a3m"), jackblits_prefix+".a3m"))
print("[Info] qMSA 第3步耗时: {} 输出: {}".format((time.time() - q3_time), prefix + ".hh3aln.gz(hh3a3m.gz)"))
print("[Info] " + "+" * 100)
第4部分:
- 搜索算法:hmsblits (hmmsearch)
- 搜库:MGnify(mgy_clusters.clean.fasta),60G
- 额外输入:使用最大NF值的a3m
- 输出:qMSA.hmsaln.gz、qMSA.hmsa3m.gz
- 时间:343.5184 (117)
源码如下:
q4_time = time.time()
print("[Info] " + "+" * 100)
print("[Info] #### run hmmsearch ####") # , flush=True)
hmmsearch_prefix = os.path.join(tmpdir,"hmmsearch")
if db_dict["hmmsearchdb"]:
c1 = overwrite_dict["hmmsearch"]
c2 = not os.path.isfile(prefix+".hmsaln.gz")
c3 = not os.path.isfile(prefix+".hmsa3m.gz")
print("[Info] 是否运行: {}, 具体条件: {} or {} or {}".format(c1 or c2 or c3, c1, c2, c3))
if c1 or c2 or c3:
# TODO: 建议使用字典
# 优先处理 jumpstart_profile_prefix,其次处理 query_profile_prefix
query_profile_prefix = jackblits_prefix # 使用最大的NF值
if jack_nf < hhb_nf:
query_profile_prefix = hhblits_prefix
jumpstart_profile_prefix = hhblits3_prefix # 使用最大的NF值
if hh3_nf < jack_nf:
jumpstart_profile_prefix = jackblits_prefix
if jack_nf < hhb_nf:
jumpstart_profile_prefix = hhblits_prefix
# generates hmmsearch_prefix.afq hmmsearch_prefix.hmm
# hmmsearch_prefix.redundant hmmsearch_prefix.nonredundant
# hmmsearch_prefix.aln
sys.stdout = open(os.devnull, 'w') # 关闭日志
hms_nf=search_metaclust(query_fasta, sequence, query_profile_prefix, jumpstart_profile_prefix,
db_dict["hmmsearchdb"].split(':'),ncpu,hmmsearch_prefix)
plain2gz(hmmsearch_prefix+".aln",prefix+".hmsaln.gz")
plain2gz(hmmsearch_prefix+".a3m",prefix+".hmsa3m.gz")
sys.stdout = sys.__stdout__ # 打开日志
print("[Info] 是否使用 qMSA 第1-3步的输出: {}".format(jumpstart_profile_prefix + ".a3m"))
else:
gz2plain(prefix+".hmsaln.gz",hmmsearch_prefix+".aln")
gz2plain(prefix+".hmsa3m.gz",hmmsearch_prefix+".a3m")
sys.stdout.write("%s and %s exists, skip hmmsearchdb\n"%(prefix+".hmsaln.gz",prefix+".hmsa3m.gz"))
hms_nf=getNf(hmmsearch_prefix)
nf = max([hh3_nf, jack_nf, hhb_nf, hms_nf])
print("[Info] NF - hhb_nf(s1): {}, jack_nf(s2): {}, hh3_nf(s3): {}, hms_nf(s4): {}"
.format(hhb_nf, jack_nf, hh3_nf, hms_nf))
print("[Info] hms_nf = {}, max nf = {} (target_nf), passed: {}".format(hms_nf, nf, hms_nf >= nf))
if hms_nf > nf: # hmmsearch replaces jackblits and hhblits result
nf = hms_nf
shutil.copyfile(hmmsearch_prefix+".aln", prefix+".aln")
shutil.copyfile(hmmsearch_prefix+".a3m", prefix+".a3m")
sys.stdout.write("Final MSA by hmmsearch with Nf >=%.1f\n"%nf)
if os.environ.get("DITADDUP", "N") == "N":
return nf
print("[Info] 数据库: {}".format(db_dict["hmmsearchdb"]))
print("[Info] qMSA 第4步耗时: {} 输出: {}".format((time.time() - q4_time), prefix + ".hmsaln.gz(hmsa3m.gz)"))
print("[Info] " + "+" * 100)
最终,根据Neff值,在4组MSA中,选出最终的MSA,作为qMSA.a3m,理论上Neff值越来越高。例如:
NF - hhb_nf(s1): 7.98306, jack_nf(s2): 20.9298, hh3_nf(s3): 22.5877, hms_nf(s4): 25.3418
MSA Len - hhb: 121, jac: 402, hh3: 492, hms: 568
源码如下:
# 选择最优的NF作为qMSA的最终输出
print("[Info] NF - hhb_nf(s1): {}, jack_nf(s2): {}, hh3_nf(s3): {}, hms_nf(s4): {}"
.format(hhb_nf, jack_nf, hh3_nf, hms_nf))
nf = max([hh3_nf, jack_nf, hhb_nf, hms_nf])
print("[Info] hms_nf = {}, max nf = {} (target_nf), passed: {}".format(hms_nf, nf, hms_nf >= nf))
if hms_nf >= nf: # hmmsearch replaces jackblits and hhblits result
nf = hms_nf
shutil.copyfile(hmmsearch_prefix + ".aln", prefix + ".aln")
shutil.copyfile(hmmsearch_prefix + ".a3m", prefix + ".a3m")
sys.stdout.write("[Info] step4 - hmmsearch MSA has %.1f Nf. Output anyway.\n" % hms_nf)
else:
if hh3_nf >= max(jack_nf, hhb_nf):
shutil.copyfile(hhblits3_prefix+".aln", prefix+".aln")
shutil.copyfile(hhblits3_prefix+".a3m", prefix+".a3m")
sys.stdout.write("[Info] step3 - hhblits3 MSA has %.1f Nf. Output anyway.\n"%hh3_nf)
elif jack_nf >= max(hh3_nf, hhb_nf):
shutil.copyfile(jackblits_prefix+".aln", prefix+".aln")
shutil.copyfile(jackblits_prefix+".a3m", prefix+".a3m")
sys.stdout.write("[Info] step2 - jackhmmer MSA has %.1f Nf. Output anyway.\n"%jack_nf)
else: # hhb_nf >= nf:
shutil.copyfile(hhblits_prefix+".aln", prefix+".aln")
shutil.copyfile(hhblits_prefix+".a3m", prefix+".a3m")
sys.stdout.write("[Info] step1 - hhblits MSA has %.1f Nf. Output anyway.\n"%hhb_nf)
# prefix 即 output 的目录
print("[Info] qMSA 执行完成 {}, 输出 {}".format((time.time() - q1_time), prefix+".aln|a3m"))
print("[Info] " + "+" * 100)
Neff,在MSA中,默认高质量阈值是128,公式如下:

1.2 dMSA (DeepMSA)
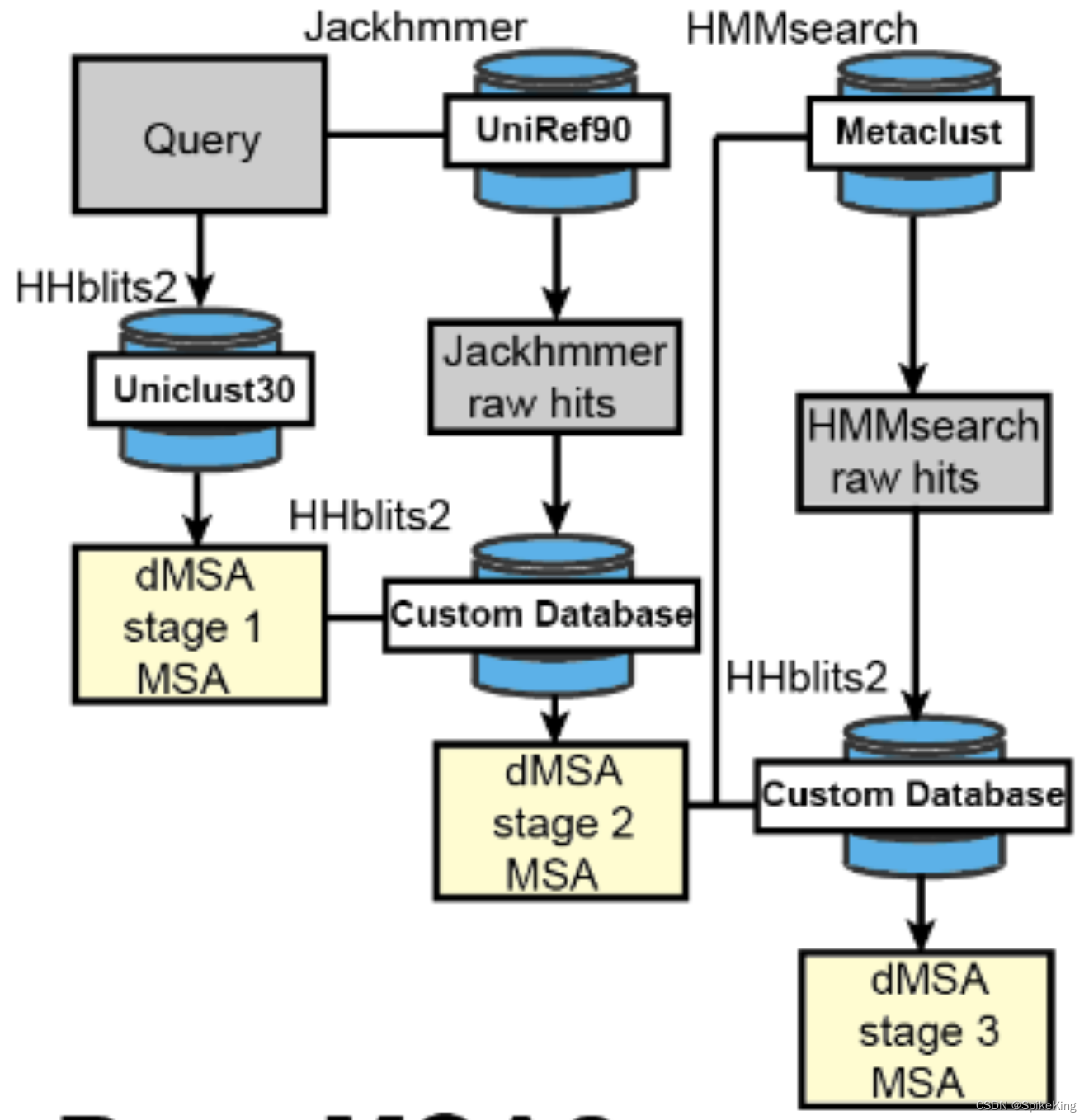
源码位于build_MSA.py,包括4个部分,即Uniclust30、UniRef90、Metaclust&MGnify,最后一步,联合搜索Metaclust&MGnify。
第1部分:与qMSA完全相同,包括输出MSA的Neff值。
第2部分:与qMSA完全相同,包括输出MSA的Neff值。
第3部分,目标就是搜索,第3部分:
- 搜索算法:hmsblits (hmmsearch)
- 搜库:Metaclust(metaclust.fasta),153G;MGnify(mgy_clusters.clean.fasta),60G
- 额外输入:使用最大NF值的a3m
- 输出:DeepMSA.hmsaln、DeepMSA.hmsa3m
- 时间:1086.5113 (117),1T大约84.9765
源码如下:
print("[Info] " + "+" * 100)
d3_time = time.time()
print("[Info] #### run hmmsearch ####") # , flush=True)
hmmsearch_prefix=os.path.join(tmpdir,"hmmsearch")
if db_dict["hmmsearchdb"]:
c1 = overwrite_dict["hmmsearch"]
c2 = not os.path.isfile(prefix+".hmsaln")
c3 = not os.path.isfile(prefix+".hmsa3m")
c4 = build_hmmsearch_db
print("[Info] 是否运行: {}, 具体条件: {} or {} or {} or {}".format(c1 or c2 or c3 or c4, c1, c2, c3, c4))
if c1 or c2 or c3 or c4:
# generates hmmsearch_prefix.afq hmmsearch_prefix.hmm
# hmmsearch_prefix.redundant hmmsearch_prefix.nonredundant
# hmmsearch_prefix.aln
sys.stdout = open(os.devnull, 'w') # 关闭日志
hms_nf=search_metaclust(query_fasta, sequence,
hhblits_prefix if jack_nf < hhb_nf else jackblits_prefix,
db_dict["hmmsearchdb"].split(':'), ncpu, hmmsearch_prefix)
shutil.copyfile(hmmsearch_prefix + ".aln", prefix + ".hmsaln")
if os.path.isfile(hmmsearch_prefix + ".a3m"):
shutil.copyfile(hmmsearch_prefix + ".a3m", prefix + ".hmsa3m")
sys.stdout = sys.__stdout__ # 打开日志
else:
shutil.copyfile(prefix+".hmsaln",hmmsearch_prefix+".aln")
if os.path.isfile(prefix+".hmsa3m"):
shutil.copyfile(prefix+".hmsa3m",hmmsearch_prefix+".a3m")
sys.stdout.write("[Info] %s exists, skip hmmsearchdb\n"%(prefix+".hmsaln"))
hms_nf = getNf(hmmsearch_prefix)
print("[Info] 数据库: {}".format(db_dict["hmmsearchdb"]))
print("[Info] dMSA 第3步耗时: {} 输出: {}".format((time.time() - d3_time), prefix + ".hmsaln(hmsa3m)"))
print("[Info] " + "+" * 100)
其余,也与qMSA相同,也是根据Neff选择最优的MSA,例如:
NF - hhb_nf(s1): 7.98306, jack_nf(s2): 20.9298, hms_nf(s3): 23.6956
2. DeepMSA2 (IMG)
检测是否需要运行IMG:
next if (-f "$datadir/JGI/$DBfasta.cdhit"); # 检查是否需要运行
第1部分:
- 使用
qhmmsearch搜索qMSA.hh3aln.gz - 输出:
DB.fasta.bq.cdhit、DB.fasta.bq.cdhit.clstr - 耗时:24.9616s
源码如下:
cp $datadir/MSA/qMSA.hh3aln.gz seq.hh3aln.gz
gzip -df seq.hh3aln.gz
if [ ! -s "seq.hh3aln" ];then
cp $datadir/MSA/DeepMSA.jacaln seq.hh3aln
fi
if [ ! -s "seq.hh3aln" ];then
cp $datadir/MSA/DeepMSA.hhbaln seq.hh3aln
fi
sed = seq.hh3aln |sed 'N;s/\\n/\\t/'|sed 's/^/>/g'|sed 's/\\t/\\n/g'| $HHLIB/bin/qhmmbuild -n aln --amino -O seq.afq --informat afa seq.hmm -
$HHLIB/qhmmer_new/qhmmsearch --cpu 192 -E 10 --incE 1e-3 -A $DBfasta.match --tblout $DBfasta.tbl -o $DBfasta.out seq.hmm $JGI/$DBfasta
$HHLIB/bin/esl-sfetch -f $JGI/$DBfasta $DBfasta.tbl|sed 's/*//g' > $DBfasta.fseqs
$HHLIB/bin/cd-hit -i $DBfasta.fseqs -o $datadir/JGI/$DBfasta.cdhit -c 1 -M 3000
第2部分:常规搜索,类似qMSA和dMSA,输出DeepJGI.aln、q3JGI.aln、q4JGI.aln三个文件。
源码如下:
my $cmd="$HHLIB/scripts/qMSA2.py -hhblitsdb=$hhblitsdb -jackhmmerdb=$jackhmmerdb -hhblits3db=$hhblits3db -hmmsearchdb=$tmpdir/DB.fasta.fseqs -tmpdir=$tmpdir/MSA $datadir/JGI/q4JGI.fasta";
...
my $cmd="$HHLIB/scripts/qMSA2.py -hhblitsdb=$hhblitsdb -jackhmmerdb=$jackhmmerdb -hmmsearchdb=$tmpdir/DB.fasta.fseqs -tmpdir=$tmpdir/MSA $datadir/JGI/q3JGI.fasta";
...
my $cmd="$HHLIB/scripts/build_MSA.py -hhblitsdb=$hhblitsdb -jackhmmerdb=$jackhmmerdb -hmmsearchdb=$tmpdir/DB.fasta.fseqs -tmpdir=$tmpdir/MSA $datadir/JGI/DeepJGI.fasta";
3. MSA 评估
使用官方的AF2 (AlphaFold2),评估MSA,区别是模型只使用model1_1、调整(relax)1次,因此,只输出1个模型,通过pLDDT进行排序。
具体步骤如下:
- 整理所有MSA,包括qMSA、dMSA和JGI-MSA,去重,保留7个MSA。
- 将不同的MSA建立不同的文件夹,文件夹中只有seq.aln、seq.a3m两种格式。
- 通过AF2预测结构,输入至MSA文件夹的seq目录。
- 整理评估结果至 AF2models 文件夹,MSA 信息位于 model.info 文件。
- 最优的MSA,位于MSA文件夹中,即 protein.a3m、protein.aln。
参考脚本:
I-TASSERmod/run_AF2_multiMSA.pythirdparty/alphafold2/run_alphafold_msa_benchmark.shthirdparty/alphafold2/run_alphafold_msa_benchmark.py
AF2模型位置位于:thirdparty/alphafold2/run_alphafold_msa_benchmark.py
model_params = data.get_model_haiku_params(
model_name=model_name, data_dir=FLAGS.data_dir)
...
# thirdparty/alphafold2/alphafold/model/data.py
def get_model_haiku_params(model_name: str, data_dir: str) -> hk.Params:
"""Get the Haiku parameters from a model name."""
path = os.path.join(data_dir, 'params', f'params_{model_name}.npz')
with open(path, 'rb') as f:
params = np.load(io.BytesIO(f.read()), allow_pickle=False)
return utils.flat_params_to_haiku(params)
官网:https://github.com/deepmind/alphafold

参考
- StackOverflow - Specifying multiple trusted hosts in pip.conf
- linux解压缩.gz文件命令
- 知乎 - CASP15蛋白结构预测冠军算法浅析