文章目录
第四章 DNA的生物合成
第一节 DNA复制的一般特征
先介绍一些乱七八糟的知识
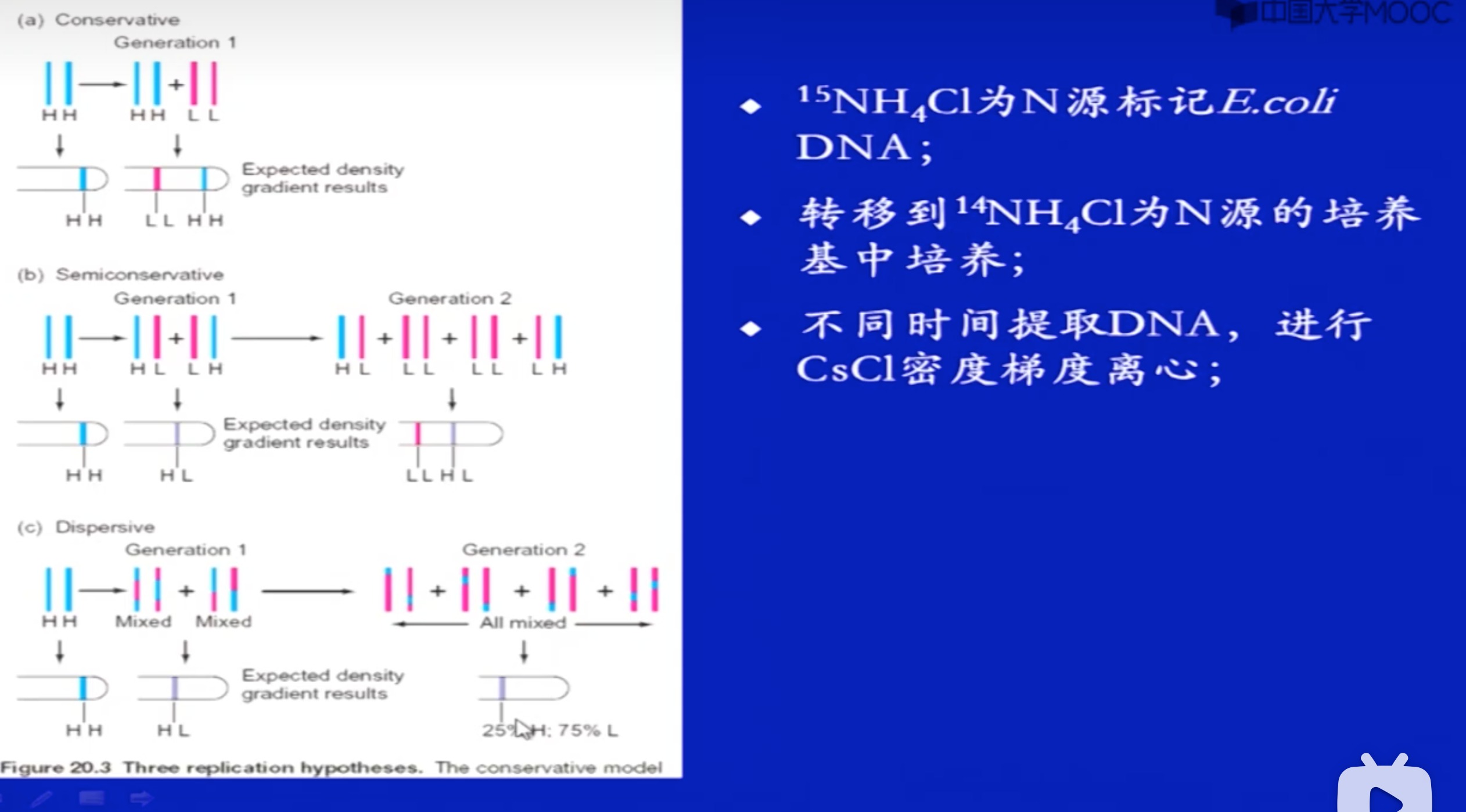
总共是三个特征,并介绍了一下相关的实验
1 DNA的半保留复制
经典实验:同位素标记,大肠杆菌实验
假设三种复制方式
实验结果
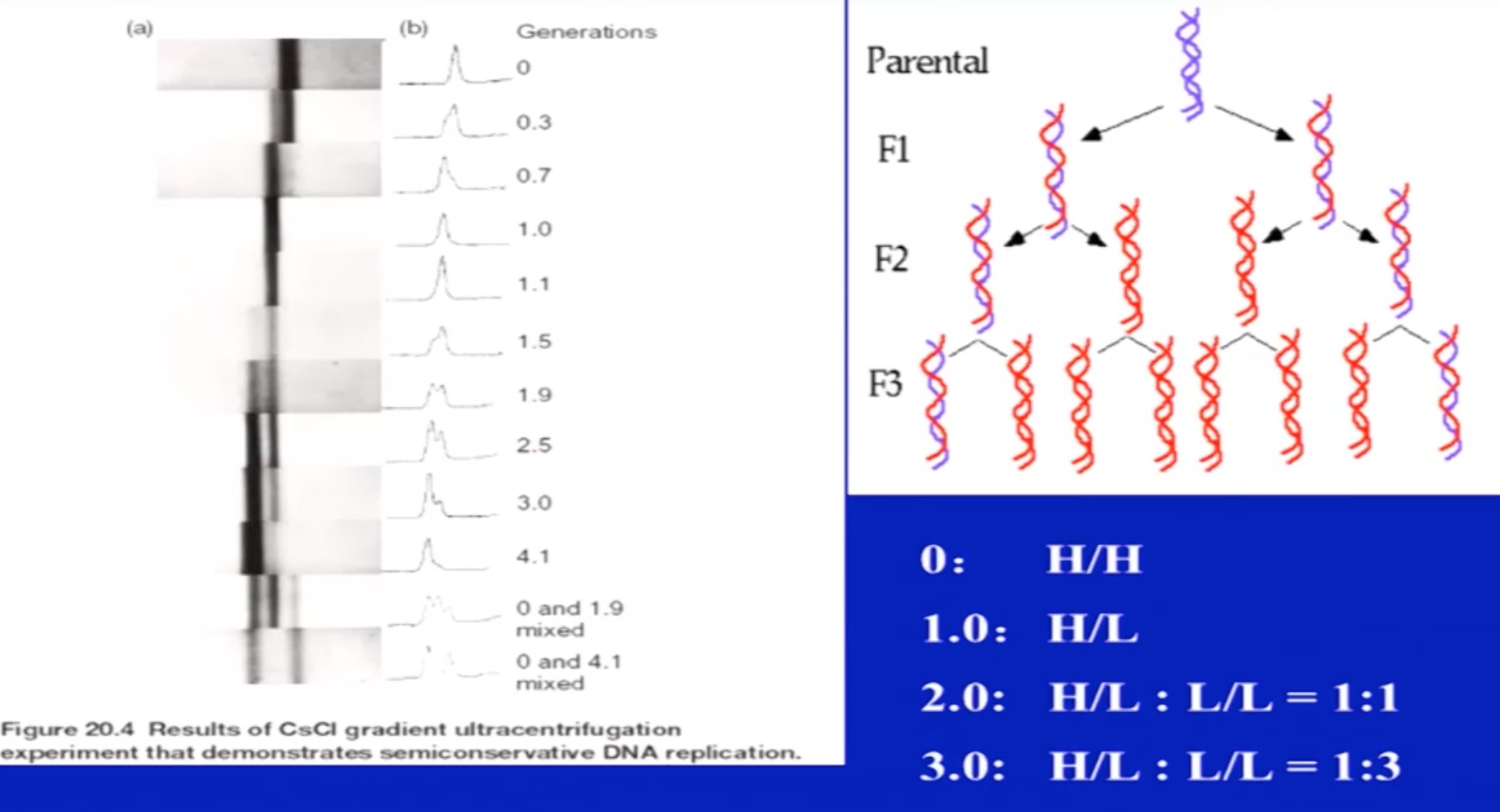
实验2
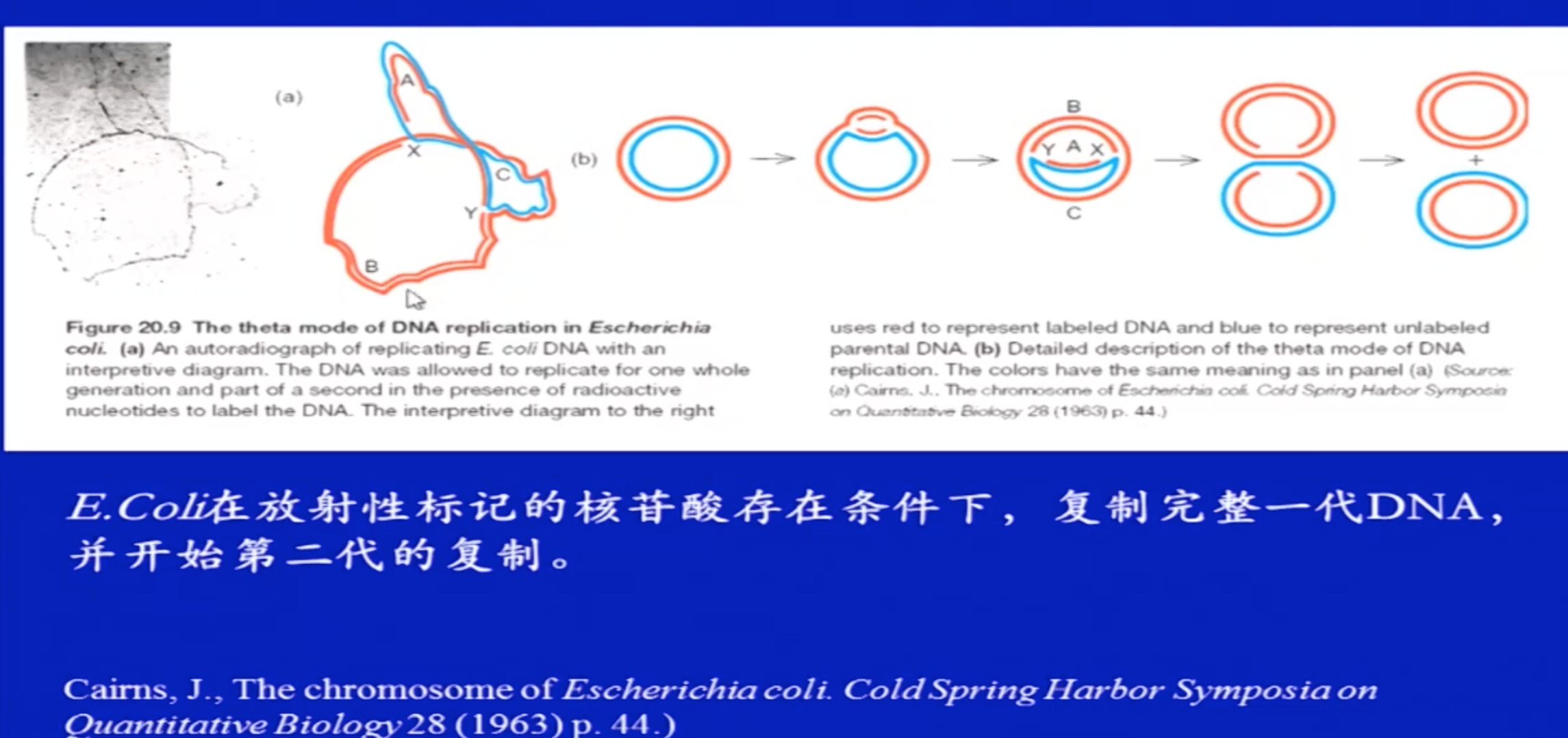
名词解释
- DNA复制
- 亲代双链DNA分子在DNA聚合酶的作用下,分别以每条单链 DNA分子为模板,聚合与自身碱基可以互补配对的游离的dNTP,合成出两条与亲代DNA分子完全相同的子代DNA分子的过程。
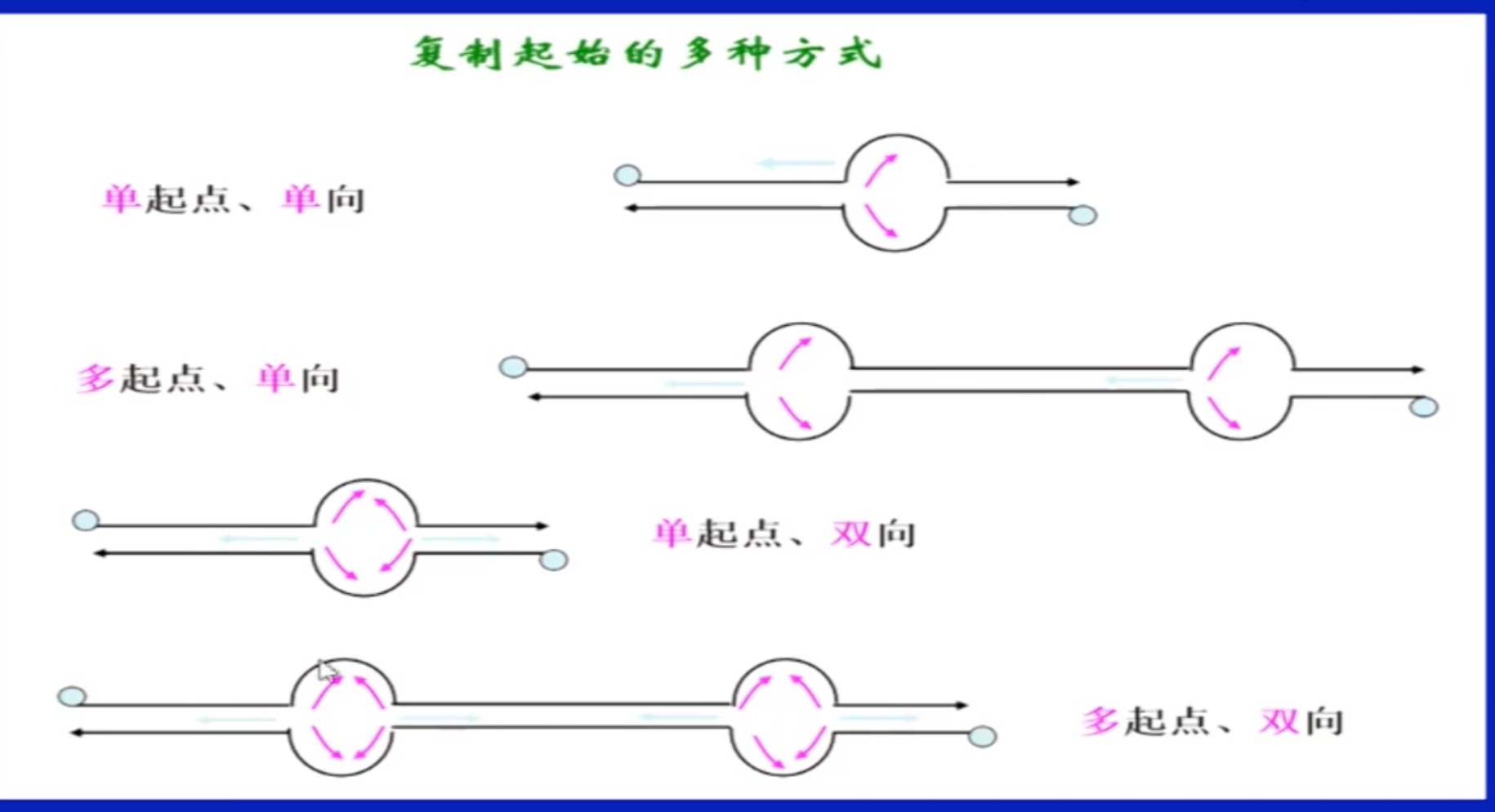
2 DNA的双向复制
- 复制从DNA分子的特定位置开始:
- 复制起点/复制原点 (origin, O);
- 能够独立进行复制的基因组单位:
- 复制子(replicon)
- 一个复制子只含有一个复制起点
- DNA链从5’一3‘延伸;
- 复制起始的多种方式
- 枯草杆菌实验
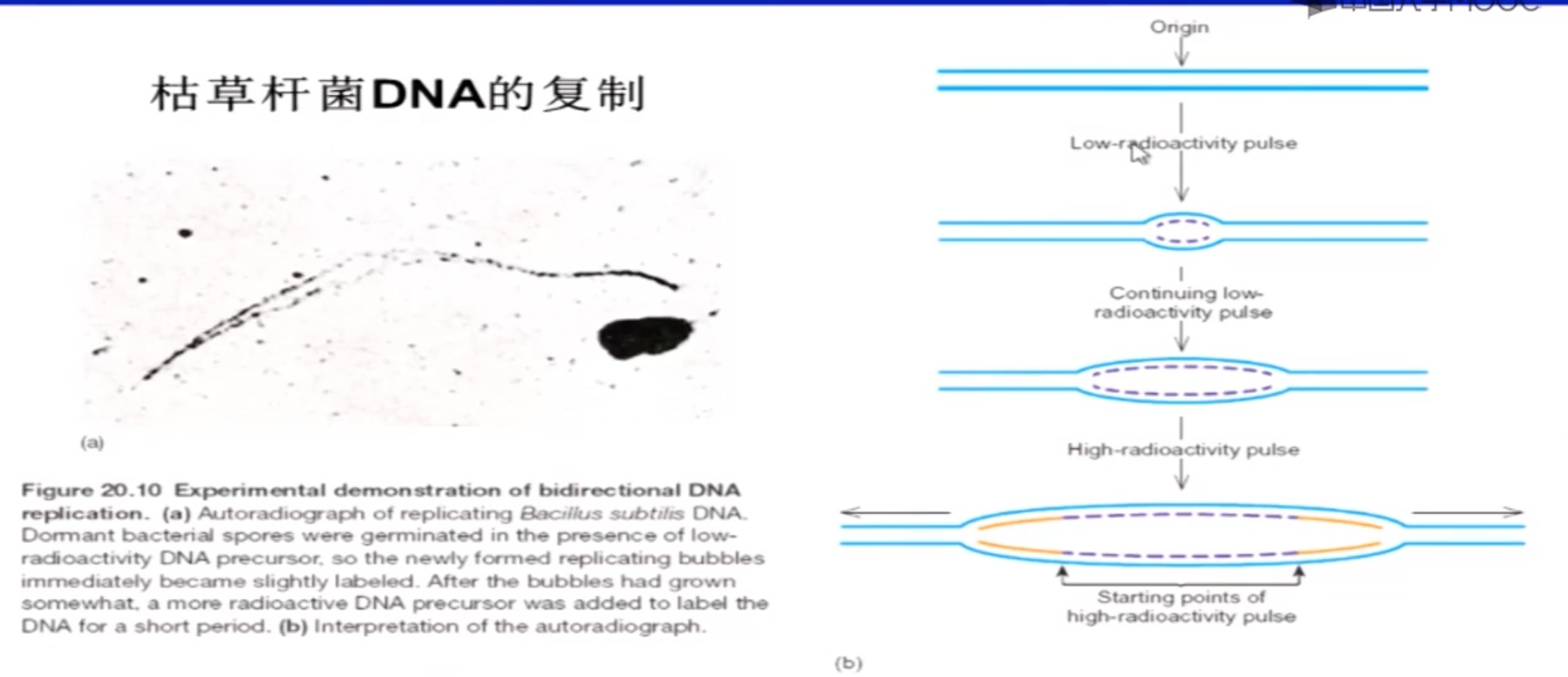
- 动物DNA实验
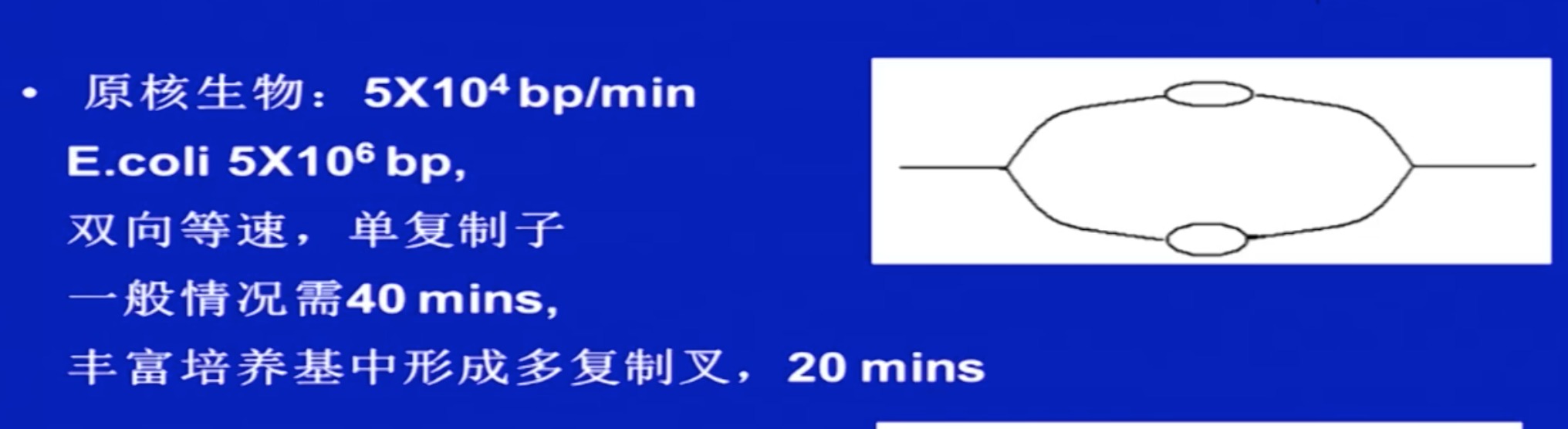
- 原核生物:多复制叉:在一轮复制还没结束的时候,复制起点直接进行新一轮的复制
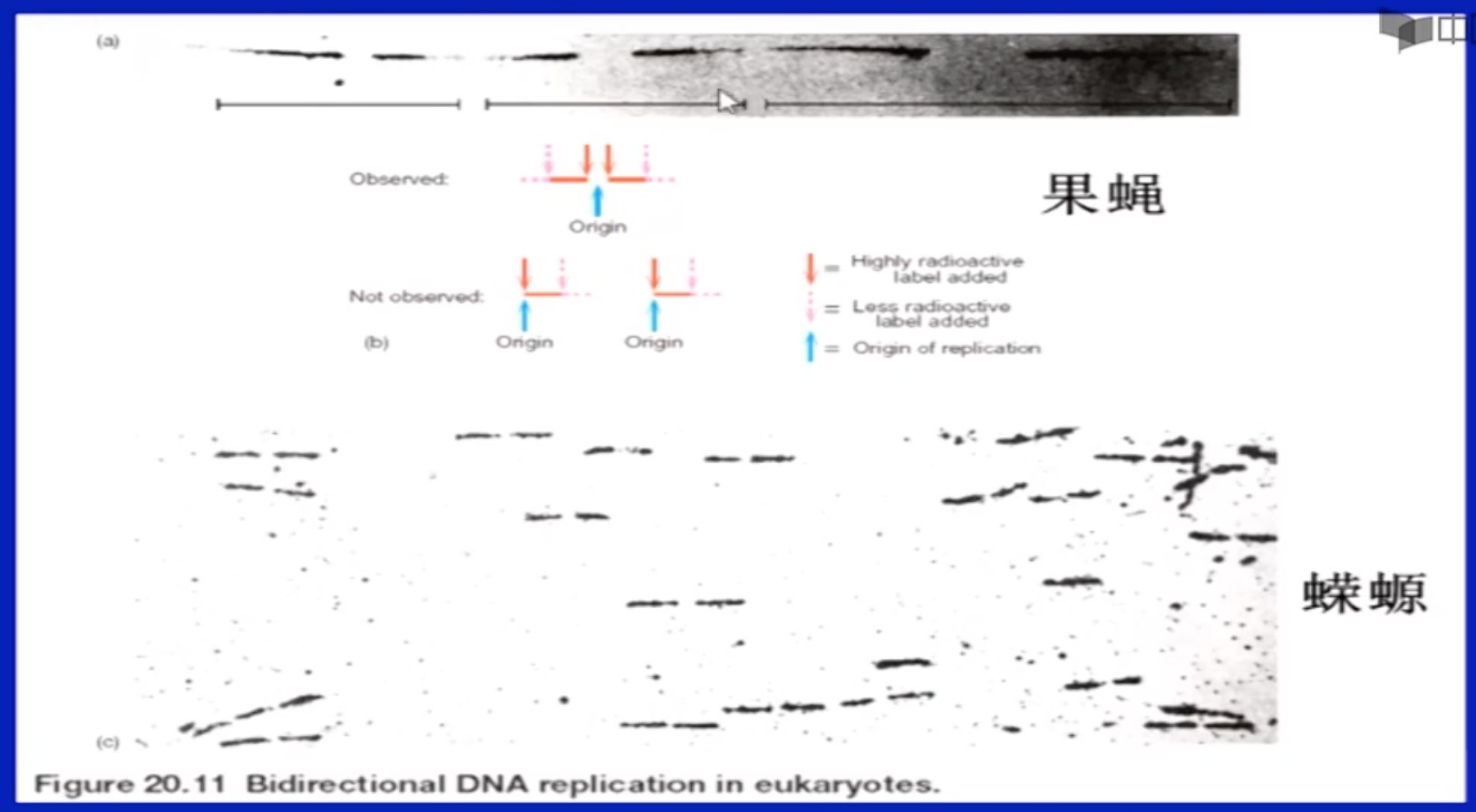
- 真核生物:多复制子,DNA复制过程中有多个复制起点

- 特殊复制方式
- 完全单向

- 滚环复制
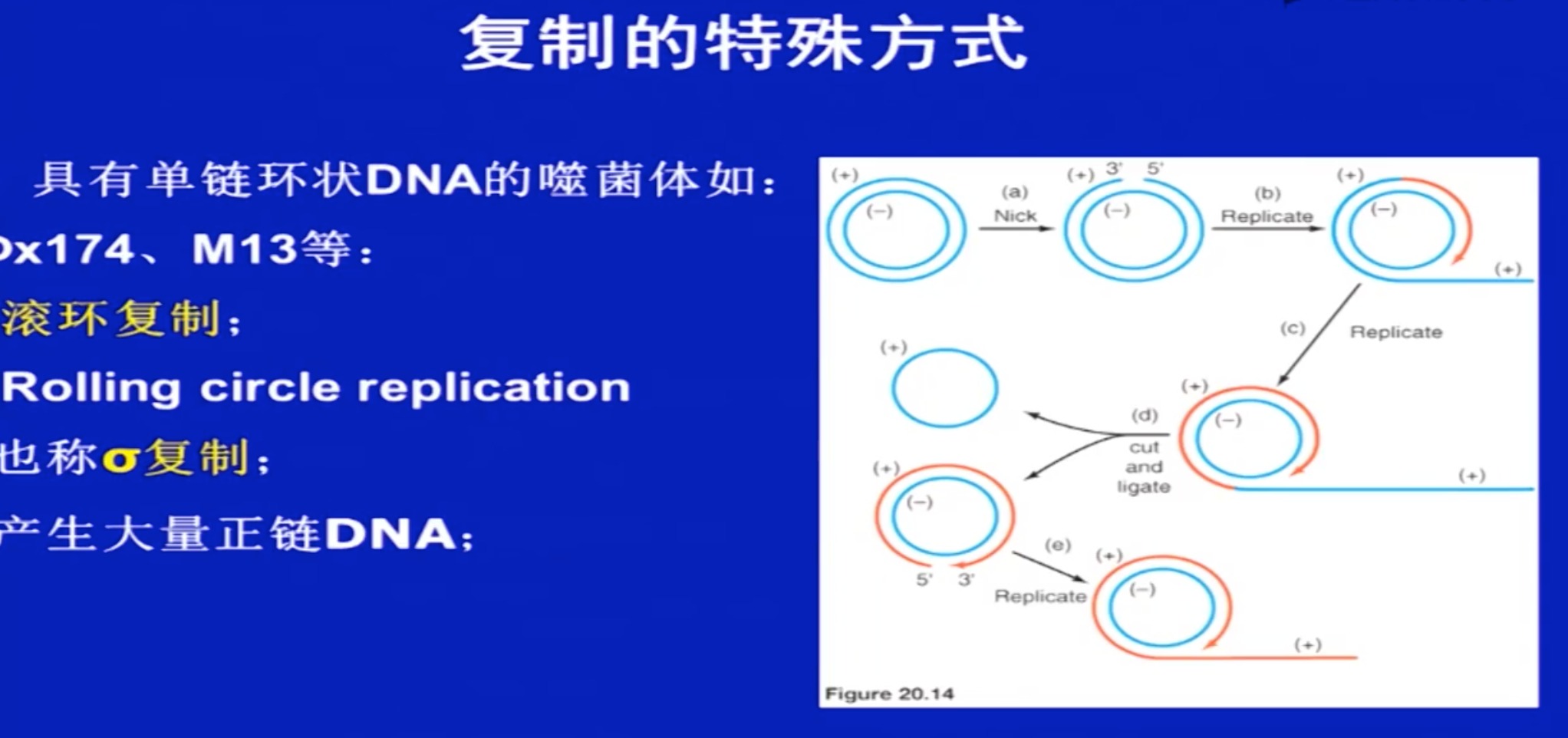
- D环复制
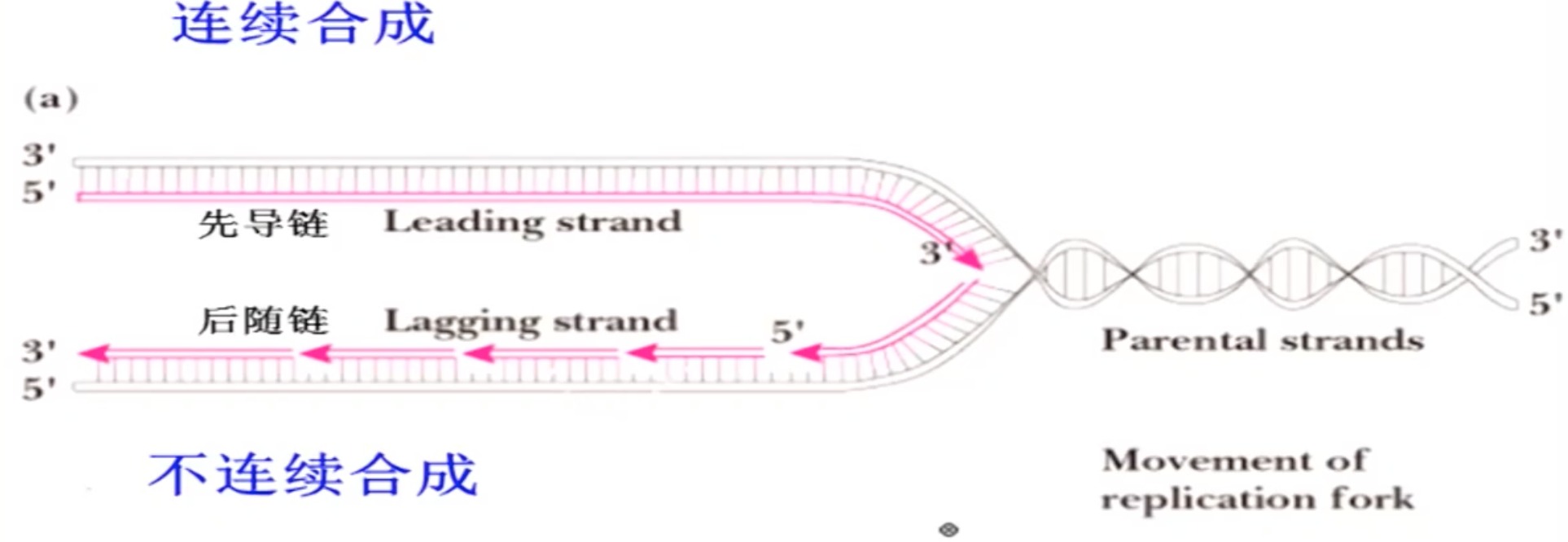
3 DNA的半不连续复制
讲了一个经典实验

一条链连续合成,一条链不连续合成
第二节 DNA复制的酶学
-
DNA的复制需要多种酶和蛋白质的参与;
-
这些酶和蛋自质在复制叉处组装成复合物、负责DNA的合成,称为复制体 (replisome)
-
ome结尾的一般都是指一些复合物
-
在这里主要讲六大类参与DNA复制的酶
1 DNA聚合酶
DNA polymerase/DNA pol
DNA dependent/directed DNA poly merase
DDDPase
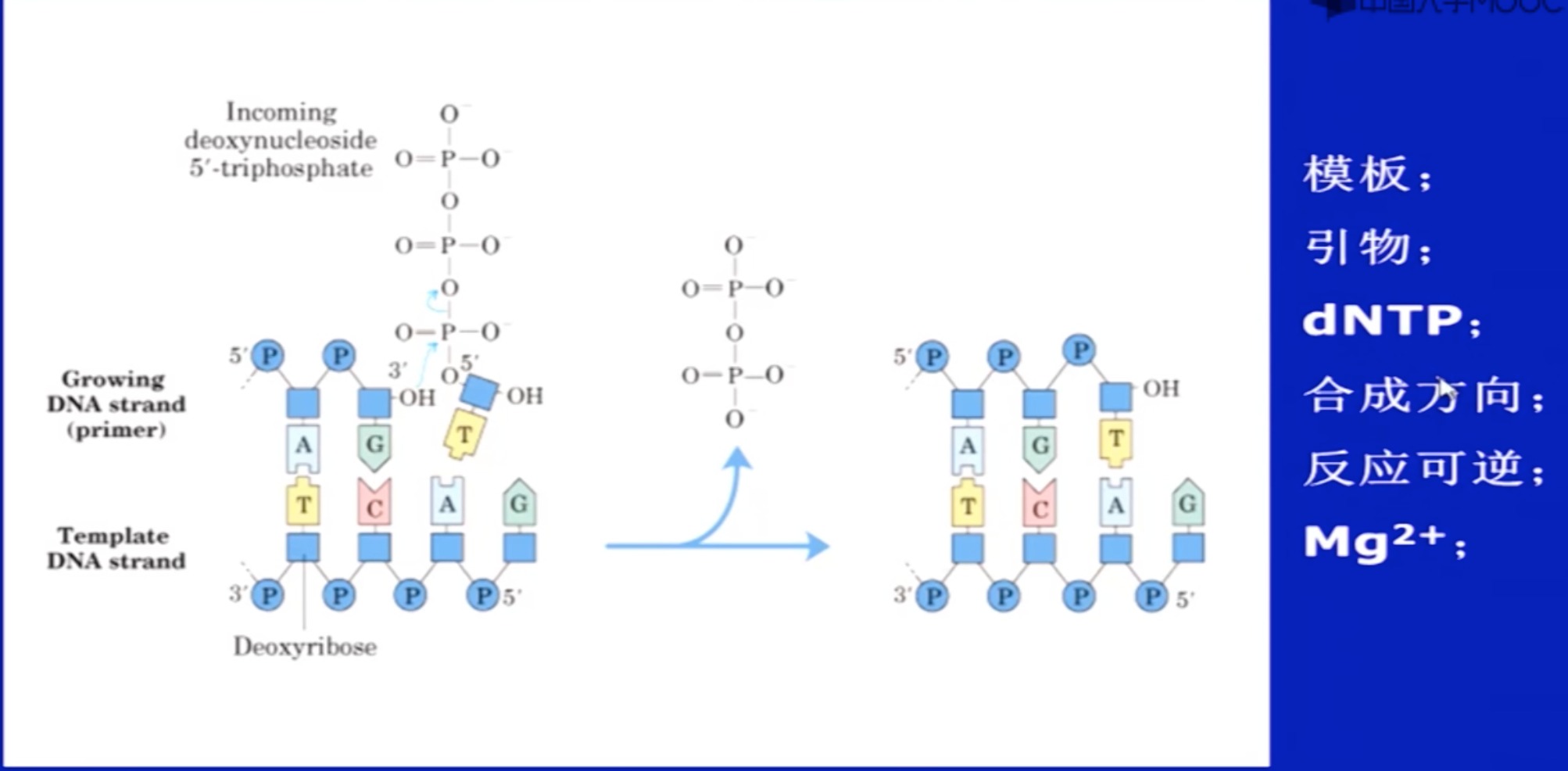
以DNA为模版来合成DNA

- 特殊之处
- 必须要有模版
- 它不能从头合成,要有引物(与RNA聚合酶差别之一)
- 在原核和真核生物之间的区别
1.1 原核生物DNA pol
-
E.coli DNA 聚合酶:
DNA pol
DNA pol IDNA pol ll
DNA pol lv
DNA pol V
1.1.1 DNA pol I
-
这个酶(DNA pol I)有很多种酶活性,以下三种最为重要(不仅有合成DNA的活性,还有水解DNA的活性)
-
多功能酶
3’ to 5’ exonuclease activity(3’ 核酸外切酶)
5‘ to 3" exonuclease activity(5’ 核酸外切酶)
5 to 3’ DNA polymerizing activity(DNA聚合酶) -
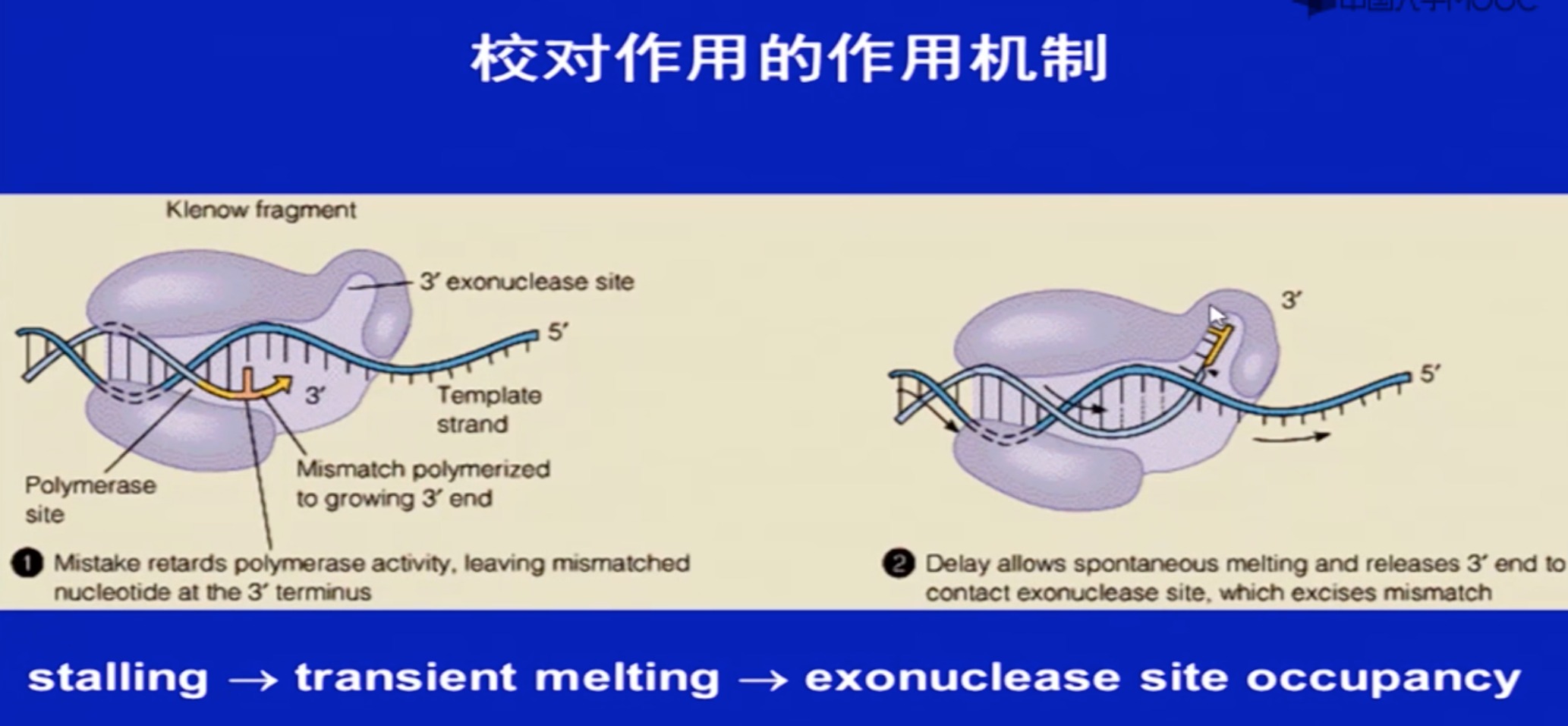
聚合酶和3’外切酶共同作用,可以有校对作用(碱基配对出错时,改正,大多数DNA聚合酶都有的。与RNA聚合酶差别之二)
-
KLENOW酶的二级结构
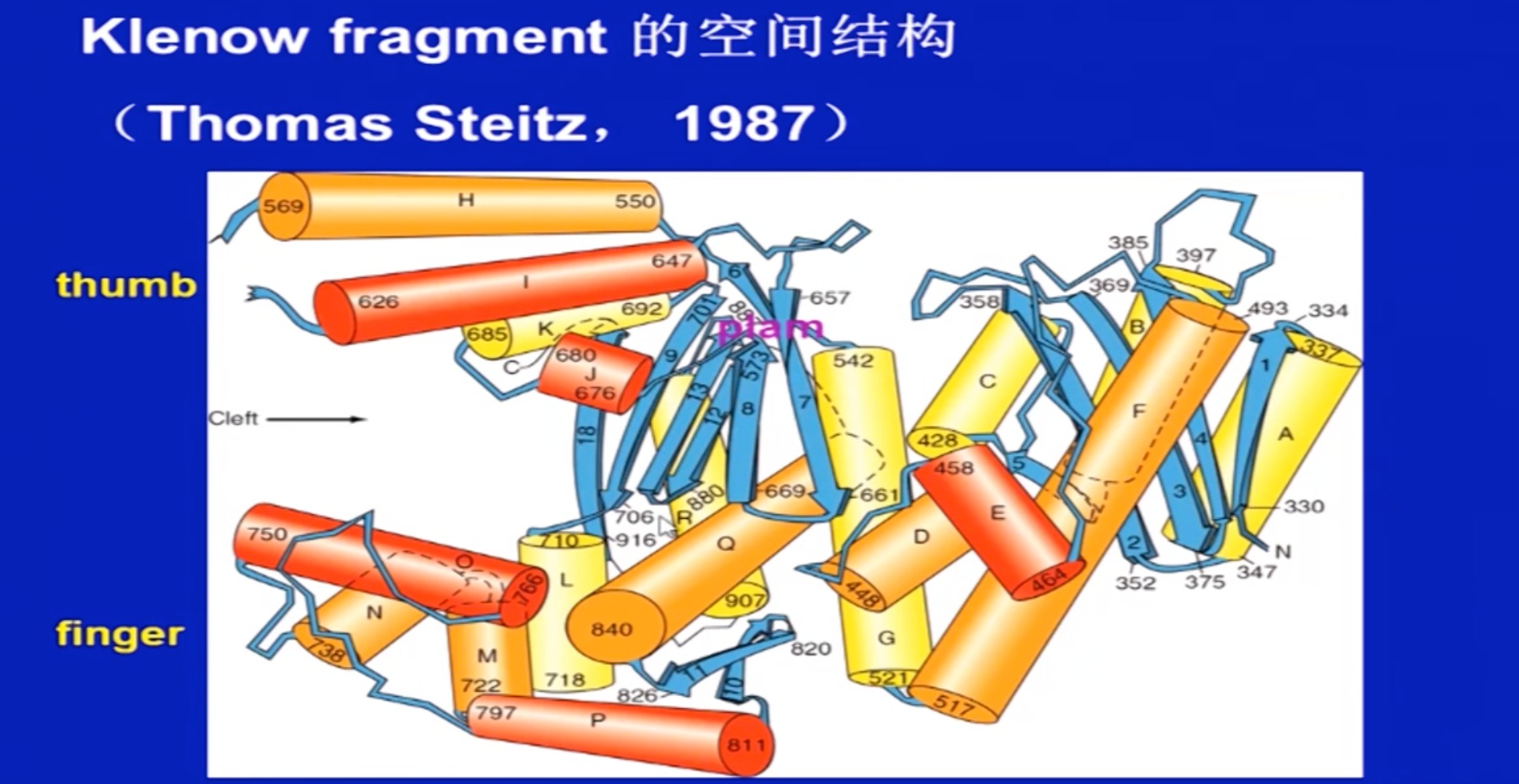
- 像一个手,可以结合DNA链
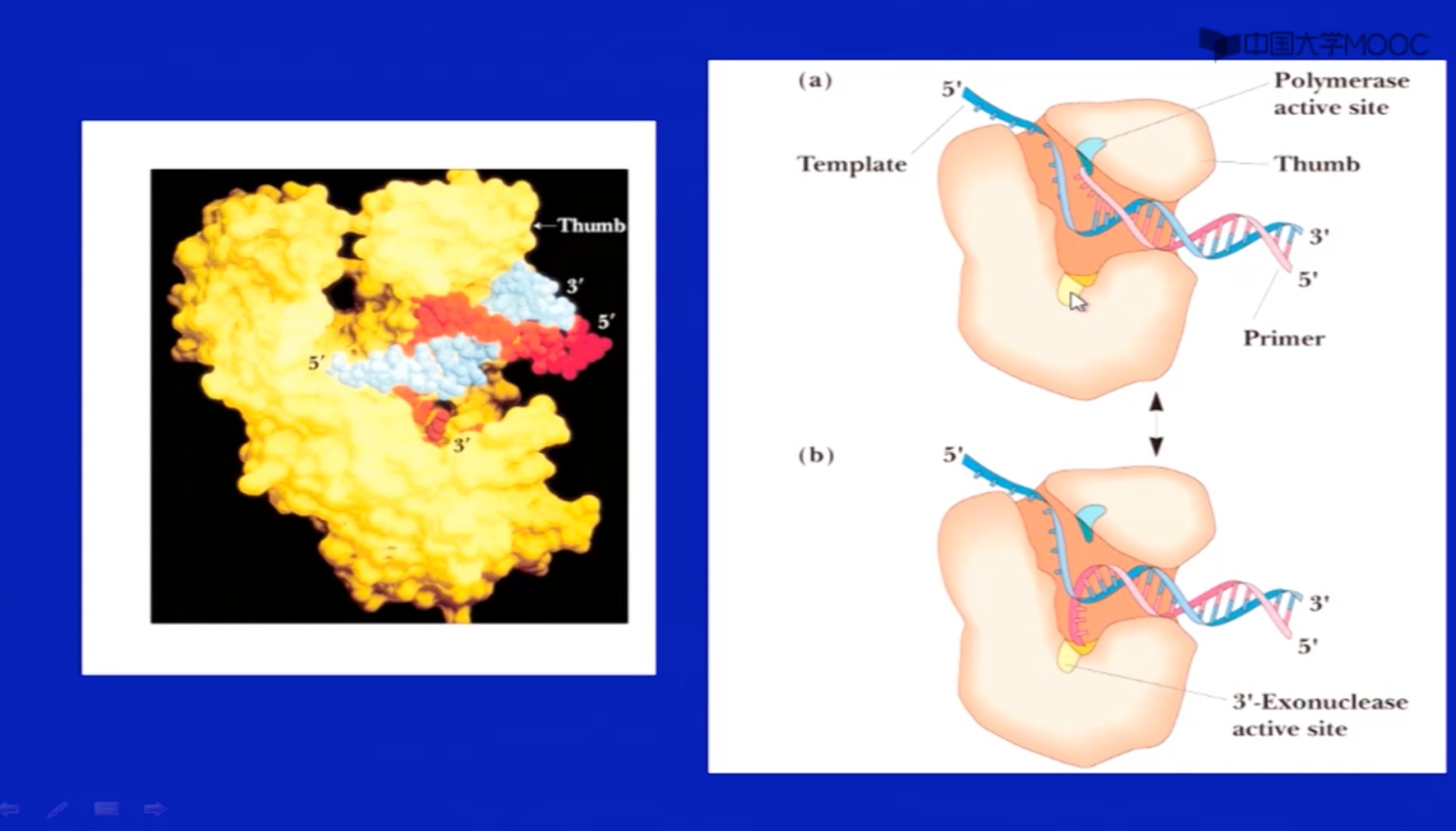
- 上面是聚合酶活性。下面是3‘外切酶活性。
- 出错的时候,3‘外切酶活性会被激活,聚合酶活性被抑制
- 正常延伸的时候,聚合酶活性会被激活,3‘外切酶活性被抑制
- 交替活动,形成校对作用
-
-
实际上,所有DNA聚合酶、RNA聚合酶的三维结构
都相似,由finger、palm、thumb三个结构域组成。 -
原核、真核、病毒的核酸聚合酶在这三个结构域的
序列和结构具有高度的保守性。 -
再讲一下5’ 核酸外切酶
- 刚才3’ 核酸外切酶只能从3’端开始一个一个核苷酸的切,且要是单链状态下的DNA,而5’ 核酸外切酶可以一个一个切,也可以一段一段切,且可以是单链状态也可以是互补配对状态
- 切刻平移
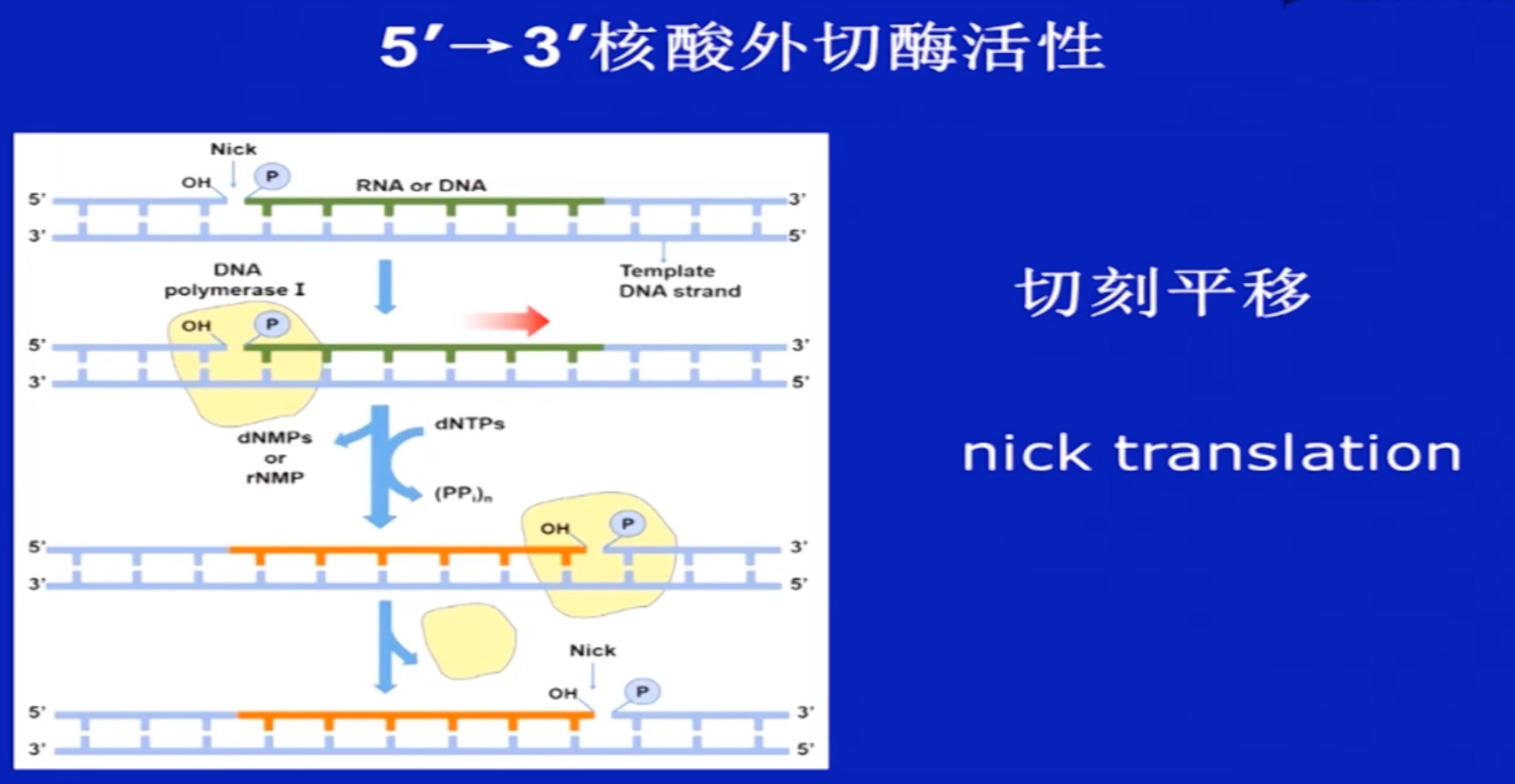
- 缺口填补
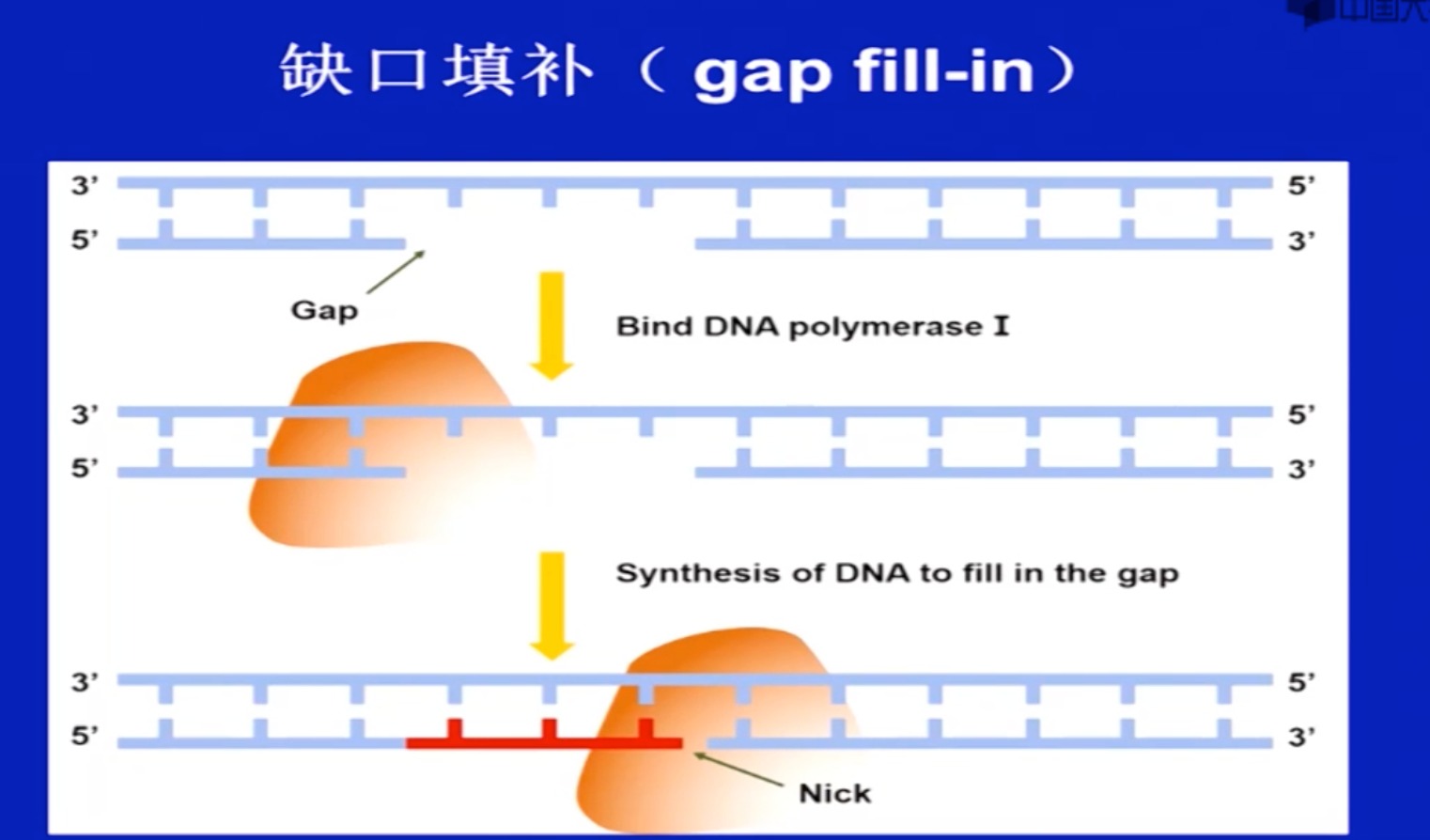
-
两种酶活性对比

-
DNA Polymerasel 在DNA复制中的作用
- 实验发现其进行性(一旦结合能复制的长度)不高
- 无法只靠这个酶就完成DNA复制
-
一系列研究发现其不是最主要的DNA聚合酶
-
DNA poll的主要生物功能
- DNA损伤修复(E.coli突变菌株 poIA-对UV非常敏感);
- Nick translation;
- Gap fill-in;
- Short lengths of DNA synthesis
1.2 DNA pol II
- 1970年分离得到;
- 聚合酶活性:2400 base/min;
- 100分子/cell;
- 有3’一5’核酸外切酶活性,没有5’一3’核酸外切酶活性;
- 在DNA repair 中起作用;
1.3 DNA pol III
-
1971年分离
-
多亚基;
-
聚合酶活性是DNA pol l的50倍;50,000base/min
10-20分子/cell; -
有3’一5’核酸外切酶活性没有5’一3’核酸外切酶活性;
-
高进行性;
-
这个酶才是重要发挥重要的,但是其含量低(导致发现的晚),活性高
-
结构

- 三个亚基靠 τ \tau τ亚基结合成一个核心酶,一个酶里有两个核心酶
- 钳载复合物:主要是帮 β \beta β亚基。装到模版链上和从模版链上解下来
-
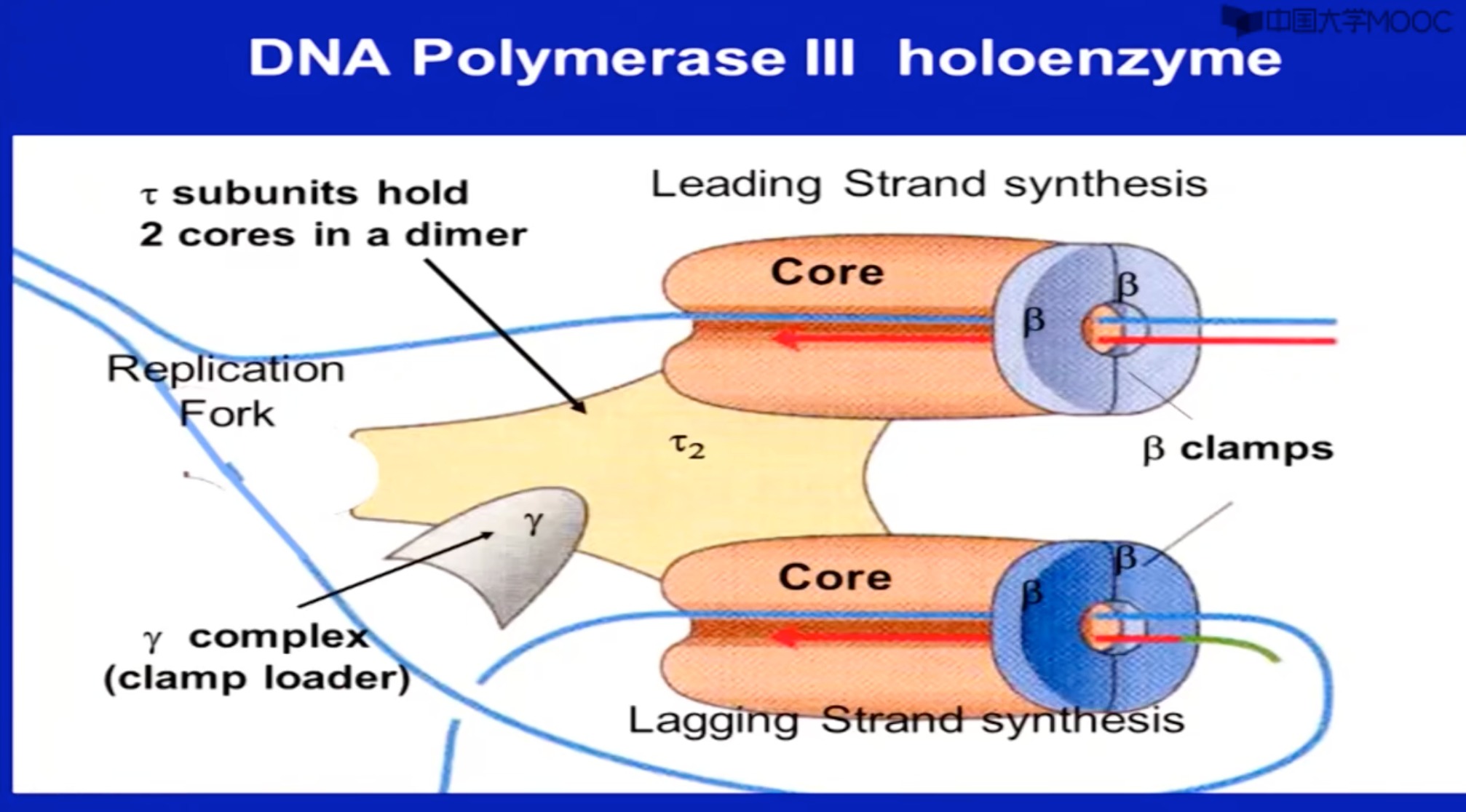
- 复制的地方只要一分子聚合酶3就够了,包含两个核心酶,各自负责一个子代的合成。
- 这两个核心酶通过
τ
\tau
τ了连在一起,说明其移动方向一样。但是两条模版链的复制方向不一样,必须是5‘到3’,那该怎么解决呢。
- 靠一个机制(后面会详细讲),可以看到图中下面那条DNA链是折回来的。以保证移动方向跟链的复制方向一致
-
聚合酶活性:
- DNA pol I holoenzyme催化DNA合成的速度在体外实验中可以达到700 nt/sec (E.coli 中DNA的合成速度是1000 nt/sec)
-
进行性:
- 在体外实验中,pol III的进行性可达 30 kb.
-
E.Col的一种温度敏感型突变菌株,在30度以卡可以正常生存,但是温度升高到45度时就难以生存;
- 原因:该菌株的polC基因 (DNApol ul a亚基)发生突变,在30度以下酶活性正常,在温度超过30度以后很容易变性失活;
-
证明DNA pol lll是E.coli(大肠杆菌)中真正的复制酶!
- II是参与DNA损伤修复、I主要是损伤修复,也参与复制
-

1.4 其它的DNA pol
- DNA pol IV 和DNA polV
- 1999年发现
- 易错的 (error-prone)聚合酶
- 在DNA损伤修复中起作用;
- DNA pol Iv在细菌生长稳定期表达;
- DNApol V在SOS反应时表达;
1.2 真核生物DNA聚合酶
包括以下五种

1.2.1 DNA pol α \alpha α
- 具有引发酶活性,负责合成RNA引物;
- 没有3’外切酶活性(无校对作用)(因为其主要是负责引物的合成,可以理解)
- 在复制起始区,先合成短的RNA引物(约10 Nt)再合成20~30 Nt的DNA,然后被pol8 和s取代:
1.2.2 DNA pol β \beta β
- 损伤修复
- DNA repair
- 最小的DNA pol;
- 进行性极低:
- 没有3"外切酶活性
1.2.3 DNA pol γ \gamma γ
- 在线粒体基质中发现;负责线粒体DNA的合成;
- 有3"外切酶活性;
1.2.4 DNA pol δ \delta δ
- 内在进行性低,但是有PCNA存在时进行性增加40倍,大大高于pola;
- PCNA ( proliferating cell nuclear antigen) 的一级结构与E.coli DNA pol 川的p亚基不同,但yeast PCNA三聚体结构与 β \beta βclamp非常相似;
- 有3"外切酶活性;
1.2.5 DNA pol ξ \xi ξ
- 内在进行性高:
- 有3’外切酶活性:

2 DNA解螺旋酶
- 原核生物和真核生物都有多种解螺旋酶;
- 促进DNA双链解开;
- 结合DNA:与碱基序列无关;多数优先结合单链区;
- 结合NTP:具有内在的依赖于DNA的 NTPase活性;水解NTP为解链提供能量;多数优先结合或只能结合ATP;
- 具有移位酶活性:沿着所结合的DNA单向移动,速度可达1000 nt/s;
- 解链酶的极性:3’解链酶,5’解链酶,双极性酶;到底哪一种才是真正参与解螺旋的呢
2.1 大肠杆菌中的解螺旋酶
-
最先在E.col中发现的三种解螺旋酶:rep helicase, helicase II、III,并未参与DNA复制。
-
从温度敏感型E.col中分离得到的DnaB才是在复制叉处解开DNA双链的解螺旋酶。
-
实验
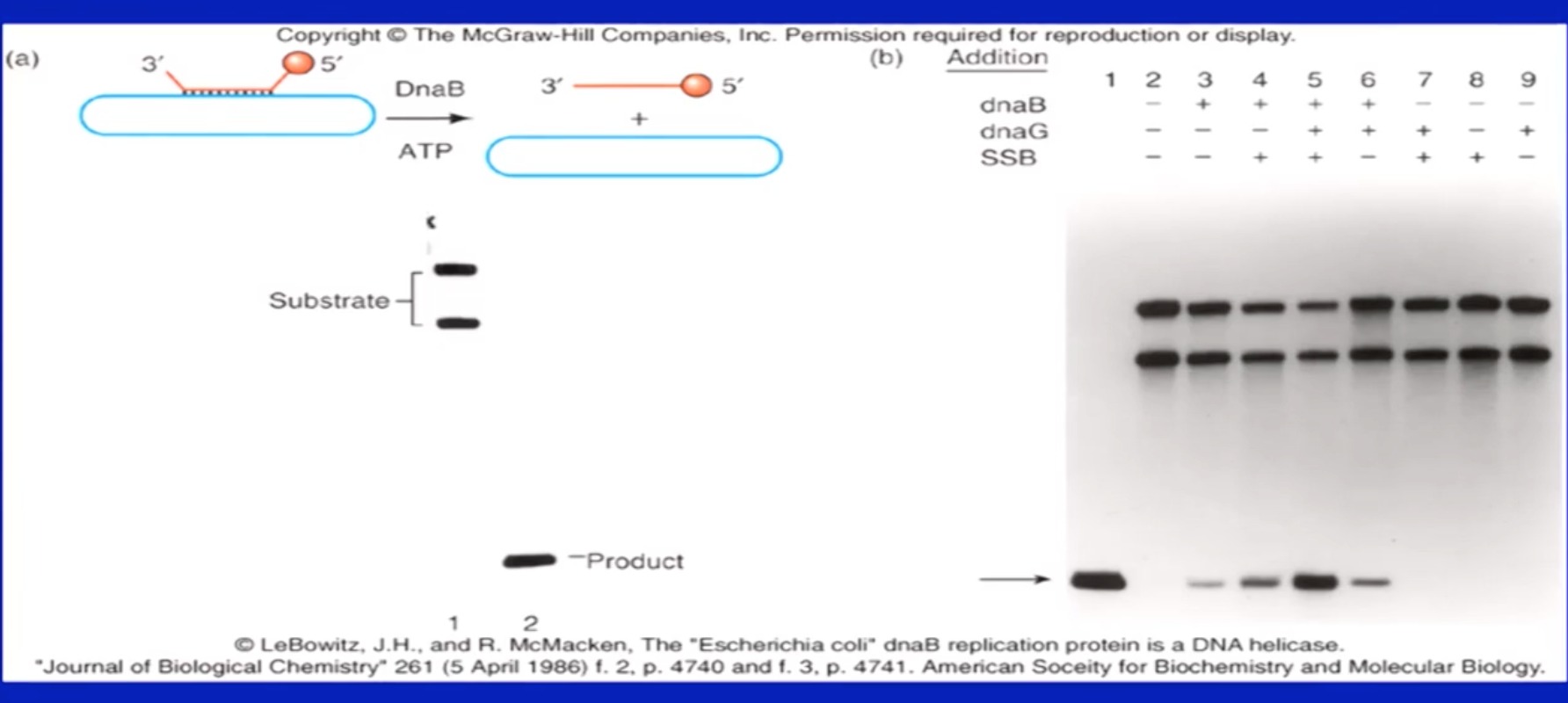
3 SSB
单链DNA结合蛋白
SSB, single-strand binding protein
- 选择性地与单链DNA结合:
- 防止单链DNA重新形成双链:
- 防止单链DNA的降解:
- 原核生物的SSB与单链DNA的结合具有协同性:
- 对于真核生物的SSB了解较少:已知一些真核生物病毒的DNA复制需要SSB的参与。
- 对原核生物的SSB了解比较多,SSB可以解螺旋后提供比较稳定的状态
4 DNA 拓扑异构酶
在复制前应该就发挥作用
- 复制叉的前进会带来额外的DNA扭曲张力(正超螺旋)
- 这种张力必需得到持续的释放,否则将最终影响DNA的复制。
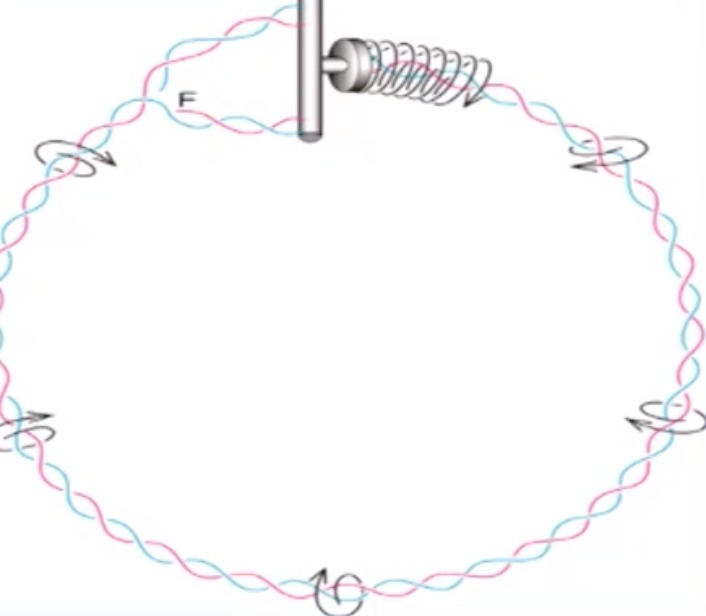
-
分类
- Type l: 催化DNA的一条链发生断製和再连接,不需要能量
- 切开,红线到了蓝线后面,减少了一次缠绕

- 切开,红线到了蓝线后面,减少了一次缠绕
- Type II: 催化DNA的两条健同时发生断裂和再连接,需要要ATP提供能量:
- 切了两条链
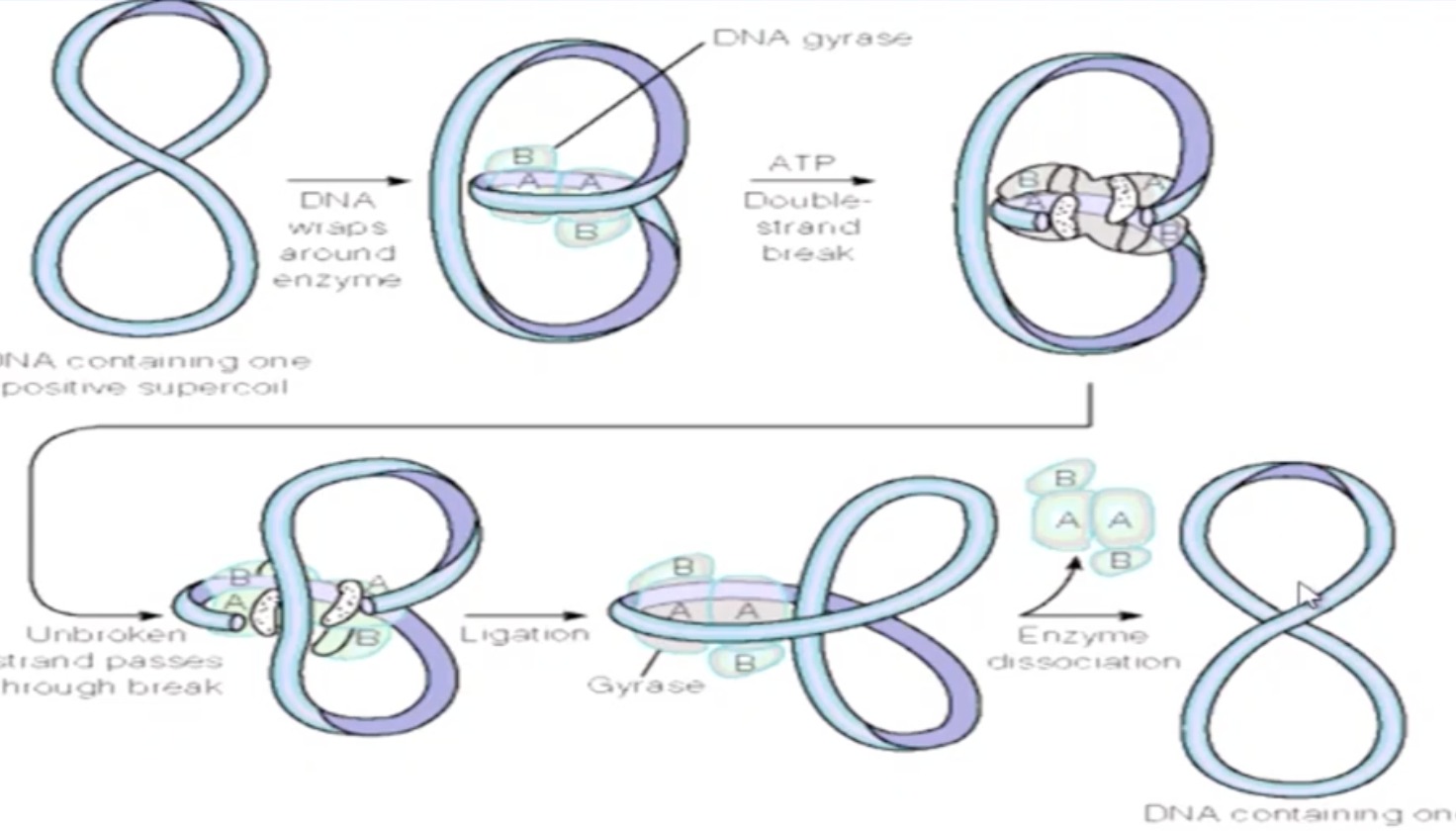
- 切了两条链
- Type l: 催化DNA的一条链发生断製和再连接,不需要能量
-
根据发现的顺序,我们命名为拓扑异构酶1、拓扑异构酶2、拓扑异构酶3,而类型1、2是根据原理来的,所以拓扑异构酶1可能是type1也可能是type2,这两个要分清楚
-
E.coli 依靠 DNA gyrase (type II topoisomerase
- 负责在复制过程中、在复制叉前消除由于复制叉前进
带来的扭曲张力:消除正超螺旋、引入负超螺旋; - DNA gyrase: A 2 B 2 A_{2}B_{2} A2B2
- 负责在复制过程中、在复制叉前消除由于复制叉前进
-
真核生物中由拓扑异构酶I负责清除复制叉前进带来的正超螺旋。
5 DNA引发酶
回顾:解螺旋酶消除张力,由解螺旋酶解开DNA,SSB结合到解开的单链上保护它,是不是下面就应该DNA聚合酶上去干活了呢
我们还需要引物,引物的合成需要特定的酶
- DNA的合成必需要有引物;
- 合成引物的酶称为:引发酶
(primase) - 合成引物的过程称为:引发
(priming); - 两条子链的合成都需要引发;
- 引物都是10-12 nt 的RNA;

-
实验
- 怎么证明引物是RNA而不是DNA?
- 怎么证明引物的长度?
-
实际上,包括大肠杆菌在内的多数原校生物不会利用自身的RNA聚合酶来合成引物,而是通过专门的酶系来完成引物的合成(一个复合物)。
E.coli 及其大多数噬菌体使用 Dna G 为 primase:
但合成引物还需要多种蛋白质的参与:
这些蛋白质组裝成预引发体:
预引发体与引发酶组装成引发体;
由引发体负责引物的合成。

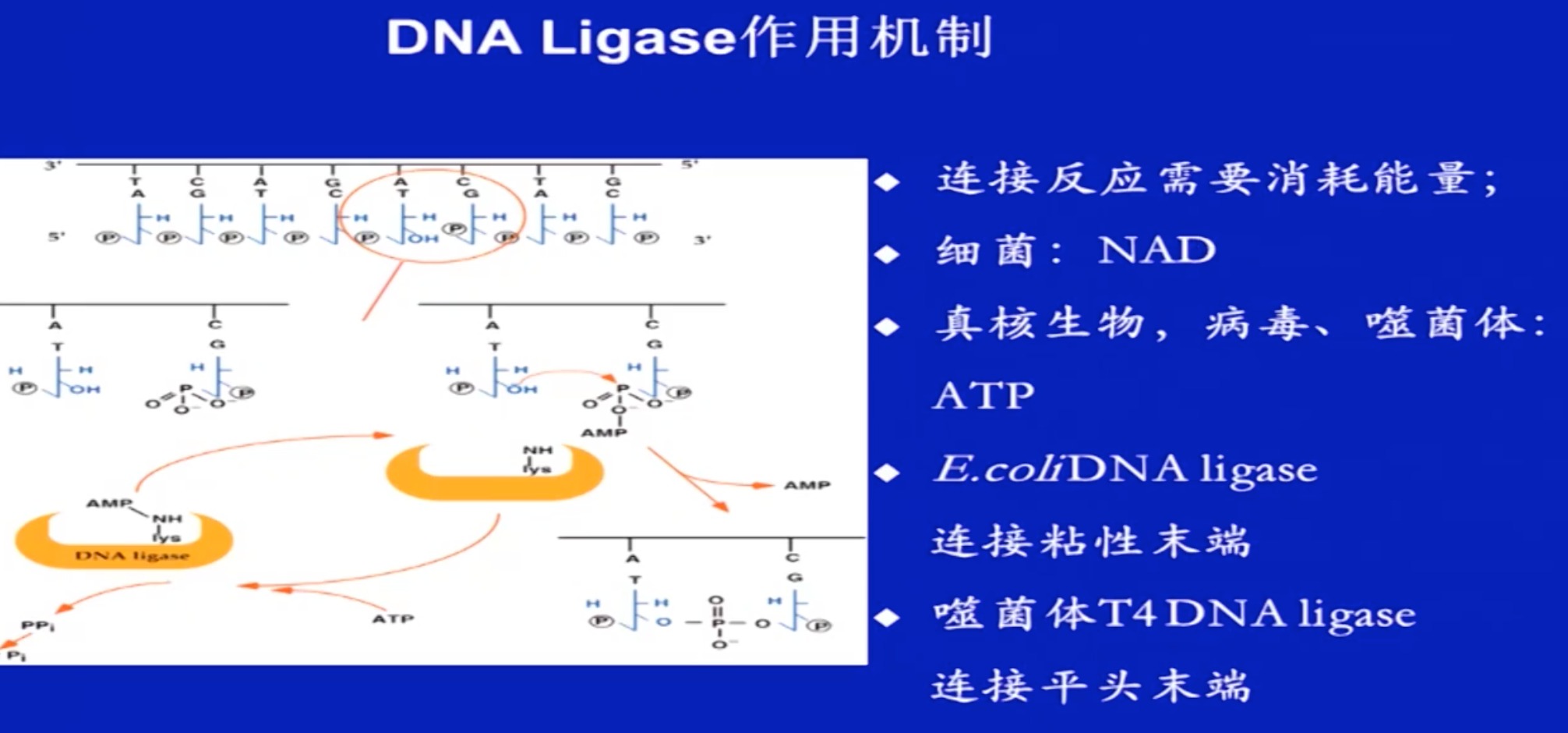
6 DNA 连接酶
- 1967年发现:
- 催化一条链的5’-磷酸末端与另一条链的3’-OH末端之间形成磷酸二酯健。
- 只能连接处于nick状态的双链DNA,不能连接游离的两条单链DNA。
第三节 DNA复制的详细机制
1 复制的起始
-
复制原点的特征(原核、真核)
-
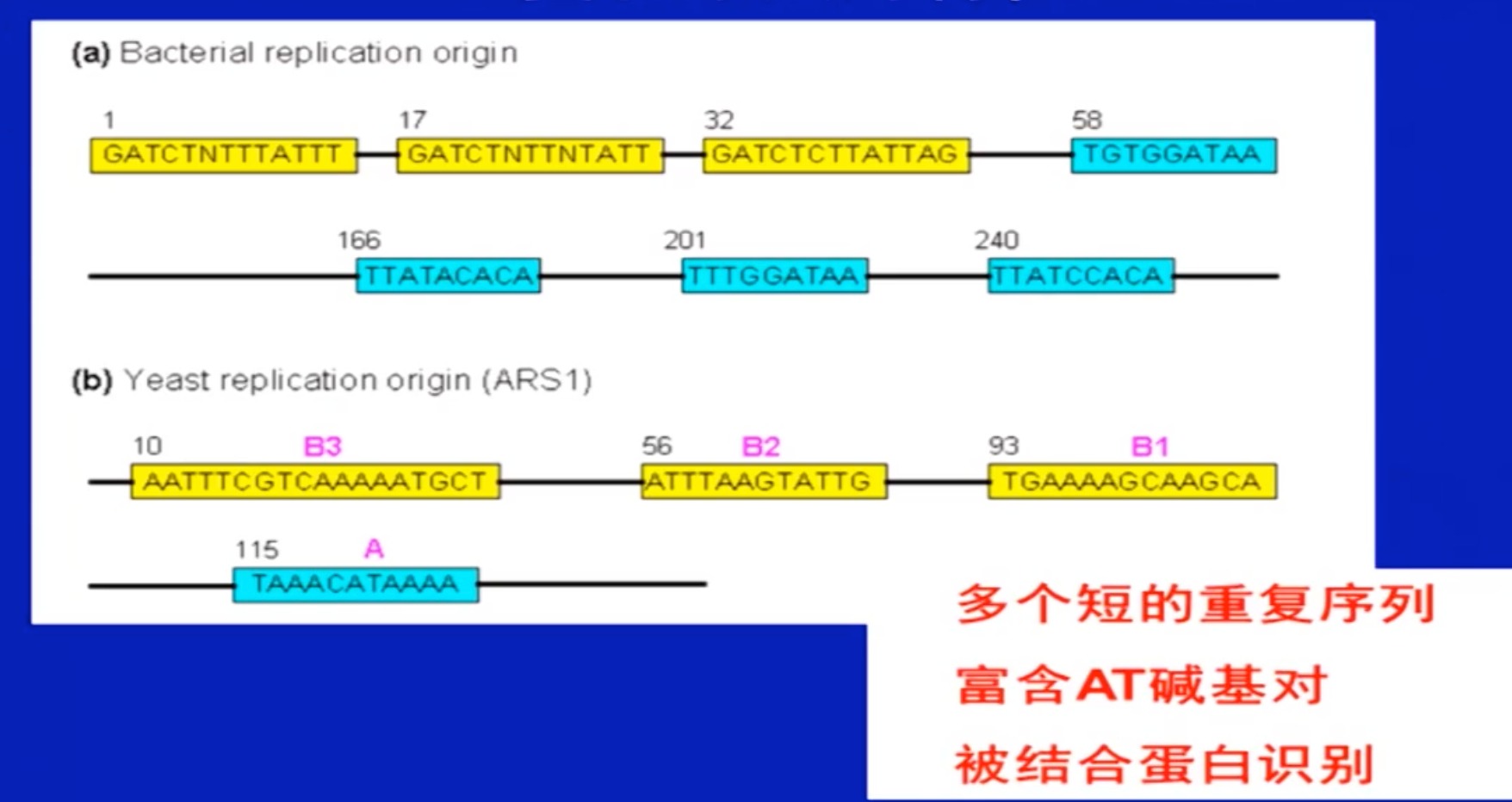
-
DNA复制原点富含AT碱基对,更容易出现所谓呼吸现象”,即氢键迅速断裂与再生,导致DNA两条链不断解链与聚合、形成瞬间的单链泡状结构。
-
DNA结合蛋白与复制原点处富含AT碱基对的重复序列的相互作用,促进了DNA复制的起始。
-
大肠杆菌、真核生物、酵母
- 不同的复制起始区各有特点
2 复制的延伸
-
原核生物中主要依靠DNA pol III负责复制过程中DNA链的延伸;
-
真核生物中主要依靠DNA pol δ \delta δ负责复制过程中DNA链的延伸;
-
原核生物DNA复制的延伸
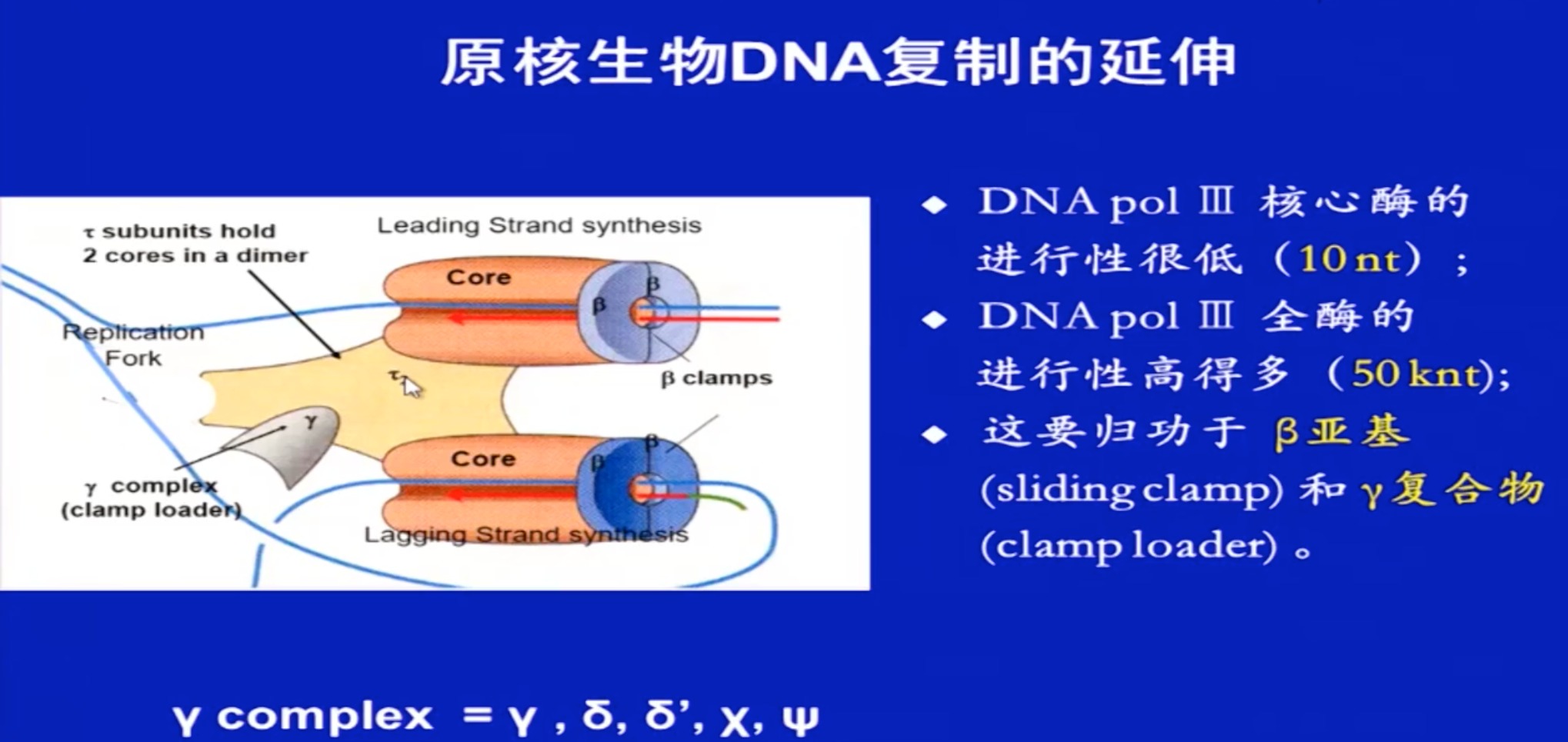
β \beta β推动酶沿着模版链滑动
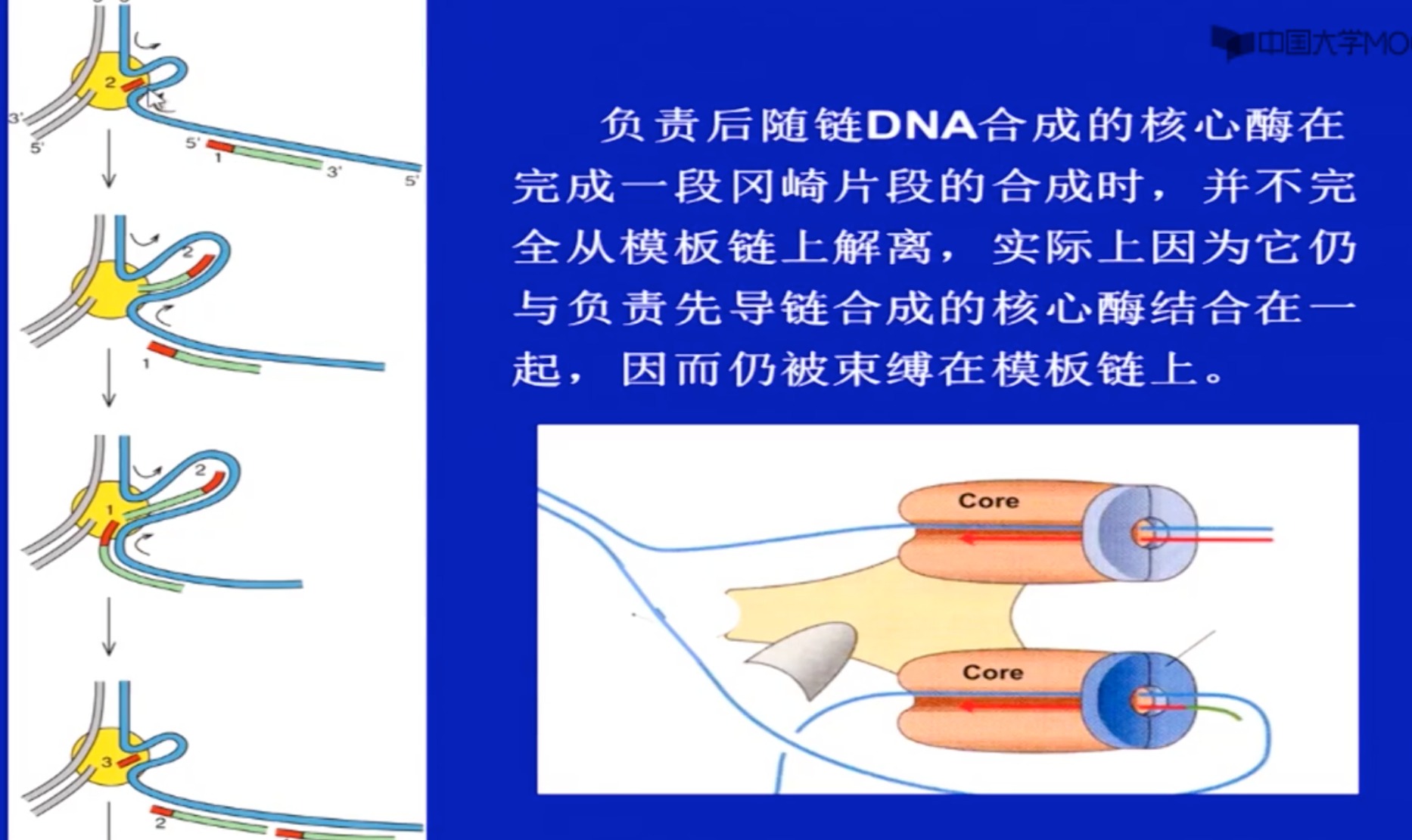
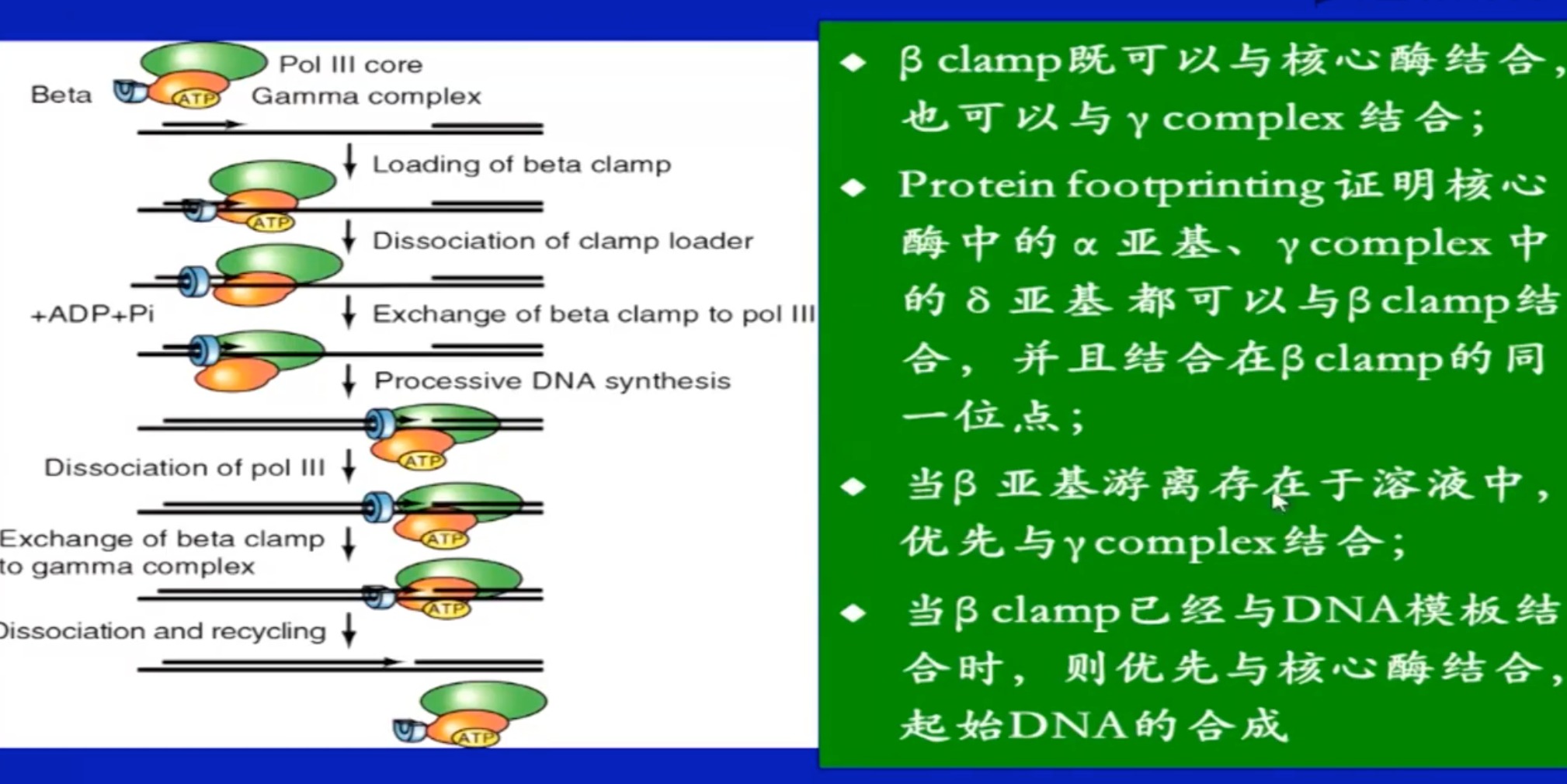
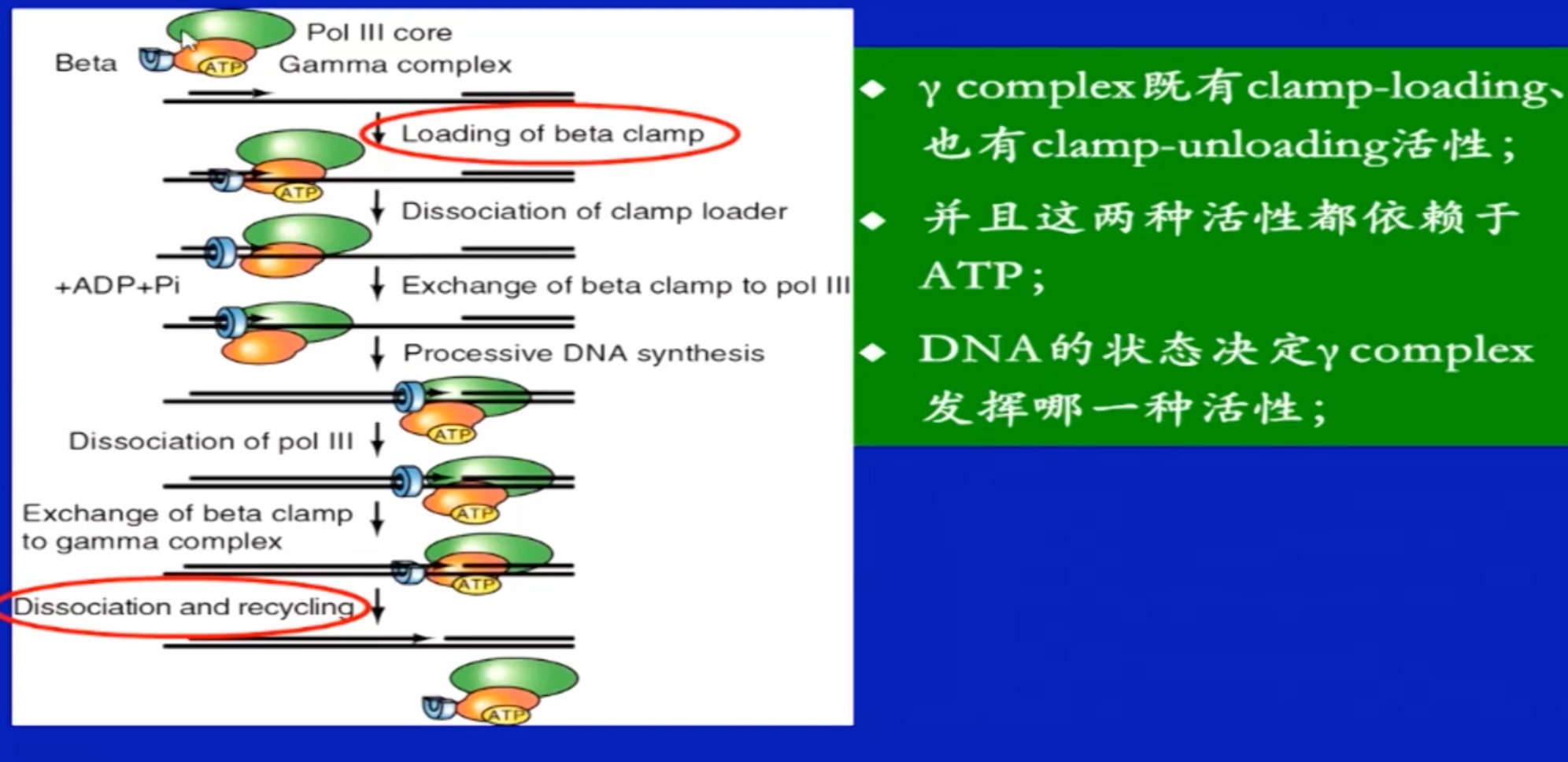
取决于三者的相互作用
- 真核生物DNA复制的延伸

3 复制的终止
滚环复制
- DNA上存在一些位点、与DNA复制的终止有关
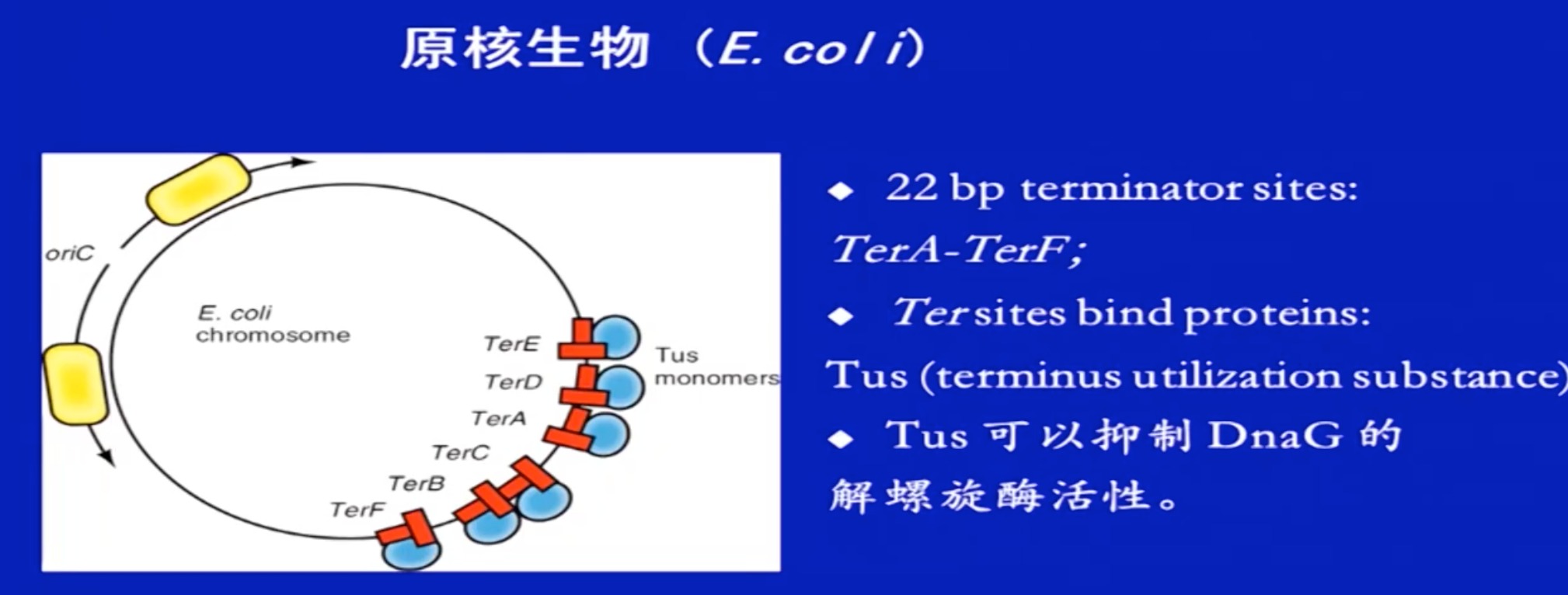
- 需要依靠拓扑异构酶去环化
4 冈崎片段的连接

DNA聚合酶和连接酶共同完成
5 端粒和端粒酶
对于真核生物来讲,真核生物的DNA合成面临末端(5‘端)菱缩的问题。
原核双链环状没有问题,而真核是双链线状
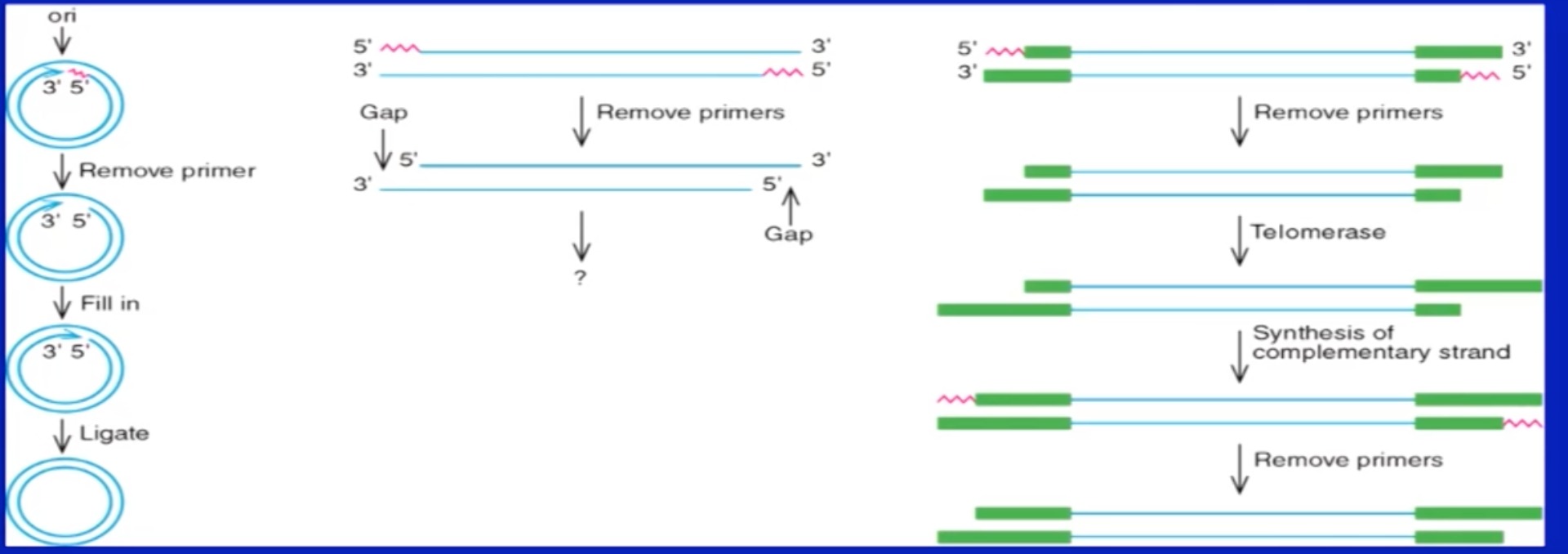
每复制一次,5‘端会少掉一点,虽然真核生物DNA5’端是无用的序列(冗余序列,其实就是端粒),但是总有一定减少的限制,所以真核生物细胞复制的轮数是有限的(当然实际情况更复杂,很多时候是中间有多个复制起点)
-
端粒和端粒酶:
-
Telomere and telomerase
-
端粒:真核生物染色体末端、短的、富含GC的重复序列:
-
端粒酶:在DNA末端添加端粒序列的酶;
-
端粒序列具有种属特异性:
-
tetrahymena:
TTGGGG
AACCCC -
vertebrates(human): TTAGGG
AATCCC
-
-
-
端粒酶本质上是逆转录酶

- 不同细胞中端粒酶活性不同,端粒长度也不同;
- 生殖细胞、肿瘤细胞中端粒酶活性高、端粒长;
- 体细胞中端粒酶活性很低、长度短;
- 正常细胞的寿命一定,称为“te Hayflick limit”
- 肿瘤细胞中端粒酶活性被激活,因此具有无限增殖的能力;
- 延长寿命的希望、治疗肿瘤的方式之一
![深度学习基础入门篇[五]:交叉熵损失函数、MSE、CTC损失适用于字识别语音等序列问题、Balanced L1 Loss适用于目标检测](https://img-blog.csdnimg.cn/img_convert/2f4a3a85fed9e7c3b0a05c2c177ea979.png)







![[STM32F103C8T6]基于stm32的循迹,跟随,避障智能小车](https://img-blog.csdnimg.cn/45c3ffdf5d3b490cab04da518cadbc29.png)