1. 基因注释
到目前为止,我们一直在处理对应于转录因子结合的 ChIPseq 峰。顾名思义,转录因子可以影响其靶基因的表达。
转录因子的目标很难单独从 ChIPseq 数据中确定,因此我们通常会通过一组简单的规则来注释基因的峰:
如果峰与基因重叠,则通常将峰注释为基因。
2. Peak 注释
ChIPseeker 是一个有用的基因峰注释包。通过在小鼠 TXDB 对象(mm10 基因组)的来源中使用预定义的注释,ChIPseeker 将为我们提供峰落在基因中的位置以及到 TSS 位点的距离的概览。
首先加载下一部分所需的库。
library(TxDb.Mmusculus.UCSC.mm10.knownGene)
library(org.Mm.eg.db)
library(GenomeInfoDb)
library(ChIPseeker)
annotatePeak 函数接受要注释的区域的 GRanges 对象、基因位置的 TXDB 对象和要从中检索基因名称的数据库对象名称。
peakAnno <- annotatePeak(macsPeaks_GR, tssRegion = c(-500, 500), TxDb = TxDb.Mmusculus.UCSC.mm10.knownGene,
annoDb = "org.Mm.eg.db")
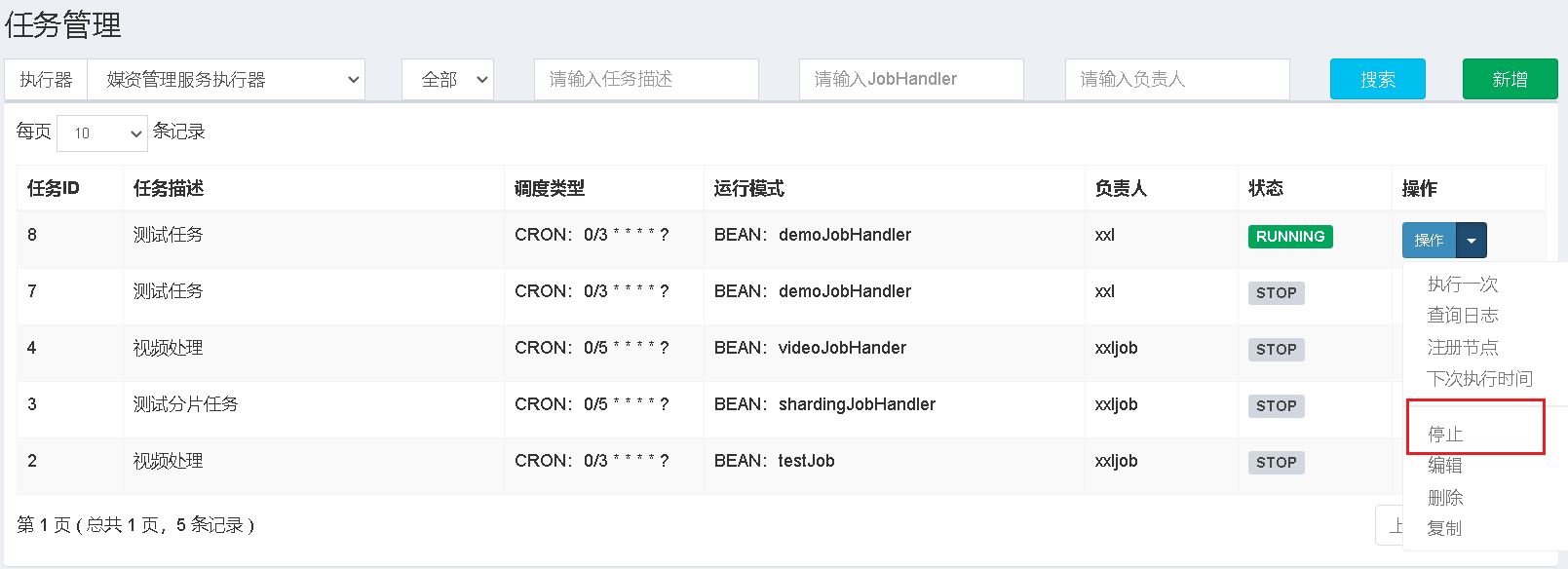
class(peakAnno)

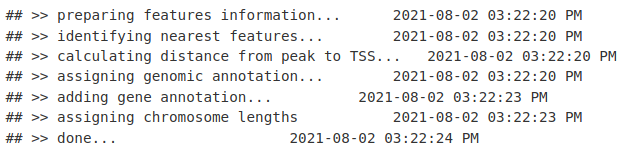
结果是一个包含峰注释和整体注释统计信息的 csAnno 对象。
peakAnno
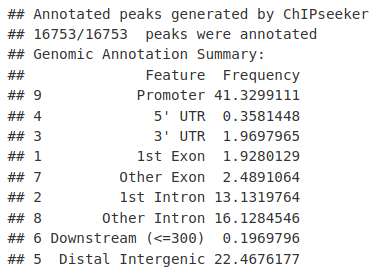
csAnno 对象包含有关基因的单个峰的注释信息。要从 csAnno 对象中提取它,ChIPseeker 函数 as.GRanges 或 as.data.frame 可用于生成具有峰及其相关基因的相应对象。
peakAnno_GR <- as.GRanges(peakAnno)
peakAnno_DF <- as.data.frame(peakAnno)
peakAnno_GR[1:2, ]
3. 可视化 Peak 注释
现在我们有了来自 ChIPseeker 的注释峰,我们可以使用 ChIPseeker 的一些绘图功能来显示基因特征中峰的分布。在这里,我们使用 plotAnnoBar 函数将其绘制为条形图,但 plotAnnoPie 会生成类似于饼图的图。
plotAnnoBar(peakAnno)
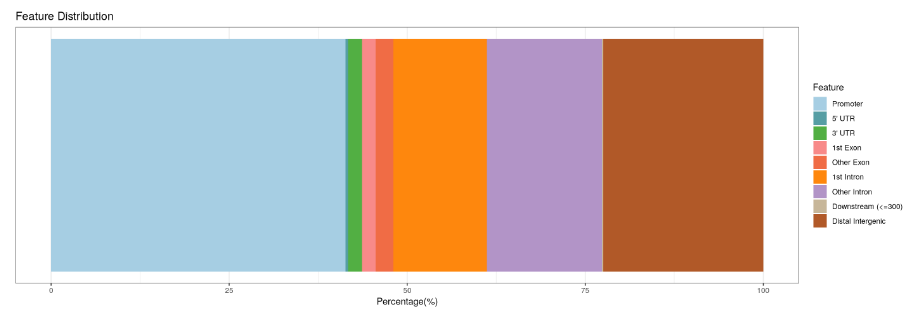
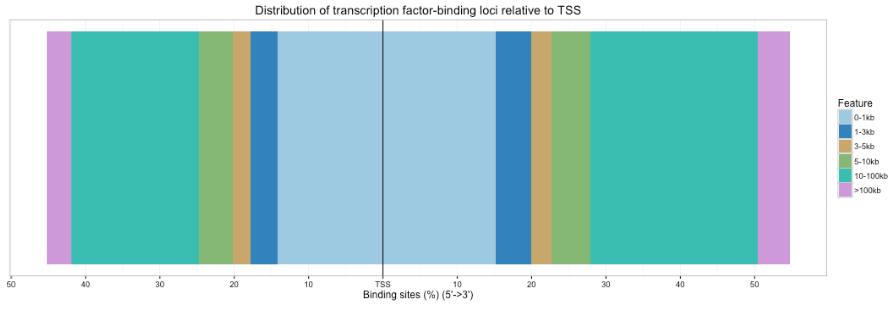
同样,我们可以绘制 TSS 站点周围峰值的分布。
plotDistToTSS(peakAnno)
ChIPseeker 还可以提供一个简洁的图来描述注释之间的重叠。
upsetplot(peakAnno, vennpie = F)

本文由 mdnice 多平台发布