目录
- 引言
- 网络架构发展
- 直接生成蛋白-蛋白复合物
DeepMind在最近的CASP14上展示了非常准确的预测。作者探索了融合相关思想的网络架构,并通过对一维序列级、二维距离图级(distance map)和三维坐标级(coordinate)的信息依次进行转换和集成的3-track网络获得了最佳性能。该网络产生的结构预测精度接近DeepMind在CASP14中的预测精度,能够快速解决具有挑战性的x射线晶体学和冷冻电子显微镜结构建模问题,并提供对目前未知结构的蛋白质功能的见解。该网络还能够仅从序列信息快速生成准确的蛋白质-蛋白质复合物模型,缩短了需要对单个亚基建模然后对接的传统方法。
引言
仅从氨基酸序列信息(amino acid sequence)预测蛋白质结构(protein structure)一直是一个挑战。历届CASP已经证明,从PDB(protein data bank)中已知蛋白质结构的大型数据库中提取信息的 AlphaFold 和 trRosetta 等深度学习方法优于那些明确建模折叠过程的传统方法。DeepMind的 AlphaFold 2 在最近的CASP14上的表现让科学界迫切想要了解除了论文中描述的框架细节,并提出问题:这样的方法能否在小规模公司中实现同样的复现效果。
在CASP14中,AlphaFold 2的进步包括:
- 从多个序列对齐(MSA,multiple sequence alignments)开始,而不是从多特征处理开始;
- 用注意力机制取代2D 卷积,该机制更好地表示序列中远距离残基之间的关系;
- 使用2-track架构,其中1D序列和2D距离图的信息被迭代转换;
- 使用SE(3)等变transformer从2-track网络直接细化生成原子坐标;
- 端到端学习,从最终生成的3D坐标通过所有网络层方向传播到输入序列优化所有网络参数;
网络架构发展
被DeepMind的结果所吸引,并以提高结构生物学研究的蛋白质结构预测精度和推进蛋白质设计为目标。作者在没有开源方法的情况下,尝试了各种各样的方法在网络的不同部分之间传递信息,最终成功获得一个2-track网络,信息沿1D序列对齐轨道和2D距离图轨道并行流动,其性能明显优于trRosetta(CASP14中次优于AlphaFold 2的方法)。
作者认为,通过扩展到在3D坐标空间中操作的第三个轨道,在序列,残基距离和方向,以及原子坐标之间提供更紧密的连接,可以获得更好的性能。作者构建了2-track模型的双层架构,并在三维backbone坐标上增加了第三个并行轨道,如图1a所示。在该架构中,信息在1D氨基酸序列,2D距离图,3D坐标之间流动。

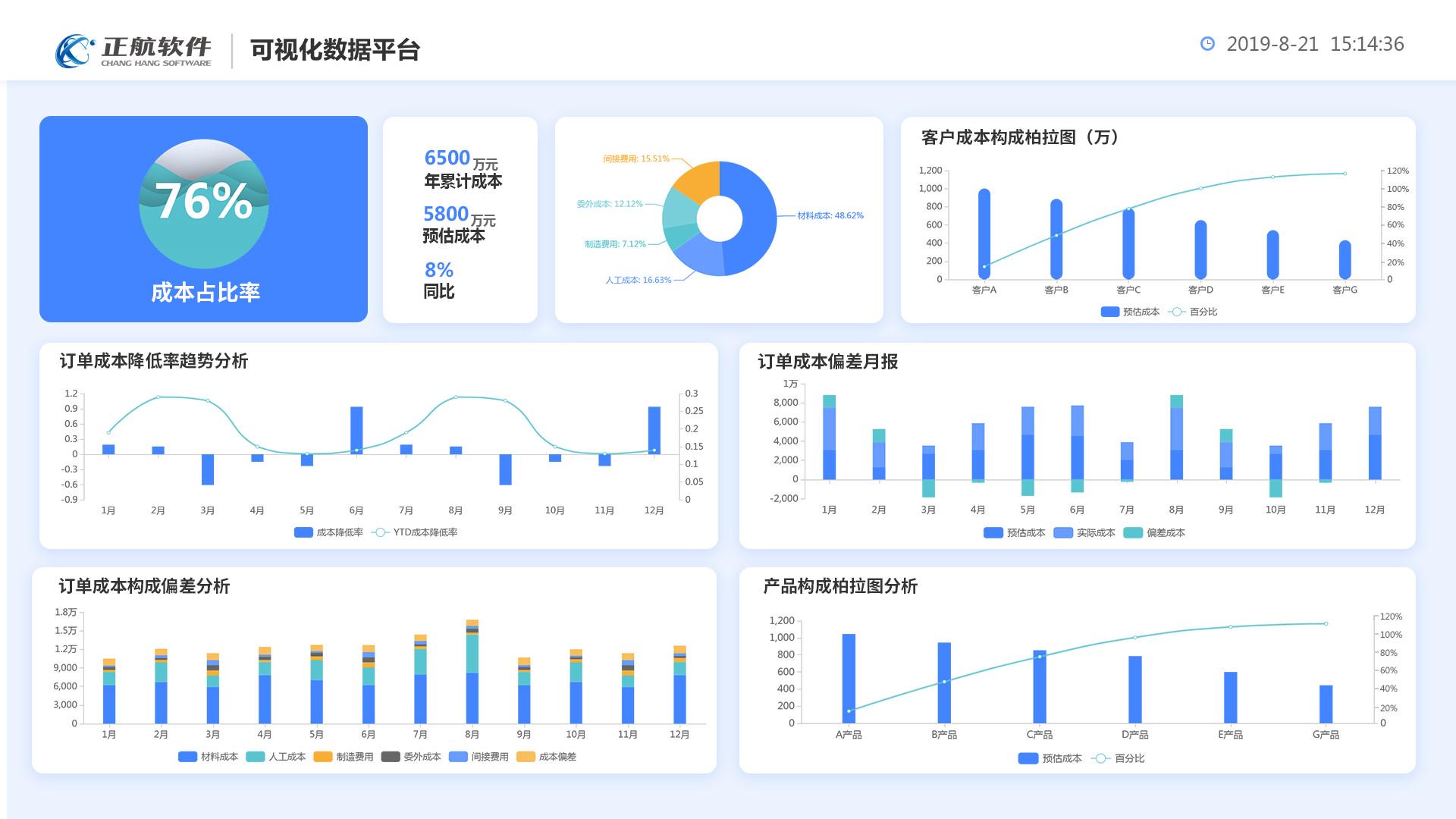
- 图1a:RoseTTAFold架构,具有1D、2D和3D注意力轨道。轨道之间的多个连接允许网络同时学习序列、距离和坐标内部和之间的关系。
- 图1b:预测方法在CASP14靶点上的tm平均得分。Zhang-server和BAKER- rosettaserver是排名前两位的服务器组,而AlphaFold2和BAKER是CASP14中排名前两位的human组;BAKER- rosettaserver和BAKER的预测是基于trRosetta的。使用双轨模型和RoseTTAFold(端到端和pyRosetta版本)进行的预测是完全自动化的。
- 图1c:预测方法在CAMEO靶点上的tm平均得分。
相比之下,在2-track的AlphaFold 2中,关于3D原子坐标的推理发生在1D和2D信息处理完成之后。由于计算机硬件的限制,我们不能直接在大型蛋白质上训练模型,因为3-track模型有数百万的参数;相反,作者向网络提供输入序列的不连续crops,这些crops由两个不连续序列组成,共跨越260个残基。为了得到最终的模型,作者结合并平均了每种crops的1D特征和2D距离和方向预测,然后使用两种方法生成最终的3D结构。首先,将预测的残基距离和方向分布输入pyRosetta以生成所有原子。其次,平均的1D和2D特征被输入SE(3)-等变层,经过从氨基酸序列到3D坐标的端到端训练,由网络直接生成坐标。作者将这些生成每个残基精度预测的网络称为RoseTTAFold。第一步方法的优点是在推理时需要低内存gpu,并且可以生成完整的侧链模型,但需要CPU花时间来进行pyRosetta结构建模。
对于初始3D结构预测,采用基于Graph Transformer的架构来生成3D结构的初始骨干坐标。输入定义为一个全连通图,其中节点表示蛋白质中的残基。预测每个节点(残基)的笛卡尔坐标。然后利用SE(3)-等变transformer进行结构更新。
直接生成蛋白-蛋白复合物
文章提出的3轨道网络的端到端版本的最后一层通过结合蛋白质序列的不连续(蛋白质的两个片段之间有断裂)特征来生成3D结构模型。文章推断,由于该网络可以无缝地处理链断裂,它可能能够直接从序列信息去预测蛋白质-蛋白质复合物的结构。与其向网络提供单个蛋白质的序列,不如输入两个或多个序列,输出两个或多个蛋白质链的骨干坐标。因此,该网络能够从序列信息直接构建蛋白质-蛋白质复合物的结构模型,缩短了为单个亚基构建模型然后进行刚体对接的过程。本文对包含两条(图2A)或三条链(图2B)的已知结构复合体的MSA测试了端到端3轨道网络,在许多情况下,得到的模型非常接近实际结构(TM-score>0.8)。

- 图2A和B从序列信息预测大肠杆菌蛋白复合物的结构。实验确定的结构在左边。RoseTTAFold模型的预测在右边;下面的tm分数表示结构相似的程度。两个链复合体如图A所示。第一个亚基为灰色,第二个亚基为彩虹色,从蓝色(N端)到红色(C端)。B中显示了三个链复合体。
- 图2C:RoseTTAFold生成的IL-12R-IL-12复合物结构符合先前发表的低温电镜密度。


![[python入门㊺] - 异常中的finally](https://img-blog.csdnimg.cn/b17f10e975de4e8b9fe0cd64604e6a6f.png)