文章目录
- 前言
- 一、如何使用TCGA数据库获取公共数据?
- 二、使用步骤
- 1.点击Cohort Builder
- 2.数据筛选
- 3. Repository
- 4.数据下载
- 4.1 继续选择筛选条件
- 4.2 添加cart并进入
- 4.3 下载
- 总结
前言
TCGA 全称 The Cancer Genome Atlas ,即癌症基因组图谱。它是一个大型的癌症研究项目,旨在通过对大量癌症患者的样本进行全面的基因组分析,来深入了解癌症的发生、发展机制。TCGA 收集了多种癌症类型的样本,包括肿瘤组织和正常组织,并对这些样本进行了基因测序、基因表达分析、DNA 甲基化分析等多种组学层面的研究。TCGA数据库中包含了多种肿瘤类型的数据,如乳腺癌、肺癌、结直肠癌、肾癌等,其中每个肿瘤类型的数据都是由不同实验室和机构采集和上传的,数据涵盖了基因组、转录组、表观基因组和临床等多个层面,可以用于基因组学、生物信息学和医学领域的研究和应用。
通过 TCGA 产生的数据,科学家们能够发现新的癌症相关基因、了解癌症的分子亚型、探索癌症治疗的潜在靶点等,这对于癌症的诊断、治疗和预防都具有重要的意义。
一、如何使用TCGA数据库获取公共数据?
- url: TCGA数据下载挂网
- 通过条件筛选过滤数据
- 下载数据
二、使用步骤
1.点击Cohort Builder

2.数据筛选
我们这里以肺腺癌的数据下载为例:
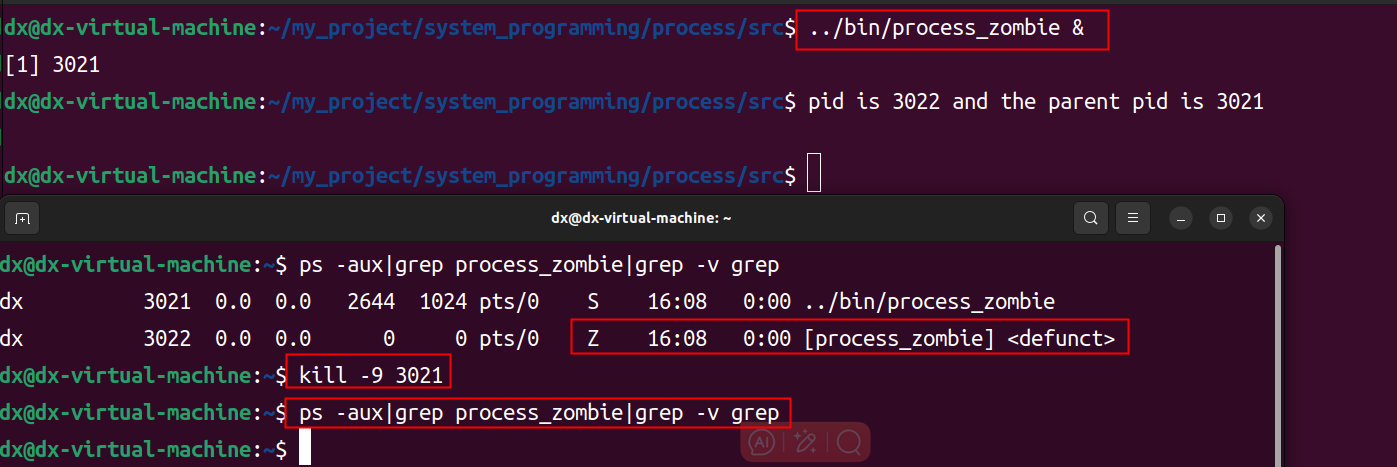
注意: GDC只是一个数据存储网站,因此不仅有TCGA的数据存储在这里,其他项目比如 APOLLO等等的数据也都收录在GDC


条件添加完成之后我们就可以看到筛选到517个样本的数据
3. Repository
在program中点击more选择TCGA项目,在project中选择不同癌种项目类型,也可以根据列出的其他样本信息进一步筛选,筛选完成点击Repository

4.数据下载
4.1 继续选择筛选条件
进入Repository后进一步进行数据类型筛选,Experimental Strategy选择RNA-seq,Data Type选择Gene Expression Quantification,然后点击Add All Files to Cart,此时上方购物车图标会显示已将524个要下载的gene_counts文件加入购物车,点击这个购物车图标进入下一个页面

4.2 添加cart并进入

4.3 下载
注意: 一般下载三个文件 – Cart,Clinical:TSV,Sample sheet
- Clinical文件解压后包含每个样本的临床信息
- Cart文件解压后即是对应每个样本通过star对比后的gene_counts文件,一个样本一个文件夹
- sample_sheet文件包含对应Cart解压后文件夹和样本对应的信息


总结
以上就是TCGA数据库下载数据的基本流程了,关于TCGA差异分析可跳转进行观看!





![Powerpaint介绍及实现局部重绘效果测试[comfyui]](https://i-blog.csdnimg.cn/direct/5238a8ad180447c8a46d9857e3f42121.png)