小罗碎碎念
本期推文主题:乳腺癌
最近我在思考一个问题,生信分析和人工智能的区别和联系是什么?细节现在是想不清楚的,因为我的知识储备还不够,但是有一点我可以确定,二者的部分本质都是一样的——强大的计算工具——试图通过借助现有的先进计算工具来辅助我们挖掘更深层次的信息。

目前来看,有一部分人是不认可医学AI的,觉得医学AI这东西只能水水论文。这确实是目前的大多数论文的一个现状,但是我们真正投入到这个研究中的人才能亲切的感受到这背后蕴藏的趋势——随着时间的推移,落地只是时间问题,等你发现需要投入大量精力的时候,已经来不及转型了——其他领域也有过很多类似的历史经验教训。
AI的本质是一个工具,纵观历史上下几千年,后来居上者从来不是某一个技术,而是掌握这个技术的同行们。当自诩天朝大国的乾隆把外国进献的先进武器丢在茅厕的时候,就应该预料到未来的某一天长缨大枪会倒在大炮下。
上面这两段话,短短几百字,确浓缩了很多人目前的处境——课题边缘化。其实冷静下来思考一下,大佬之所以能站在今天的位置,大部分还是有自己独到的眼光和能力的。“外行”指导“内行”,这事太普遍了,因为到了硕士博士阶段,都是独立负责一个方向了,导师很难做到每个方向都顾及。我们在心里吐槽老板不理解自己的时候,是不是也要反思一下,自己现在是否做出了值得重视的成果?是否称得上内行?
最后用一句我前两天分享在朋友圈的话结尾,与大家共勉——价值是自己证明的,不是靠别人赋予的。
一、人工智能驱动的组学技术在三阴性乳腺癌焦亡治疗药物配对发现中的应用

一作&通讯
| 作者类型 | 作者姓名 | 单位名称(中文) |
|---|---|---|
| 第一作者 | 欧阳 | 复旦大学药学院药剂学系,智能药物递送教育部重点实验室 |
| 第一作者 | 单 | 复旦大学药学院药剂学系,智能药物递送教育部重点实验室 |
| 第一作者 | 申 | 上海复旦大学附属肿瘤医院药剂科,复旦大学浦东医学中心 |
| 通讯作者 | 彭 | 广东省肿瘤介入诊断治疗重点实验室,珠海市人民医院 |
| 通讯作者 | 张 | 复旦大学附属华东医院放射科 |
| 通讯作者 | 王 | 复旦大学药学院药剂学系,智能药物递送教育部重点实验室 |
| 通讯作者 | 李 | 微软亚洲研究院 |
| 通讯作者 | 庞 | 复旦大学药学院药剂学系,智能药物递送教育部重点实验室 |
文献概述
这篇文章利用人工智能和组学技术发现了针对三阴性乳腺癌的焦亡疗法药物组合,为开发新型抗癌药物提供了创新策略。
焦亡是一种由Gasdermin (GSDM)蛋白家族介导的细胞死亡方式,可以引发强大的抗肿瘤免疫反应。研究者们利用大数据分析和药物数据库,建立了一个生物因子调控的神经网络(BFRegNN),用于快速筛选和优化焦亡药物组合。他们特别关注了米托蒽醌(mitoxantrone, MIT)和藤黄酸(gambogic acid, GA)的组合,并制备了仿生纳米晶体(MG@PM),以实现合理药物递送。
研究发现,MG@PM纳米晶体通过调节焦亡基因和触发焦亡级联免疫效应,在TNBC模型中显示出独特的作用机制。该研究提出了一个基于组学的目标智能复合药物发现框架,探索了一种创新的药物开发范式,重新利用现有药物,实现对难治性疾病的精准治疗。
文章还讨论了药物开发中的痛点,如低成功率、高成本和长周期,以及如何利用数据科学技术改变现状,开启智能药物开发的新时代。研究者们通过生物信息学分析、人工智能和实验验证的整合,为复合药物开发提供了一个强大的技术平台。
此外,文章还提到了研究的伦理声明、使用的材料、细胞培养、动物实验、药物候选者的探索、BFRegNN的介绍和应用、纳米晶体的制备和表征、细胞摄取、细胞毒性测试、药物效果预测、蛋白质活性测定、药理学和生物分布研究、抗肿瘤效果、体内免疫刺激实验、体内抗转移研究以及统计和可重复性分析。
代码&数据
-
代码链接:
- BFReg-NN developments: https://github.com/BoshuOuyang/BFregNN-Cox-forpyroptosis-in-TNBC/
- 作用:这是BFReg-NN(Biological Factor Regulatory Neural Network)的代码库,用于构建和训练神经网络模型,以便在研究中预测药物组合对三阴性乳腺癌治疗的效果。BFReg-NN是一个基于现有生物学知识的神经网络架构,它通过图神经网络和消息传递机制来更新和学习生物因子(如基因、蛋白质)之间的相互作用,从而预测药物对疾病治疗的影响。
-
数据集:
- Fudan University Shanghai Cancer Center TNBC datasets (FUSCCTNBC): [http://www.biosino.org/; OEP000155 node; GEO: GSE118527; SRA: SRP157974; figshare: https://doi.org/10.6084/m9.figshare.19783498.v5](http://www.biosino.org/; OEP000155 node; GEO: GSE118527; SRA: SRP157974; figshare: https://doi.org/10.6084/m9.figshare.19783498.v5)
- 作用:这些数据集包含了360名三阴性乳腺癌患者的基因表达数据和88名正常乳腺组织的数据。这些数据被用于生物信息学分析,以识别与焦亡相关的基因,并构建生存模型来预测三阴性乳腺癌患者的无复发生存期(RFS)。这些数据对于理解三阴性乳腺癌的分子特征和发现潜在的治疗靶点至关重要。
-
其他数据集:
- 蛋白质-蛋白质相互作用(PPI)网络数据:https://cn.string-db.org/
- 作用:STRING数据库提供了蛋白质-蛋白质相互作用的数据,这些数据被用来构建和分析药物候选者与焦亡基因之间的网络关系,以帮助筛选和优化药物组合。
-
质谱蛋白质组学数据:
- ProteomeXchange: PXD053939
- 作用:这些数据来自使用TMT标记的质谱蛋白质组学分析,用于研究MG@PM纳米晶体处理的4T1细胞的蛋白质表达变化。这些数据有助于揭示MG@PM诱导肿瘤细胞焦亡的分子机制。
二、AI辅助乳腺癌筛查:优化筛查路径的模拟研究

一作&通讯
| 作者角色 | 作者姓名 | 单位名称 | 单位英文名称 |
|---|---|---|---|
| 第一作者 | Helen M. L. Frazer | 圣文森特医院乳腺癌筛查中心 墨尔本圣文森特医院 | St Vincent’s BreastScreen St Vincent’s Hospital Melbourne |
| 通讯作者 | Davis J. McCarthy | 圣文森特研究所医学研究所 墨尔本大学 | St Vincent’s Institute of Medical Research, University of Melbourne |
| 通讯作者 | Helen M. L. Frazer | 墨尔本大学医学、牙科与健康科学学院 | Faculty of Medicine, Dentistry & Health Sciences, University of Melbourne |
文献概述
这篇文章通过模拟研究探讨了人工智能集成在乳腺癌筛查中的应用,发现AI能够显著提高筛查的敏感性和特异性,同时减少不必要的评估和人工阅读量。
研究团队通过使用来自澳大利亚维多利亚州的大量高质量回顾性乳房X光检查数据集,模拟了五种潜在的AI集成筛查路径,并研究了人-AI交互效应,以探索自动化偏差。
研究发现,将AI作为第二阅读者或高置信度过滤器,可以提高当前筛查结果的敏感性1.9-2.5%,特异性高达0.6%,减少4.6-10.9%的评估和48-80.7%的人工阅读。自动化偏差在多阅读者设置中会降低性能,但会提高单个阅读者的性能。这项研究为AI集成筛查路径提供了可行的方法,并指出在临床采用之前进行前瞻性研究的必要性。
研究还指出,乳腺癌是全球女性最常见的癌症,也是导致癌症死亡的主要原因。尽管存在独立双阅读过程,但仍有相当比例的参与者被错误召回或漏诊。AI辅助阅读乳房X光片有潜力通过提高准确性、服务体验和效率来改变乳腺癌筛查。研究比较了AI阅读器与人类放射科医师的表现,并探讨了AI集成到筛查路径中的不同方式。
文章详细描述了AI集成筛查路径的设计、模拟研究、以及与现有筛查标准的比较。研究使用了名为BRAIx AI Reader的AI模型,该模型基于开源的深度神经网络集成。研究结果表明,AI阅读器在单个阅读者设置中表现优于平均水平的人类阅读者,并且在多阅读者设置中,通过与人类阅读者的协作,可以进一步提高筛查的准确性和效率。
最后,文章讨论了AI集成筛查路径的潜在优势和挑战,并提出了未来研究和实施的方向。研究团队强调了在实际临床环境中采用AI之前,需要进一步的前瞻性研究和算法质量保证措施。
代码&数据
-
代码链接:
- 代码库的DOI链接:10.5281/zenodo.12633016,这是由Chun Fung Kwok 和 Michael S. Elliott 提供的,与BRAIx项目相关的代码库。在文章中,这个代码库用于模拟AI读者在乳腺癌筛查中的操作,并验证外部结果。
-
数据集:
- ADMANI数据集:这是文章中使用的主要数据集,由BreastScreen Victoria提供。它包含了用于研究的乳房X光检查图像和非图像数据。这个数据集在文章中用于进行AI集成筛查路径的详细模拟研究。
- 中国乳腺X线影像数据集(CMMD):这是一个公开可用的数据集,可以从 Cancer Imaging Archive 获取。文章中提到,这个数据集用于验证AI读者的性能。
- Screen-age Women - Case-control(CSAW-CC)数据集:这个数据集可以通过 SND database 请求获取。它在文章中用于进一步的验证和比较研究。
- BreastScreen Reader Assessment Strategy Australia(BREAST Australia):这个数据集可以通过 BreastScreen Australia 请求获取。它同样用于AI读者性能的评估和验证。
三、基于ctDNA的机器学习模型在癌症相关静脉血栓栓塞预测中的应用

一作&通讯
| 作者角色 | 作者姓名 | 单位名称 | 单位英文名称 |
|---|---|---|---|
| 第一作者 | Justin Jee | 纪念斯隆凯特琳癌症中心 | Memorial Sloan Kettering Cancer Center |
| 通讯作者 | Simon Mantha | 纪念斯隆凯特琳癌症中心,威尔康奈尔医学院 | Memorial Sloan Kettering Cancer Center, Weill Cornell Medicine |
文献概述
这篇文章通过分析癌症患者的循环肿瘤DNA(ctDNA)数据,发现ctDNA的存在与静脉血栓栓塞(VTE)风险增加独立相关,并开发了一种机器学习模型,该模型在预测VTE方面优于传统风险评分。
研究的主要目的是探讨循环肿瘤DNA(ctDNA)检测与癌症患者发生静脉血栓栓塞(VTE)风险之间的关系,并开发一种基于液体活检数据的机器学习模型来预测VTE。
研究者分析了三个血浆测序队列,包括4141名非小细胞肺癌(NSCLC)或其他癌症患者的泛癌发现队列、1426名同样癌症类型的前瞻性验证队列,以及463名晚期NSCLC患者的国际泛化队列。研究发现,ctDNA的检测与VTE风险独立相关,与临床和影像学特征无关。基于液体活检数据训练的机器学习模型在预测VTE方面的表现优于之前的风险评分,如Khorana评分。
在现实世界的数据中,如果检测到ctDNA,使用抗凝治疗与较低的VTE率相关。然而,未检测到ctDNA的患者(ctDNA阴性)并未从抗凝治疗中获益。这些结果为液体活检可能改善VTE风险分层提供了初步证据,除了临床参数外,还需要进行干预性、随机的前瞻性研究来确认液体活检在指导癌症患者抗凝治疗中的临床效用。
文章还讨论了VTE在癌症患者中的医疗成本、发病率和死亡率,以及预防性抗凝治疗的重要性。目前,基于Khorana评分的预防措施存在局限性,例如许多高风险患者并未发展为VTE,而许多发展为VTE的患者Khorana评分并不高。研究还提到了ctDNA检测的潜在机制,包括与中性粒细胞胞外陷阱(NETs)的关联,以及ctDNA与更积极的肿瘤生理状态之间的联系。
最后,文章指出了研究的局限性,包括ctDNA液体活检在临床上的普遍应用程度有限,以及需要进一步研究来确定ctDNA在不同癌症类型中与VTE风险的关联。作者呼吁进行随机研究,以评估基于液体活检的预防措施或降级治疗在临床上的应用。
代码&数据:
-
代码链接:
- CEDARS: 用于自然语言处理(NLP)的代码库,帮助识别和注释患者记录中的血栓栓塞事件。
- PINES: 与CEDARS配合使用的NLP工具,用于提高识别癌症相关血栓事件的敏感性。
-
数据集:
- cBioPortal for the Discovery and Validation cohorts: 提供了发现队列和验证队列的基因组数据,用于研究ctDNA与VTE风险之间的关系。
- cBioPortal for the Generalizability cohort: 提供了泛化队列的基因组数据,用于验证研究结果的普适性(研究ID: nsclc_ctdx_msk_2022)。
- Clinical data for all cohorts: 提供了所有队列的临床数据,用于支持研究的统计分析和模型训练。
四、利用代谢组学分析预测乳腺癌治疗后长期毒性

一作&通讯
| 角色 | 姓名 | 单位名称 |
|---|---|---|
| 第一作者 | Max Piffoux | 里昂Leon Berard中心,法国 |
| 通讯作者 | Olivier Tredan | 里昂Leon Berard中心,法国 |
文献概述
这篇文章通过非靶向高分辨率代谢组学分析,研究了预测乳腺癌治疗引起的神经和代谢毒性的可能性,并发现代谢组学资料能提高对这些毒性的预测准确性。
研究设计:
- 研究使用了来自法国前瞻性CANTO队列的992名雌激素受体(ER)阳性/HER2阴性乳腺癌患者的非靶向高分辨率代谢组学资料。
- 研究采用了基于残差的建模策略,包括发现和验证队列,以评估机器学习算法的性能,并考虑了混杂变量。
主要发现:
- 自适应最小绝对收缩和选择算子(adaptive LASSO)显示出良好的预测性能,有限的乐观偏差,并允许为未来的转化研究选择感兴趣的代谢物。
- 低频代谢物和未注释代谢物的添加增加了预测能力。
- 代谢组学在预测各种神经和代谢毒性方面提供了额外的性能。
结论:
- 非靶向高分辨率代谢组学通过考虑环境暴露、与微生物群相关的代谢物和低频代谢物,可以更好地预测毒性。
研究背景:
- 乳腺癌幸存者中有相当一部分会经历长期治疗相关的毒性,这会对心理、功能和社会产生重要影响。
- CANTO研究是一项大型前瞻性法国国家临床研究,自2012年以来已招募了超过11,400名患者,整合了医疗数据、患者报告的结果和生物数据,以预测长期乳腺癌治疗相关的毒性。
实验方法:
- 研究分析了992名患者的基线血清代谢组,这些患者接受了手术治疗、放化疗和激素治疗(ER阳性,HER2阴性乳腺癌),这是一个被认为有高毒性风险的人群。
- 研究使用了多种机器学习算法,包括惩罚回归方法,如最小绝对收缩和选择算子(LASSO)和自适应LASSO,以识别对毒性预测和未来临床转化感兴趣的代谢物。
材料和方法:
- 描述了血清处理、代谢组资料获取、随机化、样本准备、数据获取、质量控制、提取和预处理等步骤。
- 详细描述了代谢物注释、代谢组数据集、临床数据获取、统计分析等方法。
临床相关性:
- 预测乳腺癌治疗后的长期神经和代谢毒性是困难的。使用机器学习算法分析诊断时的高分辨率血清代谢组资料,通过反映患者的代谢、环境暴露和微生物群,为临床检查增加了额外的预测性能。
五、智能病理诊断:钙钛矿纳米晶体与机器学习在癌症检测中的应用

一作&通讯
| 作者角色 | 姓名 | 单位名称(中文) |
|---|---|---|
| 第一作者 | Jimei Chi | 中国科学院分子科学卓越研究中心/绿色印刷北京市重点实验室 |
| 通讯作者 | Meng Su | 中国科学院分子科学卓越研究中心/绿色印刷北京市重点实验室 |
| 通讯作者 | Yanlin Song | 中国科学院分子科学卓越研究中心/绿色印刷北京市重点实验室 |
文献概述
这篇文章介绍了一种基于钙钛矿纳米晶体探针和机器学习算法的新型癌症病理诊断方法,能够快速准确地区分肿瘤和正常组织,提高癌症诊断的效率和准确性。
- 研究背景:传统的荧光免疫组化技术在肿瘤细胞和正常细胞的鉴别上存在局限性,如肿瘤细胞的异质性和缺乏大数据分析。
- 研究目的:开发一种机器学习驱动的成像方法,使用钙钛矿纳米晶体探针,快速病理诊断五种类型的癌症(乳腺癌、结肠癌、肝癌、肺癌和胃癌)。
- 方法:
- 生物分析了五种不同癌症中survivin蛋白的表达。
- 制备了修饰有survivin抗体的高效率钙钛矿纳米晶体探针,能够在单细胞水平上识别癌组织。
- 利用机器学习从1000张荧光图像中提取和分析荧光强度和病理纹理形态特征。
- 结果:
- 与传统荧光探针相比,钙钛矿纳米晶体探针的肿瘤与正常组织(T/N)比率提高了10.3倍,能在10分钟内成功区分肿瘤和相邻正常组织。
- 机器学习分类的乳腺癌、结肠癌、肝癌、肺癌和胃癌的接收者操作特征曲线下面积(AUC)值超过90%,正确预测了92%的阳性患者的肿瘤器官。
- 结论:该方法展示了高T/N比率探针在精确诊断多种癌症中的优势,有助于提高手术切除的准确性和降低癌症死亡率。
文章还详细描述了钙钛矿纳米晶体探针的制备和表征、survivin基因在癌症中的表达分析、病理切片的染色和成像,以及机器学习模型的建立和验证。研究结果表明,该方法在提高病理诊断的效率和准确性方面具有重要的临床应用潜力。
重点关注
Figure 3 提供了关于survivin蛋白在五种癌症(乳腺癌、结肠癌、肝癌、肺癌和胃癌)中的表达情况以及使用钙钛矿纳米晶体探针和FITC标记的比较分析。
(a) survivin在正常和配对癌症组织中的表达比较
- 目的:展示survivin蛋白在正常组织和相应癌症组织中的表达差异。
- 方法:通过免疫荧光技术,使用针对survivin的抗体对组织切片进行染色,并通过荧光显微镜观察和比较荧光强度。
- 意义:高表达的survivin通常与肿瘤的发生和发展相关,这有助于识别和区分癌症组织。
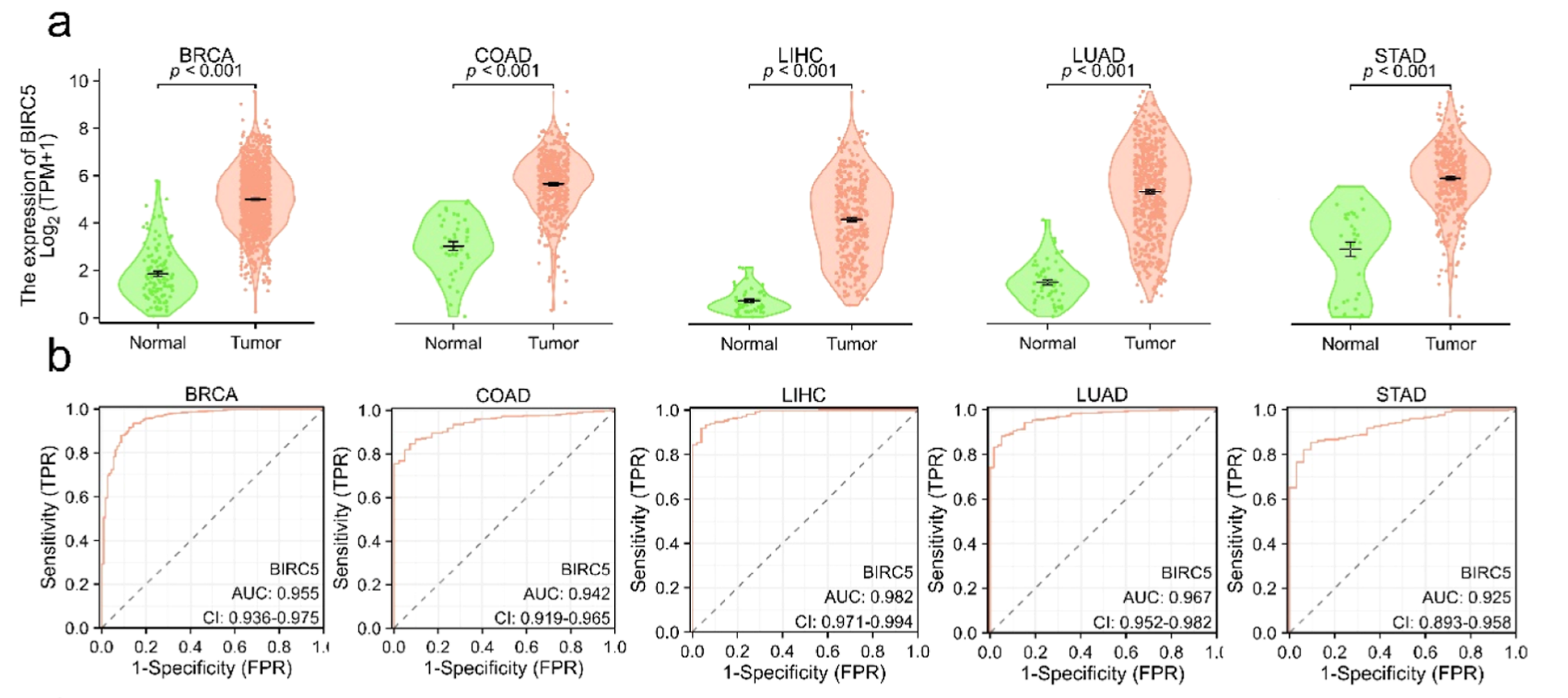
(b) ROC曲线和相应的AUC值
- ROC曲线:接收者操作特征曲线(Receiver Operating Characteristic curve),用于展示分类模型在所有可能的阈值下真正例率(敏感性)和假正例率(1-特异性)。
- AUC值:ROC曲线下面积(Area Under the Curve),用于评估模型的诊断性能。AUC值越接近1,表示模型的分类性能越好。
- 意义:通过计算AUC值,可以量化不同癌症类型中survivin表达的诊断价值。
© 免疫荧光成像
- 目的:通过比较FITC标记的BIRC5和DSPE-PNCs@BIRC5在癌症和相邻正常组织中的染色效果,展示钙钛矿纳米晶体探针的特异性和灵敏度。
- 方法:使用FITC标记的BIRC5和DSPE-PNCs@BIRC5对组织切片进行染色,然后通过荧光显微镜观察。
- 结果:与FITC标记相比,DSPE-PNCs@BIRC5显示出更强的荧光信号,表明其具有更高的肿瘤组织与正常组织(T/N)比率。
- H&E染色:作为对照的常规组织染色方法,用于观察组织形态。
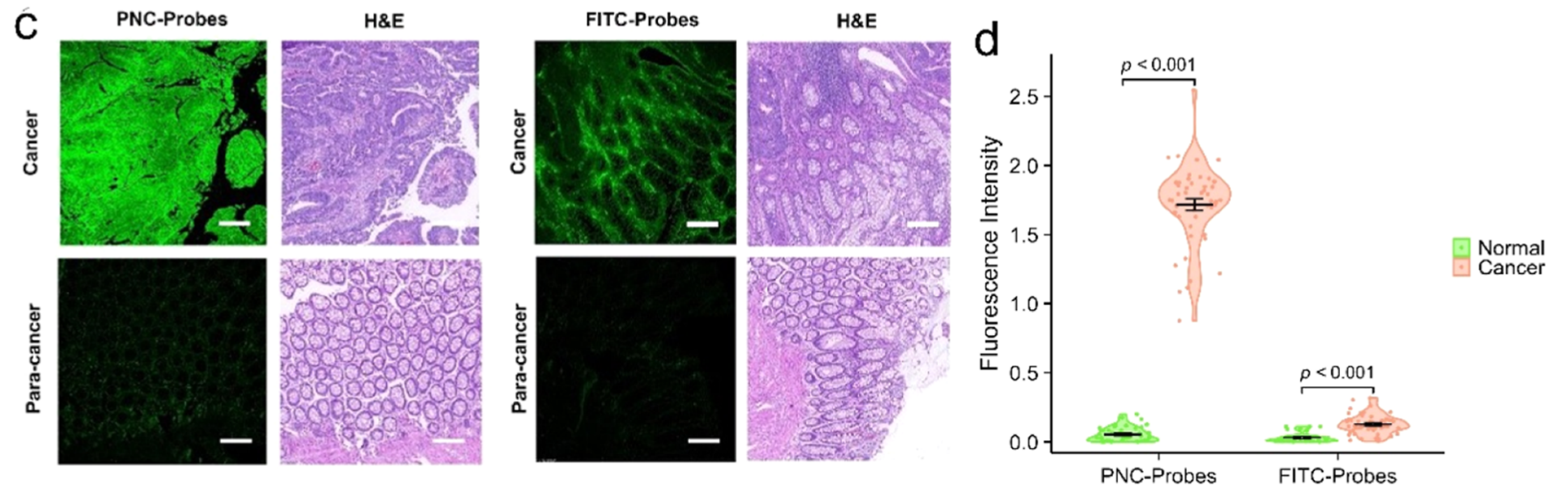
(d) 荧光强度分析统计
- 目的:定量分析FITC标记的BIRC5和DSPE-PNCs@BIRC5在免疫荧光染色中的荧光强度。
- 意义:通过统计分析,可以进一步验证DSPE-PNCs@BIRC5探针在癌症诊断中的有效性和优越性。
六、STdGCN:利用图卷积网络进行空间转录组数据的细胞类型分解

一作&通讯
| 作者角色 | 姓名 | 单位名称(中文) |
|---|---|---|
| 第一作者 | 李 | 西北大学预防医学系 |
| 通讯作者 | 罗 | 西北大学预防医学系,协作AI医疗中心 |
文献概述
这篇文章介绍了一种名为STdGCN(Spatial Transcriptomics deconvolution using Graph Convolutional Networks)的新型图模型,它利用单细胞RNA测序(scRNA-seq)数据作为参考,来对空间转录组(ST)数据中的细胞类型进行分解。
空间转录组技术能够在保持组织空间背景的情况下测量转录组,为疾病病理学研究提供了宝贵的见解。然而,这些技术由于细胞分辨率和通量之间的权衡而面临限制。
STdGCN通过整合scRNA-seq数据中的表达谱和ST数据中的空间定位信息来实现细胞类型分解。通过这种方法,STdGCN在多个数据集上的广泛基准测试中展示了其优越的性能,超越了17个现有的最先进模型。在人类乳腺癌Visium数据集中,STdGCN能够描绘出基质细胞、淋巴细胞和癌细胞的分布,有助于肿瘤微环境分析。在人类心脏ST数据中,STdGCN识别了组织发育过程中内皮-心肌细胞通讯的变化。
文章还详细描述了STdGCN的工作原理,包括如何识别细胞类型标记基因、生成伪斑点、构建表达图和空间图以及如何通过图卷积网络(GCN)进行信息传播。此外,文章还提供了STdGCN在不同数据集上的基准测试结果,展示了其在细胞类型分解方面的准确性和鲁棒性。
最后,文章讨论了STdGCN的潜在应用,包括在复杂组织环境中探索细胞组成和空间组织之间的复杂相互作用,以及如何通过STdGCN提高我们对空间分辨转录组学及其在各种生物过程和疾病状态中含义的理解。
代码&数据
-
STdGCN GitHub代码仓库
- 链接:https://github.com/luoyuanlab/stdgcn
- 作用:提供了STdGCN模型的实现代码,允许其他研究者下载、使用和参考该模型来进行空间转录组数据的细胞类型分解。
-
STdGCN Zenodo代码存档
- 链接:https://doi.org/10.5281/zenodo.12775443
- 作用:作为代码的存档,提供了STdGCN模型的另一个下载源,确保了研究的可持续性。
-
seqFISH数据集
- 链接:https://content.cruk.cam.ac.uk/jmlab/SpatialMouseAtlas2020/
- 作用:提供了小鼠胚胎的空间转录组数据,用于评估STdGCN模型的性能。
-
seqFISH+数据集
- 链接:通过Giotto包获取:
getSpatialDataset(dataset = “seqfish_SS_cortex”, method = “wget”) - 作用:提供了小鼠体感皮层的空间转录组数据,用于模型的基准测试和性能评估。
- 链接:通过Giotto包获取:
-
MERFISH数据集
- 链接:https://datadryad.org/stash/dataset/doi:10.5061/dryad.8t8s2448
- 作用:提供了小鼠下丘脑前区的空间转录组数据,用于模型的基准测试和性能评估。
-
10X Genomics Visium人类乳腺癌数据集
- 链接1:https://doi.org/10.5281/zenodo.4739739
- 链接2:https://doi.org/10.5281/zenodo.3957257
- 作用:提供了人类乳腺癌的空间转录组数据,用于展示STdGCN在实际生物医学数据上的应用和效果。
-
人类心脏空间转录组数据
- 链接:https://www.spatialresearch.org/resources-published-datasets/doi-10.1016j-cell-2019-11-025/
- 作用:提供了人类心脏发育过程中的空间转录组数据,用于验证STdGCN在复杂组织环境中分解细胞类型的能力。
重点关注
图2展示了四个代表性的多细胞合成基准空间转录组(ST)切片,这些切片分别对应四种不同的ST平台:seqFISH、MERFISH、seqFISH+和slide-seq。
这些数据集被用来评估STdGCN模型的性能。
- seqFISH:用于小鼠胚胎的细胞图谱分析。
- MERFISH:用于小鼠前视区的细胞图谱分析。
- seqFISH+:用于小鼠体感(SS)区域的细胞图谱分析。
- slide-seq:用于小鼠睾丸区域的细胞图谱分析。
在这些研究中,每个切片被划分为多个正方形像素区域(虚线表示)。每个正方形像素区域内的细胞随后被合并成合成斑点。合成斑点的细胞类型比例通过饼图(B, D, F, 和 H)展示。
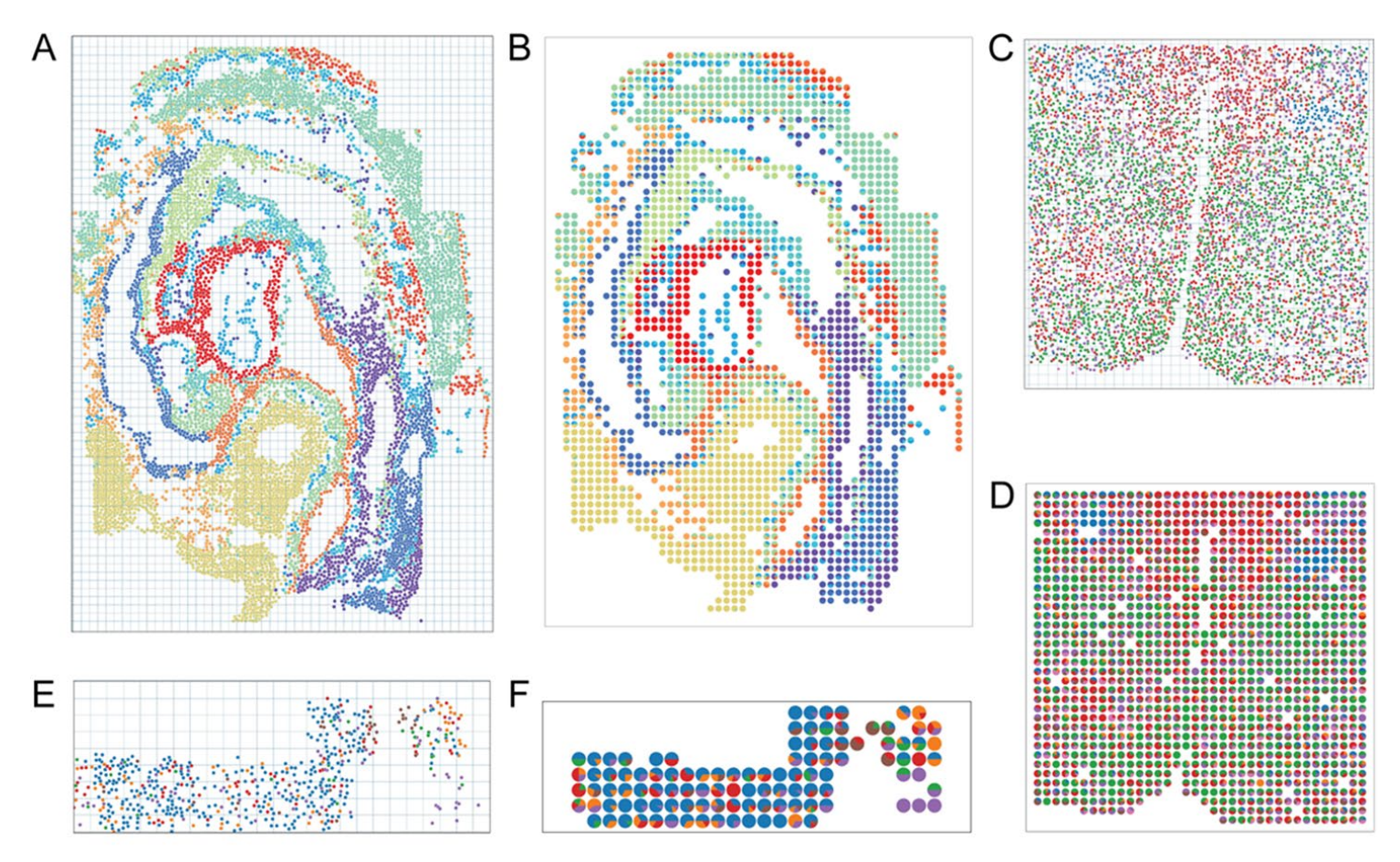

这个过程的目的是为了模拟真实的空间转录组数据,其中每个“合成斑点”代表了在实际ST数据中可能观察到的一个特定区域内的细胞群体。通过这种方法,研究者能够评估STdGCN模型在从合成斑点的基因表达数据中准确推断细胞类型比例的能力。
在文章中,这些图表和描述用于展示STdGCN模型在不同ST平台数据上的应用效果,以及模型在处理来自不同来源和具有不同特性的空间转录组数据时的准确性和鲁棒性。这些基准测试结果对于验证STdGCN模型的有效性至关重要。