微生态研究搞了n多年,益生菌还是那么几个,为什么?
一个共识是单一菌株的添加往往难以解决复杂问题。微生物群体的相互作用和平衡不应被忽视。实际上,我们需要在群体层面进行添加或干预,才能真正发挥益生菌的作用。然而,目前我们缺乏一种精确的检测工具来区分本底微生物与添加的益生菌,以及评估它们的竞争力和活性。
WISH-tags(WISH-标签技术)正是在这样的背景下应运而生,为我们提供了一种全新的尝试。随着这一技术的不断成熟,我们可以预见,未来将有更多创新的研究涌现。

研究背景
尽管通过扩增子序列变异的分类提高了细菌群落的分辨率,但基于16S rRNA的分析仍无法捕获微生物群落内部的真实遗传变异。具有相同标记基因序列的菌株可能构成一个细菌种群,其中多态性位点与有益或有害的性状相关。
主要结果
01、WISH标签的设计和生成
开发的WISH标签仅在唯一条形码区域的40bp上有所不同,这确保了任何两个标签之间的足够独特性。其中包含用于qPCR的独特反向引物的结合位点,它与通用正向引物(也用于NGS)一起,可以通过qPCR定量鉴定给定的WISH标签。所得扩增产物长88bp,在qPCR产物的最佳长度范围,而NGS扩增子的引物位点除了突出的序列长度外,还间隔以产生120bp的片段。独特条形码双向测序,有助于减轻测序错误的影响。
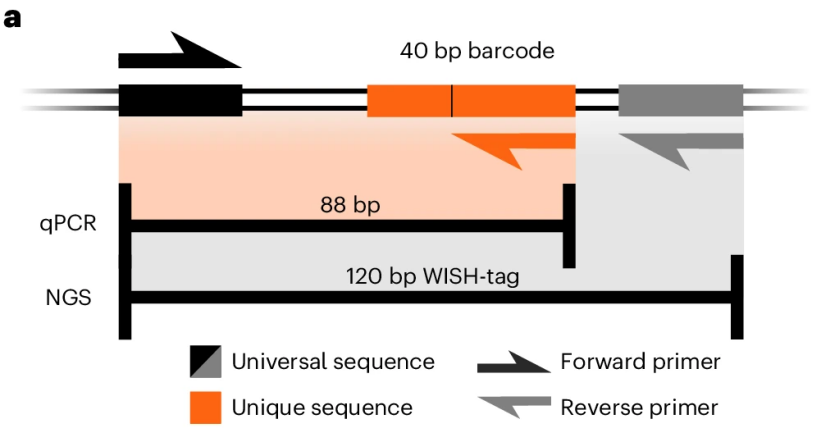
图1:WISH标签构建的设计和原理
主要结果
02、利用qPCR和NGS验证WISH-tag的扩增
为了验证WISH标签的性能,使用qPCR和NGS对其进行了评估。独特的引物不会与不匹配的条形码序列结合,证明了WISH标签对qPCR的高特异性。通过NGS评估了所有62个WISH标签,并将它们组合成8个浓度相等的WISH标签的混合物。除两个异常值外,背景噪声和信号之间的分离均为五个数量级,标签在计数数量方面的检测一致,表明它们的表现同样出色。标签也可以在大范围稀释中可靠地用于量化标签。
图2:WISH标签扩增的验证
WISH标签被引入到小鼠和植物微生物群落的模型和非模型细菌成员中。研究发现,小鼠肠道对迟到的沙门氏菌表现出定殖抵抗,而叶片对迟到的鞘氨醇单胞菌的容纳程度则与它们在晚接种时间点的存在量成正比。这表明WISH标签是解密横跨生物系统的微生物群落组装背后的种群动态的有力工具。
结论与意义
研究开发了WISH标签,允许使用qPCR和NGS对其进行定量。插入模型和非模型细菌后,WISH标签可以应用于特定的微生物组或多个微生物组。实验性地验证了62个标签的性能,并显示它们可以以高精度进行区分。
总之,WISH-tags是一种新型的基因组条形码系统,可用于高分辨率追踪微生物群落中单株水平的种群动态,有助于阐明微生物组装的潜在机制。在未来的研究中使用WISH标签可能会为揭示具有亚菌株水平分辨率的组成规则和推进潜在机制的研究提供有价值的见解。期待与您合作,共同探索微生态研究的新篇章!
参考文献
Daniel, B.B.J., Steiger, Y., Sintsova, A. et al. Assessing microbiome population dynamics using wild-type isogenic standardized hybrid (WISH)-tags. Nat Microbiol 9, 1103–1116 (2024).