说明
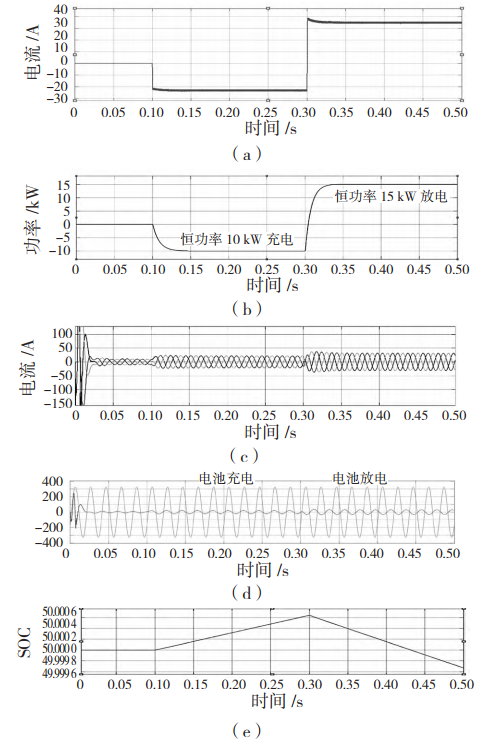
首先假设数据由多个正态分布叠加而成,这个场景应该也是比较有普遍意义的。
内容
数据还是之前产生的三波
import numpy as np
import matplotlib.pyplot as plt
from sklearn.mixture import GaussianMixture
# 生成示例数据
np.random.seed(0)
data1 = np.random.normal(loc=20, scale=5, size=300)
data2 = np.random.normal(loc=50, scale=10, size=700)
data3 = np.random.normal(loc=70, scale=30, size=500)
data = np.concatenate([data1, data2,data3])
进行拟合并画图
# 拟合 GMM
gmm = GaussianMixture(n_components=3, random_state=0)
gmm.fit(data.reshape(-1, 1))
# 生成拟合的 GMM 密度曲线
xmin, xmax = plt.xlim()
x = np.linspace(data.min(), data.max(), 1000)
logprob = gmm.score_samples(x.reshape(-1, 1))
pdf = np.exp(logprob)
# 绘制直方图和拟合的 GMM 密度曲线
plt.hist(data, bins=30, density=True, alpha=0.6, color='g', label='Data')
plt.plot(x, pdf, 'k', linewidth=2, label='GMM')
# 添加图例和标签
plt.title('Gaussian Mixture Model Fit')
plt.xlabel('Value')
plt.ylabel('Density')
plt.legend()
# 显示图形
plt.show()

从图像上看,是不太对的,看起来只有两个波。但是看模型拟合的参数是对的
gmm.means_
array([[49.35769361],
[75.36264743],
[20.41493429]])
再看标准差
import numpy as np
from sklearn.mixture import GaussianMixture
# 获取每个成分的标准差
std_devs = np.sqrt(gmm.covariances_).flatten()
print(f"Standard Deviations of each component: {std_devs}")
Standard Deviations of each component: [ 9.57180175 27.62374819 5.43236064]
已经非常接近真相了。
很有意思,把几种豆子混在一起撒进去,然后还能分出来。
如果假设的n_components为7,乍一看不出问题,之后再用其他来进行检验。之前我还认为多给的类会几种在一起,现在看来不是这样。
gmm.means_
array([[ 45.51781429],
[ 76.17244197],
[ 30.74652342],
[ 94.62139471],
[ 56.99546783],
[ 19.9330969 ],
[117.56899306]])
方差
Standard Deviations of each component: [ 5.20720528 7.61508437 7.3278159 8.05410436 5.92218708 7.04771942
15.85456659]