小罗碎碎念
今天这期推文是6月份前半个月的文献总结,从90篇文章中挑了12个与病理AI相关的开源项目。
这一期推文先介绍6个项目,明天再介绍剩下的6个,信息量比较大,建议反复阅读。
一个项目的完成,无非就三个要素——人+数据+模型,所以我这期推文还分析了每一个项目采用的数据集。

后续还会不定期分享公开数据集,以及使用方式,敬请关注!!
今日其余推文
- 从国自然立项情况浅谈一下医学AI的未来发展趋势|个人观点·24-08-28
- Histolab:切割病理切片并添加网格输出的工具|项目分享·24-08-28
一、弱监督深度学习在全切片图像上的应用

一作&通讯
| 作者类型 | 姓名 | 单位名称(中文) |
|---|---|---|
| 第一作者 | Gabriele Campanella | 纪念斯隆凯特琳癌症中心病理科,纽约,美国 |
| 通讯作者 | Thomas J. Fuchs | 纪念斯隆凯特琳癌症中心病理科,纽约,美国;威尔康奈尔医学院研究生院医学科学系,纽约,美国 |
文献概述
这篇文章提出了一种使用弱监督深度学习在大规模切片图像上进行临床级计算病理学分析的新方法。
这个系统由Gabriele Campanella和Thomas J. Fuchs领导的研究团队开发,旨在通过减少对大规模手动注释数据集的需求,来提高病理学的临床决策支持系统的发展和部署。
研究团队面临的主要挑战是,传统的病理图像分析依赖于专家知识进行特征提取,这些方法在临床应用中的性能并不总是足够。而深度学习在图像分类任务上取得了巨大成功,特别是在医学图像分析领域,有时甚至能够达到临床影响的程度。然而,病理数据的生成面临着缺乏大型注释数据集的挑战,这在一定程度上是由于数字病理学的新近性和切片数字化的高成本。
为了解决这些问题,研究团队收集了大规模的计算病理学数据集,并提出了一种新的框架,无需像素级注释即可在大规模上训练分类模型。他们使用了多实例学习(Multiple Instance Learning, MIL)的方法,仅使用报告的诊断作为训练标签,避免了昂贵和耗时的逐像素手动注释。
研究结果表明,该系统在前列腺癌、基底细胞癌和乳腺癌转移至腋窝淋巴结的测试中,所有癌症类型的曲线下面积(AUC)都超过了0.98。这意味着,该系统能够在保持100%灵敏度的同时,排除65-75%的切片,从而提高病理学家的工作效率。
此外,研究还探讨了模型在不同放大倍数下的性能,以及模型对数据集大小的依赖性。通过可视化技术,研究者们还展示了模型学习到的特征空间,以及不同切片聚合方法的比较。
最后,文章讨论了将这种深度学习模型应用于临床实践的可能性和挑战,包括模型对不同扫描仪技术变化的泛化能力,以及与完全监督学习相比,弱监督学习在处理大规模、多样化数据集方面的优势。研究团队认为,他们的工作为计算决策支持系统在临床实践中的应用奠定了基础。
代码仓库
MIL-nature-medicine-2019
https://github.com/MSKCC-Computational-Pathology/MIL-nature-medicine-2019
数据集
-
前列腺核心活检数据集 (Prostate core biopsy dataset):
- 描述:包含24,859张切片,涵盖836名患者的数据。
- 作用:用于训练和评估深度学习模型,以识别前列腺癌。
-
皮肤数据集 (Skin dataset):
- 描述:包含9,962张切片,涵盖5,325名患者的数据,包括多种皮肤病变,如基底细胞癌(BCC)。
- 作用:用于训练和评估模型对不同皮肤病变的分类能力,尤其是识别基底细胞癌。
-
乳腺癌转移至淋巴结数据集 (Breast metastasis to lymph nodes dataset):
- 描述:包含9,894张切片,涵盖2,703名患者的数据,其中包括乳腺癌的宏观转移、微观转移或孤立肿瘤细胞(ITCs)。
- 作用:用于评估模型在检测乳腺癌转移至腋窝淋巴结的能力。
-
CAMELYON16挑战数据集 (CAMELYON16 challenge dataset):
- 描述:这是一个公共的数字病理数据集,包含400名患者的切片图像,以及详尽的像素级注释。
- 作用:用于与研究中提出的弱监督学习方法进行比较,评估模型在乳腺癌转移检测任务上的性能。
-
MSK乳腺癌转移数据集 (MSK breast cancer metastases dataset):
- 描述:这是由研究团队公开分享的数据集,包含130张去标识化的切片图像,涵盖78名患者的腋窝淋巴结标本。
- 作用:提供了一个公共的测试基准,允许其他研究人员评估他们的模型或方法在相同的数据集上的表现。
二、CLAM:高效弱监督学习在全切片图像病理诊断中的应用

一作&通讯
| 角色 | 姓名 | 单位名称(中文) |
|---|---|---|
| 第一作者 | Ming Y. Lu | 布莱根妇女医院病理科,哈佛医学院,波士顿,美国 |
| 第一作者(共同) | Drew F. K. Williamson | 布莱根妇女医院病理科,哈佛医学院,波士顿,美国 |
| 第一作者(共同) | Tiffany Y. Chen | 布莱根妇女医院病理科,哈佛医学院,波士顿,美国 |
| 通讯作者 | Faisal Mahmood | 布莱根妇女医院病理科,哈佛医学院,波士顿,美国 |
文献概述
这篇文章提出了一种新的深度学习框架CLAM,用于高效、弱监督的全切片图像分析,能够在无需手动标注的情况下,准确分类和识别多种癌症亚型。
CLAM方法旨在解决传统深度学习方法在处理全切片图像时面临的一些挑战,如需要大量的手动标注或在弱监督设置下需要大量带标注的数据集。
-
背景介绍:介绍了数字病理学和人工智能在客观诊断、预后预测和治疗反应预测方面的潜力,以及深度学习在医学成像中的应用。
-
CLAM方法:提出了一种新的深度学习框架,使用基于注意力机制的学习来识别具有高诊断价值的子区域,并使用实例级聚类来约束和细化特征空间。
-
数据效率:展示了CLAM方法在数据效率方面的优势,即使在使用较少的训练标签的情况下也能实现高性能。
-
多任务适用性:证明了CLAM方法不仅适用于二分类问题,还能推广到多类别分类和亚型问题。
-
泛化能力:展示了CLAM模型在独立测试队列中的泛化能力,包括智能手机显微镜图像和活检切片。
-
解释性:CLAM能够生成解释性热图,帮助可视化和理解模型用于预测的每个切片的相对贡献和重要性。
-
开源工具:提供了一个易于使用的Python包和交互式演示,以便研究人员和临床医生可以利用CLAM进行研究和诊断。
-
实验结果:通过在肾细胞癌、非小细胞肺癌亚型以及淋巴结转移检测等多个数据集上的实验,证明了CLAM方法的有效性。
-
讨论:讨论了CLAM在计算病理学中的应用前景,以及未来可能的研究方向,如扩展到其他问题、开发更具上下文感知力的方法等。
-
方法和数据集:详细描述了CLAM方法的实现细节,以及使用的数据集和它们的来源。
文章强调了CLAM作为一种弱监督学习方法,在临床和研究环境中对计算病理学的广泛适应性,以及它在提高数据处理效率和模型解释性方面的潜力。
代码仓库
https://github.com/mahmoodlab/CLAM
数据集
- 公共数据集:
- TCGA RCC数据集:包含肾细胞癌(RCC)的全切片图像,用于模型开发和评估。
- TCGA NSCLC数据集:包含非小细胞肺癌(NSCLC)的全切片图像,用于肺癌亚型分类。
- CPTAC NSCLC数据集:同样包含NSCLC的全切片图像,与TCGA数据集结合使用。
- Camelyon16和Camelyon17数据集:包含乳腺癌淋巴结转移检测的全切片图像,用于模型的跨验证和独立测试。
- 独立测试队列(BWH内部数据集):
- BWH RCC WSI数据集:来自布莱根妇女医院(BWH)的肾细胞癌全切片图像,用于评估模型的泛化能力。
- BWH NSCLC WSI数据集:来自BWH的非小细胞肺癌全切片图像,包括切除和活检样本,用于测试模型性能。
- BWH淋巴结转移(乳腺癌)WSI数据集:乳腺癌淋巴结转移的全切片图像,用于独立测试模型。
三、CellViT:利用视觉变换器实现细胞核精确分割与分类的深度学习模型

一作&通讯
| 角色 | 姓名 | 单位名称(中文) |
|---|---|---|
| 第一作者 | Fabian Hörst | 埃森大学医院人工智能医学研究所(IKIM) |
| 通讯作者 | Jan Egger | 埃森大学医院人工智能医学研究所(IKIM) |
文献概述
这篇文章是关于一种名为CellViT的新型深度学习架构,它基于视觉变换器(Vision Transformers),用于精确的细胞分割和分类。
这项研究由德国埃森大学医院(University Hospital Essen)的人工智能医学研究所(Institute for AI in Medicine, IKIM)和其他几个研究所的研究人员共同完成。
主要贡献:
- 提出了一种用于自动化细胞核实例分割的新方法,使用基于视觉变换器的深度学习架构。
- 在PanNuke数据集上训练和评估了CellViT,这是一个具有挑战性的细胞实例分割数据集,包含19种组织类型中的近200,000个注释细胞核。
- 展示了大规模领域内和领域外预训练的视觉变换器的优越性,通过利用最近发布的Segment Anything Model和预训练在1.04亿个组织图像块上的ViT-encoder。
- 在PanNuke数据集上实现了最先进的细胞核检测和实例分割性能,平均全景质量(mean panoptic quality)达到0.50,A1-detection分数达到0.83。
- 提供了一种能够快速应用于千兆像素全切片图像(WSI)的框架,使用1024×1024像素的大推理补丁尺寸,与传统的256像素大小补丁相比,推理速度快1.85倍。
研究背景:
- 癌症是全球严重的疾病负担,病理学家通过显微镜评估组织样本来确定潜在的治疗途径。
- 数字化组织样本(全切片图像,WSI)的使用使得计算机视觉算法的应用成为可能。
技术细节:
- CellViT基于Vision Transformer,这是一种使用注意力机制来捕获局部和全局上下文信息的基于令牌的神经网络。
- 与传统的卷积神经网络(CNN)相比,ViT能够理解图像中所有细胞之间的关系,利用长距离依赖性显著提高分割性能。
- 通过预训练模型,CellViT能够处理医疗领域数据量有限的问题。
实验结果:
- CellViT在PanNuke数据集上的表现超越了现有的细胞实例分割模型。
- 通过使用大型推理补丁,CellViT能够更快地处理千兆像素的WSI。
代码和数据集:
- 文章提供了相关数据集的链接以及CellViT的代码,这些资源是公开可用的。
文章强调了CellViT在精确分割和分类细胞方面的潜力,特别是在处理大规模数据集时的效率和准确性。
代码仓库
https://github.com/TIO-IKIM/CellViT
数据集
这篇文章中提到了以下几个数据集,以及它们在文章中的作用:
-
PanNuke 数据集
- 文章中提到:“CellViT is trained and evaluated on the PanNuke dataset, which is one of the most challenging nuclei instance segmentation datasets, consisting of nearly 200,000 annotated nuclei into 5 clinically important classes in 19 tissue types.”
- 作用:PanNuke 数据集是用于训练和评估 CellViT 模型的主要数据集。它是一个具有挑战性的细胞实例分割数据集,包含19种不同组织类型中的近200,000个被注释的细胞核,分为5个临床重要类别。这个数据集用于验证 CellViT 模型在细胞核分割和分类任务上的性能。
-
MoNuSeg 数据集
- 文章中提到:“The MoNuSeg (Kumar et al., 2020, 2017) dataset serves as an additional dataset for nuclei segmentation.”
- 作用:MoNuSeg 数据集用于额外的细胞核分割任务。与 PanNuke 数据集不同,MoNuSeg 数据集规模较小,并且没有将细胞核分为不同的类别。在本文中,MoNuSeg 数据集被用来评估模型的泛化能力,即在未见过的数据集上的表现。
-
CoNSeP 数据集
- 文章中提到:“We utilized the colorectal nuclear segmentation and phenotypes (CoNSeP) dataset by Graham et al. (2019) to analyze extracted cell embeddings (see Section 3.3) of detected cells on an external validation dataset.”
- 作用:CoNSeP 数据集被用于分析检测到的细胞的提取细胞嵌入向量(cell embeddings)。这个数据集提供了一个外部验证集,用于评估 CellViT 模型提取的细胞嵌入向量在新类别上的适应性和分类能力。
这些数据集在文章中的作用主要是支持模型的开发、训练、评估和验证。通过在不同的数据集上进行测试,研究人员能够展示 CellViT 模型的有效性、泛化能力以及在不同组织类型中的分割和分类性能。
四、深度学习助力巴雷特食管的大规模组织病理学筛查

一作&通讯
| 角色 | 姓名 | 单位名称(英文) | 单位名称(中文) |
|---|---|---|---|
| 第一作者 | Kenza Bouzid | Microsoft Health Futures | 微软健康未来 |
| 第一作者 | Harshita Sharma | Cyted Ltd | 赛泰德有限公司 |
| 通讯作者 | Marcel Gehrung | Cyted Ltd | 赛泰德有限公司 |
| 通讯作者 | Javier Alvarez-Valle | Microsoft Health Futures | 微软健康未来 |
文献概述
这篇文章讨论了一种基于弱监督深度学习的方法,用于在组织病理学中大规模筛查巴雷特食管(Barrett’s esophagus,简称BE)。
BE是食管腺癌(esophageal adenocarcinoma,简称EAC)的癌前状况,及时检测可以提高患者的生存率。目前,CytospongeTFF3测试是一种非内镜的微创程序,用于诊断BE中的肠上皮化生(intestinal metaplasia,简称IM),但这种方法依赖于病理学家对H&E染色和免疫组化生物标记TFF3的评估,这种资源密集型的临床工作流程限制了在高危人群中的大规模筛查。
为了提高筛查能力,研究人员提出了一种深度学习方法,可以直接从常规染色的H&E玻片中检测BE,这种方法仅依赖于诊断标签,消除了对昂贵的局部专家注释的需求。研究团队在两个临床试验数据集上训练并独立验证了他们的方法,总共涵盖了1866名患者。他们发现,H&E模型在发现和外部测试数据集上分别达到了91.4%和87.3%的AUROC(接收者操作特征曲线下面积),与TFF3模型相当。
研究还提出了两种半自动化的临床工作流程,可以分别将病理学家的工作量减少到48%和37%,而不影响诊断性能,使病理学家能够优先处理高风险病例。
文章强调,早期发现癌症为患者提供了最佳的长期生存和生活质量的机会。BE作为EAC的癌前组织,为早期检测和治疗EAC提供了机会。然而,目前只有不到20%的BE患者被诊断出来,导致大多数EAC病例在没有早期治疗可能性的情况下被诊断。目前,BE的标准诊断测试是内镜活检和组织病理学检查,但这种方法资源密集,限制了在大规模筛查中的使用。
研究人员使用深度学习模型来改善筛查覆盖率,这些方法在癌症检测和分类方面展示了有希望的诊断性能。他们还探讨了如何将深度学习模型集成到临床实践中,以减少病理学家的手动工作量,并提高筛查的覆盖率和成本效益。
最后,文章讨论了研究的局限性和未来的研究方向,包括提高H&E BE-TransMIL模型的性能,以及如何将深度学习模型更好地集成到临床工作流程中。
代码仓库
这篇文章中提到了以下代码链接及其在文章中的作用:
-
Microsoft Health Intelligence Machine Learning toolbox (himl)
- 作用:BE-TransMIL代码库中包含了himl作为子模块,它提供了数据预处理、网络架构以及训练和评估弱监督深度学习模型所需的代码和库要求。
-
hi-ml-cpath
- 作用:这是himl toolbox中的一个特定部分,专门用于计算病理学(computational pathology)的深度学习模型。它包含了使用himl软件的详细说明和文档。
数据集
这篇文章中提到了两个主要的数据集,它们在研究中的作用如下:
-
DELTA实施研究数据集 (DELTA implementation study)
- 作用: 这个数据集包含了1141个病例,包括H&E(苏木精-伊红)染色和TFF3(三叶因子3)染色的全切片图像。这些图像来自DELTA研究的参与者。DELTA是一个前瞻性试验,包括已知的BE患者和反流筛查患者。该数据集被用来训练和验证所提出的深度学习模型,以检测BE。
-
BEST2多中心临床试验数据集 (BEST2 multi-center clinical trials dataset)
- 作用: 这个数据集包含了725个病例的H&E染色图像,来自BEST2临床试验的参与者。BEST2试验的所有患者都在Cytosponge操作后一小时内进行了内镜检查。该数据集被用作外部验证集,以评估模型在独立队列中的泛化能力。
五、基于深度学习的双核细胞全切片图像到补丁级别的细粒度检测方法

一作&通讯
| 角色 | 姓名 | 单位名称 | 单位英文名称 |
|---|---|---|---|
| 第一作者 | Geng Hu | 北京航空航天大学医学工程学院 | Beihang University, School of Engineering Medicine |
| Baomin Wang | 同上 | 同上 | |
| Boxian Hua | 同上 | 同上 | |
| Dan Chen | 同上 | 同上 | |
| 通讯作者 | Lihua Hu | 北京大学第一医院心脏科 | Peking University First Hospital, Department of Cardiology |
| Guiping Hu | 北京航空航天大学医学工程学院大数据精准医疗北京市重点实验室 | Beihang University, School of Engineering Medicine, Key Laboratory of Big Data-Based Precision Medicine (Beihang University), Ministry of Industry and Information Technology |
文献概述
这篇文章提出了一种基于深度学习的新型多任务检测方法,通过在全切片图像上进行粗略检测和在补丁级别上进行细粒度分类,提高了双核细胞(BCs)检测的准确性和效率,有助于恶性肿瘤风险评估。
双核细胞的准确快速检测在预测白血病和其他恶性肿瘤的风险方面起着重要作用。然而,使用显微镜图像手动计数BCs既耗时又主观,而传统的图像处理方法由于染色质量和BC显微镜图像中形态特征的多样性而表现不佳。
为了克服这些挑战,文章提出了一种受BC结构先验启发的多任务方法,该方法在WSI级别进行粗略检测,并在补丁级别进行细粒度分类。粗略检测网络是一个基于圆形边界框的多任务检测框架,用于细胞检测和核中心关键点检测。圆形表示减少了自由度,与通常的矩形框相比,减轻了周围杂质的影响,并且可以在WSIs中旋转不变。检测核中的关键点可以帮助网络感知,并用于后续细粒度分类中的无监督颜色层分割。
细分类网络由基于颜色层掩模监督的背景区域抑制模块和基于变压器的全局建模能力的键区域选择模块组成。此外,文章首次提出了一个无监督和未配对的细胞质生成器网络,用于扩展长尾分布数据集。
最终,在BC多中心数据集上进行的实验表明,所提出的BC细检测方法在几乎所有评估标准上都优于其他基准,为癌症筛查等任务提供了明确和支持。
文章的主要贡献包括:
- 提出了一种基于BC圆形形状和双核结构的基于圆形表示的多任务检测模型,用于WSI级别的粗略BC检测。
- 在补丁级别提出了一个由区域抑制和选择模块组成的细粒度分类网络,用于BC的细分类。
- 提出了一个无监督和未配对的细胞质生成器网络,用于数据扩展。
文章还回顾了相关工作,介绍了具体实施方法,并展示了结果、讨论/分析和结论。
代码仓
https://github.com/geng007/FBCNet
数据集
这篇文章中提到了用于训练和评估所提出方法的数据集,具体如下:
-
数据源和预处理:
- 数据来源:数据来自中国的三个医疗单位,包括重庆疾控中心(Data Source A)、包头医学院(Data Source B)和河南省职业病防治研究所(Data Source C)。
- 样本处理:这些血液样本数据由北京大学公共卫生学院的实验室统一处理,并制作成全切片图像(WSIs)。
- 预处理方法:由于WSIs尺寸很大,难以直接输入网络进行检测或其他下游任务,因此使用滑动窗口方法将WSIs切割成统一的512×512像素的小块。
-
检测数据准备:
- 多中心数据混合:将三个数据中心的数据混合,根据比例形成训练集和测试集。
- 多中心交叉验证:选择两个数据中心的数据作为训练集,另一个数据中心的数据作为测试集。
- 类别分布:数据集包括总计11,188个双核细胞(BCs),涵盖8152个正常BCs,1536个微核BCs(MNs),765个核芽BCs(NBs)和735个核桥BCs(NPBs)。
-
分类数据准备:
- 数据生成:包含通过细胞质生成器网络(CGNet)生成的数据。
- 数据平衡:为了确保数据平衡,随机选择1353个正常BCs作为分类数据集,以匹配MNs、扩展的NBs和NPBs的数量。
- 分类数据集组成:总共包括1353个正常BCs,1536个MNs,1421个NBs和1103个NPBs。
-
数据集的作用:
- 粗略检测:使用MS COCO检测评估指标和F1分数对BC进行评估。
- 细粒度分类:使用准确度和混淆矩阵对BC进行评估,包括正常BCs和异常BCs的分类,使用接收者操作特征(ROC)曲线、AUC(ROC曲线下面积)、敏感性和特异性作为标准。
- 定量评估:使用结构相似性指数度量(SSIM)对细胞质生成器网络生成的BCs进行定量评估。
这些数据集在文章中的作用是为所提出的BC检测和分类方法提供了必要的训练和测试数据,以验证方法的有效性和准确性。通过在这些数据集上进行实验,作者能够展示其方法在实际医疗图像分析任务中的性能。
六、SynCLay框架:基于用户定义布局的合成组织学图像生成

一作&通讯
| 角色 | 姓名 | 单位名称(中文) |
|---|---|---|
| 第一作者 | Srijay Deshpande | 华威大学计算机科学系组织图像分析中心 |
| 通讯作者 | Nasir Rajpoot | 华威大学计算机科学系组织图像分析中心;伦敦的艾伦·图灵研究所;考文垂和华威郡大学医院的病理学系;伯明翰的Histofy有限公司 |
文献概述
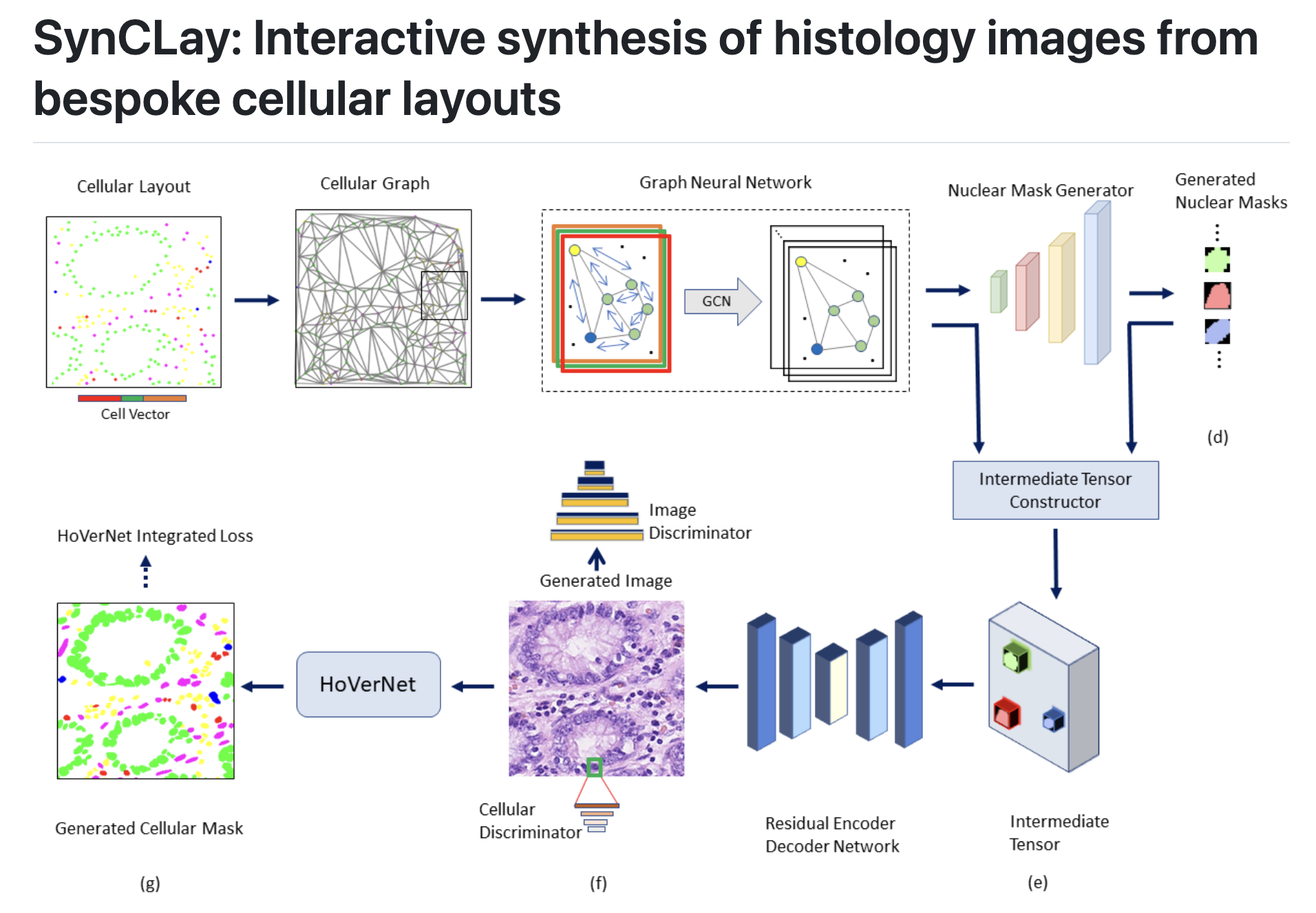
这篇文章介绍了一个名为SynCLay(Synthesis from Cellular Layouts)的新型框架,它能够根据用户定义的细胞布局生成现实且高质量的组织学图像。
SynCLay框架通过结合用户定义的细胞布局和使用生成对抗网络(GANs)以对抗方式训练,能够生成具有注释细胞边界的合成组织学图像。这些合成图像有助于研究肿瘤微环境中不同类型细胞的作用,并且可以通过最小化数据不平衡的影响来帮助设计准确的细胞组成预测器。
文章的主要贡献包括:
- 提出了一个交互式框架SynCLay,能够从定制的细胞布局生成组织图像,并允许用户控制细胞布局以生成自定义的组织图像。
- 该方法能够从用户定义的参数(如癌症分化等级和不同类型细胞的细胞密度)生成逼真的合成组织学图像及其相关的细胞计数。
- 将基于核分割模型(称为HoVerNet)的核形态损失函数整合到框架中,以提高生成的核的质量,并且能够同时生成核分割掩模。
- 通过训练病理学家的帮助评估合成图像的真实性,并展示了生成的图像与真实图像的质量相当。
- 突出了使用合成生成的结肠组织学图像数据对下游细胞组成预测和核存在检测任务的好处。
此外,文章还讨论了相关工作,包括在计算病理学中生成对抗网络(GANs)的应用,以及如何使用条件GANs来生成高质量的合成组织学图像。作者还提供了详细的实验和结果,包括使用Frechet Inception Distance(FID)和结构相似性指数(SSIM)等定量指标来评估生成图像的质量,并通过病理学家的反馈来评估图像的真实性。
最后,文章还进行了消融研究,以验证SynCLay框架设计的重要性和损失组件的有效性,并探讨了未来的研究方向。
代码仓库
https://github.com/Srijay/SynCLay-Framework
数据集
这篇文章中提到了两个数据集,它们在文章中的作用如下:
-
CoNiC数据集
- 全称: Colon Nuclei Identification and Counting challenge
- 作用: CoNiC数据集包含4981张结肠组织学的Haematoxylin和Eosin染色图像,以及相应的细胞核分割掩模。这些图像和掩模与不同的细胞类型相关,如上皮细胞、淋巴细胞、浆细胞、嗜酸性粒细胞、嗜中性粒细胞和结缔组织。文章中使用这个数据集来训练和测试SynCLay框架,以生成逼真的合成组织学图像,并进行细胞组成的评估。
-
PanNuke数据集
- 全称: Pan-cancer Nuclei dataset
- 作用: PanNuke数据集包含半自动生成的细胞核实例分割掩模,涵盖了19种不同的组织类型。该数据集由481个视场组成,其中312个是从20,000多张全切片图像中随机抽取的,这些图像来自不同的数据源和放大倍率。文章中使用这个数据集进一步训练和测试SynCLay框架,以验证其生成的合成图像在不同组织类型中的适用性和多样性。
这两个数据集为SynCLay框架提供了丰富的、经过标注的组织学图像资源,使得框架能够学习并生成具有不同细胞类型和结构的合成图像。通过这些数据集,研究人员能够评估SynCLay生成的图像在视觉上的真实性,并通过病理学家的反馈进一步验证其临床应用潜力。此外,这些数据集还支持了对框架进行消融研究,以了解不同组件对生成图像质量的影响。