生信碱移
VIM 缺失值处理
在组学分析中,尤其是在公开数据库的挖掘中,数据缺失是一个常见问题,这可能是由于样本处理、测量误差或技术限制等多种原因引起。例如,在转录组学研究中,某些基因的表达数据可能因实验失败而缺失;在使用TCGA数据库的样本进行临床关联分析时,某些样本的部分临床数据存在缺失。
常规情况下,面对数据缺失最常用的解决方法包括:
-
删除法(直接删除包含缺失值的样本或变量,这种是最常见的)
-
朴素的方法:比如均值填补(用该变量的均值填补缺失值)
-
更复杂的插补方法(如回归填补、k-最近邻填补和迭代模型填补),原理其实就是根据数据的相似性进行聚类或者建模,来获得缺失的信息。
虽然已经存在多种算法来填充缺失数据,但是由于方法的实现缺乏便捷的工具,使得多数研究者还是倾向于删除存在缺失数据的样本。小编最近冲浪的时候看到了一个R包 VIM,早在 2016 年便发表在 Journal of Statistical Software [IF: 5.4]杂志,目前已经有了600+被引。

▲ DOI: 10.18637/jss.v074.i07
VIM 包旨在通过可视化方法探究和分析数据中缺失值的结构,利用内置的填充方法对这些缺失值进行填补,并通过可视化工具验证填充过程。目前,VIM 实现了四种缺失数据的填充方法:hotdeck 填充、k 最近邻填充、回归填充和迭代鲁棒模型填充。简单介绍一下它们的原理以及在 VIM 相应的函数名:
-
hotdeck(): hotdeck 填充,选择数据集中最相似的记录,并用它们的值来替换缺失值。 -
kNN(): 通过 KNN 算法找到数据集中最接近的几个样本,然后用这些邻居样本的平均值来填补缺失值。 -
regressionImp(): 用回归模型预测缺失值,用该样本其他已知变量预测缺失变量的值。 -
matchImpute(): 通过匹配算法填补缺失值 -
irmi(): 迭代的填充方法,逐步使用数据中的所有信息来填补缺失值,每一步中,将一个变量作为目标变量进行填充,同时使用其它变量的信息来预测这个目标变量的缺失值。
本文主要介绍一下该包的使用示例,感兴趣的铁子可以自行在下面网站了解更多内容:
-
https://github.com/statistikat/VIM/
1 R包的安装与引用
该 R 包支持从 CRAN/GitHub 来源进行安装:
## Install release version from CRAN
install.packages("VIM")
## Install development version from GitHub
remotes::install_github("statistikat/VIM")
library(VIM)
2 示例数据介绍
使用内置数据集sleep,为了方便各位理解就把行名改成了 GEO 数据库的样本格式,列名则是一些相应的样本临床性状,比如睡眠、体重什么的:
data(sleep)
rownames(sleep) <- paste0("GSM222013", 1:nrow(sleep))
sleep[1:5, 1:5]
# BodyWgt BrainWgt NonD Dream Sleep
#GSM2220131 6654.000 5712.0 NA NA 3.3
#GSM2220132 1.000 6.6 6.3 2.0 8.3
#GSM2220133 3.385 44.5 NA NA 12.5
#GSM2220134 0.920 5.7 NA NA 16.5
#GSM2220135 2547.000 4603.0 2.1 1.8 3.9
注意到,前五列中的Dream和Sleep是有缺失值的。对于这两对存在缺失的数据变量,可以使用VIM提供的可视化方法进行指定展示:
x <- sleep[, c("Dream", "Sleep")]
marginplot(x)
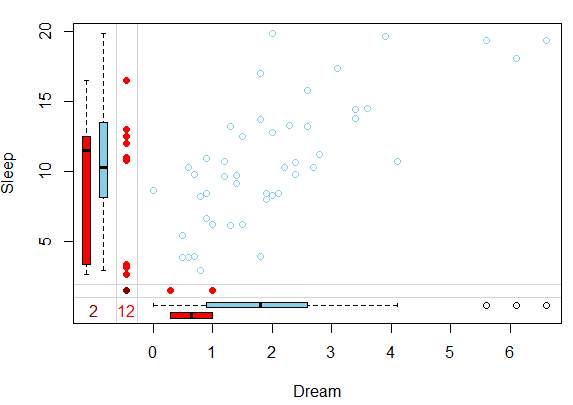
▲ 两对变量的缺失值展示:横纵坐标分别表示样本的两种变量Sleep与Dream;图中每一个点代表一个样本;点的颜色代表这个样本的数据缺失情况(蓝色点样本的Sleep与Dream皆不存在缺失值,红色点样本的Sleep或Dream存在一个缺失值,棕色点样本的Sleep与Dream都缺失了);数字代表相应颜色样本的数量(在上图中,棕色的数字2代表共有两个样本的Sleep与Dream皆缺失,红色的12代表共有12个样本的Sleep或Dream存在一个缺失值);箱式图代表存在缺失的样本与未缺失样本的指定变量差异,比如对于靠Sleep轴的那对箱式图,红色的箱子代表只存在Dream缺失的样本(由虚线隔开,在棕色点上面的那些红点),即此时具有Sleep数值(具有y轴)但由于Dream缺失所以在x轴上无法显示,此时箱式图比较的则是这些样本与未发生缺失的蓝色样本的Sleep数值差异(另一对同理)。
当然,也可以看看整个数据表的缺失情况:
a <- aggr(sleep, plot = FALSE)
plot(a, numbers = TRUE, prop = FALSE)
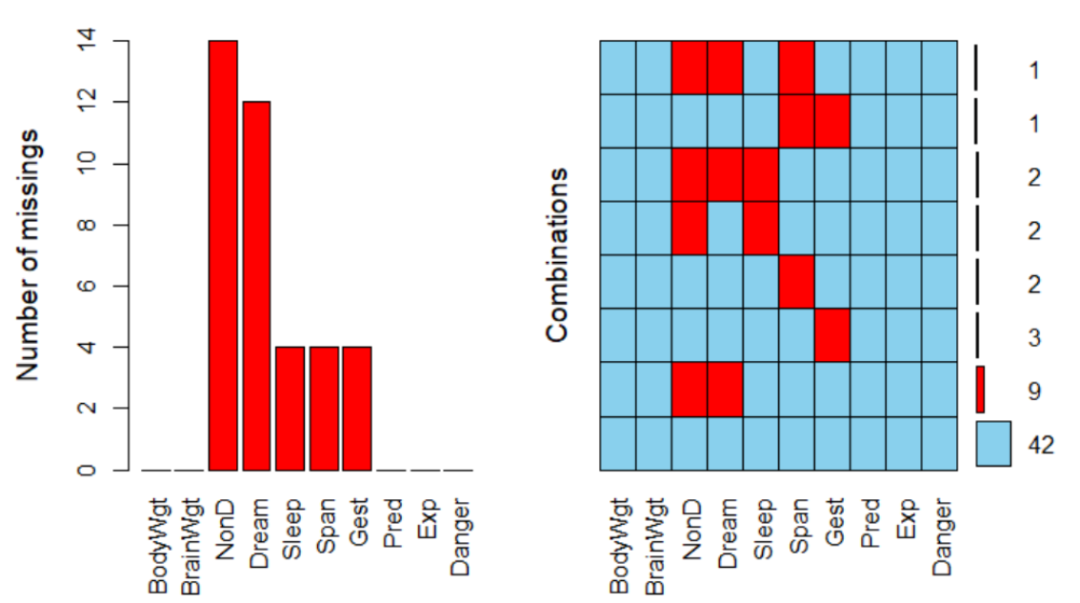
▲ 数据缺失情况的整体概括:左图代表各种变量的缺失情况,可以看到NonD的缺失值是最多的,在14个样本中存在缺失;右图展示的是样本的缺失情况,从下往上看,可以看到42个样本不存在任何缺失,9个样本存在NonD与Dream的缺失。
3 KNN算法填充缺失
① 使用 KNN 算法填补整个sleep的缺失值
sleep_imputed <- kNN(sleep)
rownames(sleep_imputed) <- rownames(sleep)
sleep_imputed[1:5, 1:5]
# BodyWgt BrainWgt NonD Dream Sleep
#GSM2220131 6654.000 5712.0 3.2 0.8 3.3
#GSM2220132 1.000 6.6 6.3 2.0 8.3
#GSM2220133 3.385 44.5 12.8 2.4 12.5
#GSM2220134 0.920 5.7 10.4 2.4 16.5
#GSM2220135 2547.000 4603.0 2.1 1.8 3.9
由kNN函数返回的表其实和原先的表没有什么区别,只是使用算法将NA的数据进行了填充。这里只演示了kNN的结果,老铁们也可以试试前面说到的其他函数算法。
② 简单自定义一个函数提取数据,可视化一下Dream和Sleep列缺失值的填充情况:
# 自定义函数提取数据用于可视化
index_finder <- function(x, x_imputed){
index <- c()
for (i in x) {
index_i <- grep(i, colnames(x_imputed))
index <- c(index, index_i[1])
}
for (i in x) {
index_i <- grep(i, colnames(x_imputed))
index <- c(index, index_i[2])
}
return(index)
}
# 可视化一下指定列插补的结果
select <- c("Dream", "Sleep")
x_imputed <- sleep_imputed[, index_finder(select, sleep_imputed)]
marginplot(x_imputed, delimiter = "_imp")
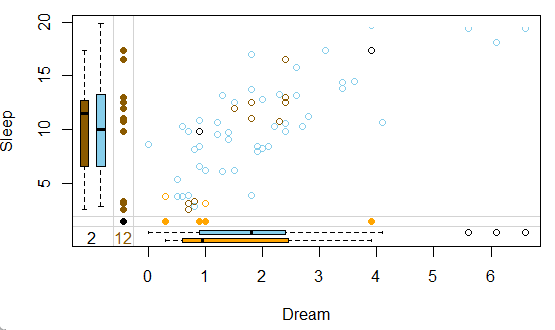
▲ 展示指定两对变量的数据填充情况:看图的方法还是一致的,只是颜色的注释变了,棕色和黄色分别是不同变量经过算法填充的数据(即原本是缺失值,通过算法进行了填充),所以这个时候在所有的蓝色样本中会出现这些填充完数据的样本。
简单易用
还有可视化的功能
对于一些难免存在缺失的数据
还是有一定应用价值的
至于有没有必要查漏补缺
还是看结果吧
算法只是理由,更多情况还是结果导向
欢迎点击关注