欢迎大家关注全网生信学习者系列:
- WX公zhong号:生信学习者
- Xiao hong书:生信学习者
- 知hu:生信学习者
- CDSN:生信学习者2
介绍
本文通过多元方差分析和典型相关分析研究微生物(species)、细胞因子(cytokine)和短链脂肪酸(SCFA)之间的相关关系。以下是两种分析的定义:
**多元方差分析(Multivariate Analysis of Variance,简称MANOVA)是一种统计方法,用于同时分析多个因变量(dependent variables)**对一个或多个自变量(independent variables)的影响。它是一种扩展了单变量方差分析(ANOVA)的技术,允许研究者检验多个响应变量是否受到一个或多个分类自变量的影响。
-
多维数据:MANOVA处理的是多维数据集,即每个观测值都有多个响应变量的测量值。
-
线性模型:它基于线性模型,其中每个因变量可以表示为自变量的线性组合加上误差项。
-
假设检验:MANOVA检验的核心是假设检验,主要检验自变量对因变量的总体影响是否显著。这包括检验自变量的主效应、交互效应以及它们对因变量的联合效应。
-
协方差矩阵:MANOVA考虑了因变量之间的相关性,通过分析协方差矩阵来评估这种相关性。
-
Wilks’ Lambda, Pillai’s Trace, Hotelling’s Trace, Roy’s Largest Root:这些都是MANOVA中常用的统计量,用于检验自变量对因变量的影响。
加载R包
library(readr)
library(openxlsx)
library(compositions)
library(tidyverse)
library(mia)
library(ggpubr)
导入数据
大家通过以下链接下载数据:
- 百度网盘链接:https://pan.baidu.com/s/1fz5tWy4mpJ7nd6C260abtg
- 提取码: 请关注WX公zhong号_生信学习者_后台发送 复现gmsb 获取提取码
df_v1 <- read_csv("./data/GMSB-data/df_v1.csv", show_col_types = FALSE)
bias_corr_species <- read_csv("./data/GMSB-data/results/outputs/bias_corr_species.csv")
raw_species <- readRDS("./data/GMSB-data/rds/primary_ancombc2_species.rds")
sig_species <- read.xlsx("./data/GMSB-data/results/outputs/res_ancombc2.xlsx", sheet = 1)
数据预处理
-
提取差异物种丰度表
-
合并分组变量和差异物种丰度表
df_v1 <- df_v1 %>%
dplyr::filter(group1 != "missing")
# Microbiome data
bias_corr_species <- bias_corr_species %>%
dplyr::rowwise() %>%
dplyr::filter(grepl("Species:", species)|grepl("Genus:", species)) %>%
dplyr::mutate(species = ifelse(grepl("Genus:", species),
paste(strsplit(species, ":")[[1]][2], "spp."),
strsplit(species, ":")[[1]][2])) %>%
dplyr::ungroup()
# Significant taxa from ANCOM-BC2
sig_species <- sig_species %>%
dplyr::filter(p_val < 0.05) %>%
.$taxon
# Subset significant taxa
df_da_species <- bias_corr_species %>%
dplyr::filter(species %in% sig_species)
df_da_species <- t(df_da_species)
colnames(df_da_species) <- df_da_species[1, ]
df_da_species <- data.frame(df_da_species[-1, , drop = FALSE], check.names = FALSE) %>%
rownames_to_column("sampleid") %>%
dplyr::mutate(across(-1, as.numeric))
df_da_species[is.na(df_da_species)] <- 0
# 合并分组标签和物种丰度表
df_add_species <- df_v1 %>%
dplyr::left_join(df_da_species, by = "sampleid")
df_add_species <- data.frame(df_add_species)
sig_species <- make.names(sig_species)
head(sig_species)
[1] "A.muciniphila" "A.onderdonkii" "Anaerovibrio.spp." "B.adolescentis" "B.caccae"
[6] "B.fragilis"
函数
-
lm_eqn:提取线性模型结果 -
plot_scatter:两个变量的散点图,关联关系
lm_eqn <- function(m){
a <- unname(coef(m))[1]
b <- unname(coef(m))[2]
p_val <- summary(m)$coef[2, "Pr(>|t|)"]
b <- ifelse(sign(b) >= 0,
paste0(" + ", format(b, digits = 2)),
paste0(" - ", format(-b, digits = 2)))
eq <- substitute(paste(italic(y) == a, b, italic(x), ", ", italic(p) == p_val),
list(a = format(a, digits = 2), b = b,
p_val = format(p_val, digits = 2)))
return(as.character(as.expression(eq)))
}
plot_scatter <- function(df, var_name, tax_name, y_lab) {
df_fig <- df %>%
dplyr::select(all_of(c(var_name, tax_name))) %>%
dplyr::rename(y = var_name) %>%
tidyr::pivot_longer(-y, names_to = "tax", values_to = "x")
df_lm <- df_fig %>%
dplyr::group_by(tax) %>%
dplyr::do(fit = lm(formula = y ~ x, data = .)) %>%
dplyr::summarise(eq = lm_eqn(m = fit))
df_ann <- df_fig %>%
dplyr::group_by(tax) %>%
dplyr::summarise(y = ifelse(mean(y, na.rm = TRUE) > 0,
0.5 * max(y, na.rm = TRUE),
0.2 * abs(mean(y, na.rm = TRUE))),
x = median(x, na.rm = TRUE)) %>%
dplyr::mutate(eq = df_lm$eq,
y_max = 1.05 * y)
fig <- df_fig %>%
ggplot(aes(x = x, y = y)) +
geom_point(alpha = 0.8, color = "#BEAED4") +
geom_smooth(method = "lm", se = TRUE, color = "skyblue",
formula = y ~ x) +
facet_wrap(.~tax, scales = "free") +
labs(x = "Micorbial abundances",
y = y_lab,
title = NULL) +
geom_blank(data = df_ann, aes(y = y_max)) +
geom_text(data = df_ann, aes(x = x, y = y, label = eq), size = 3,
parse = TRUE, inherit.aes = FALSE) +
theme_bw() +
theme(strip.background = element_rect(fill = "white"),
legend.position = "bottom",
plot.title = element_text(hjust = 0.5))
return(fig)
}
Microbiome vs. cytokines
差异物种和细胞因子的关联分析,采用**多重协方差分析(MANCOVA, Multivariate Analysis of Covariance)**方法来评估细胞因子和微生物物种之间的多变量关系
-
因变量:细胞因子
-
自变量:差异菌
t_formula <- as.formula(paste0("cbind(crp, cd14, cd163) ~ ",
paste0(sig_species, collapse = " + ")))
fit <- manova(t_formula, data = df_add_species)
summ <- summary(fit, test = "Pillai")
df_summ <- data.frame(summ$stats) %>%
rownames_to_column("Taxon") %>%
dplyr::transmute(Taxon,
approx.F = signif(approx.F, 2),
num.Df, den.Df,
P = round(`Pr..F.`, 2)) %>%
dplyr::filter(Taxon != "Residuals") %>%
dplyr::arrange(P)
head(df_summ)
cowplot::plot_grid(
plot_scatter(
df = df_add_species,
var_name = "crp",
tax_name = "Lachnospira.spp.",
y_lab = "crp"),
plot_scatter(
df = df_add_species,
var_name = "cd14",
tax_name = "Lachnospira.spp.",
y_lab = "cd14"),
plot_scatter(
df = df_add_species,
var_name = "cd163",
tax_name = "Lachnospira.spp.",
y_lab = "cd163"),
ncol = 2, align = "hv")
| Taxon | approx.F | num.Df | den.Df | P | |
|---|---|---|---|---|---|
| 1 | Lachnospira.spp. | 3.2 | 3 | 212 | 0.02 |
| 2 | RFN20.spp. | 2.8 | 3 | 212 | 0.04 |
| 3 | Succinivibrio.spp. | 1.9 | 3 | 212 | 0.13 |
| 4 | B.uniformis | 1.4 | 3 | 212 | 0.25 |
| 5 | Bifidobacterium.spp. | 1.4 | 3 | 212 | 0.25 |
| 6 | B.fragilis | 1.3 | 3 | 212 | 0.28 |
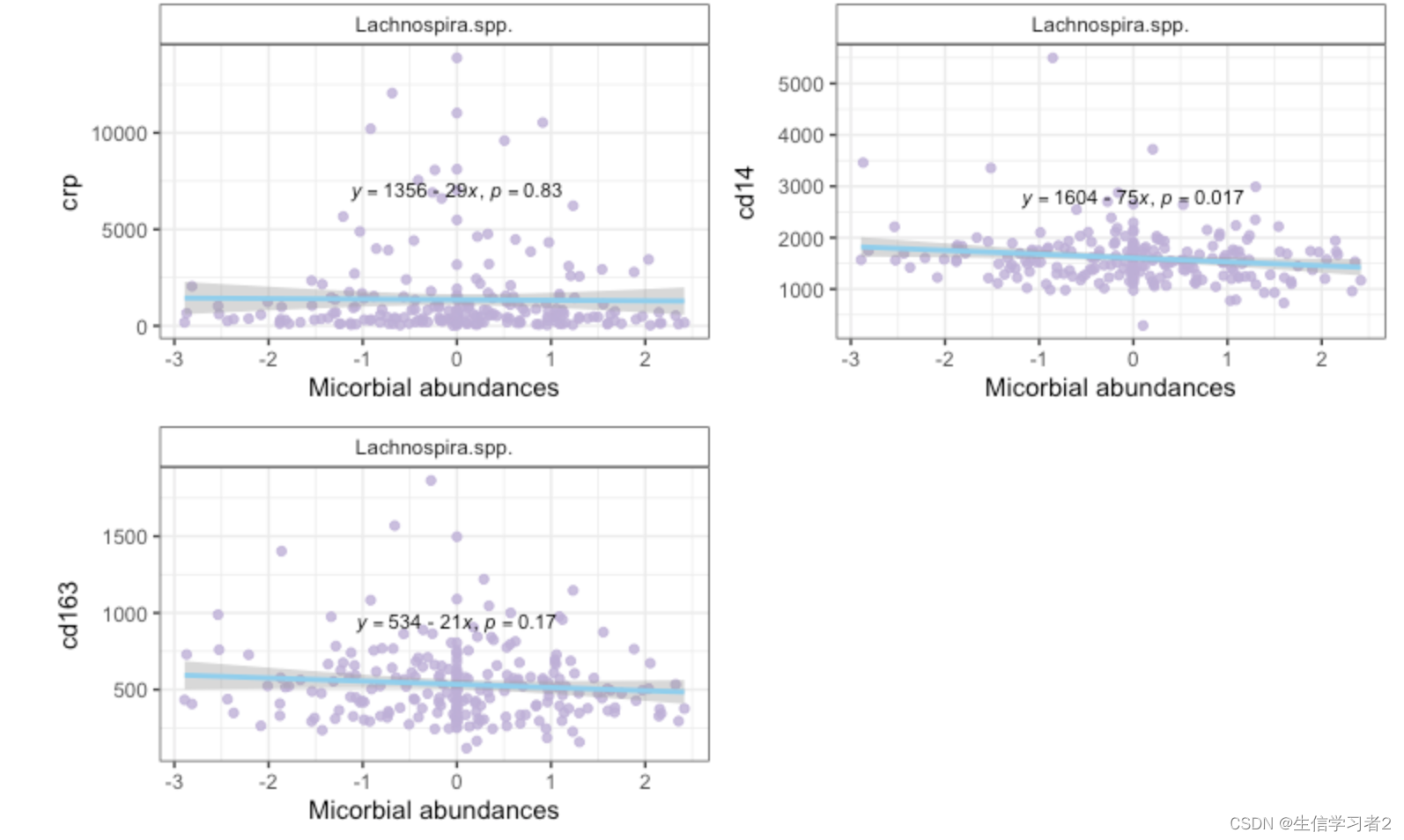
结果:自变量species对因变量细胞因子的检验结果
-
自变量
Lachnospira.spp.p值小于0.05,这表示它对至少一个因变量(crp, cd14, cd163)产生了影响,可以通过散点图查看结果; -
自变量
Lachnospira.spp.和因变量cd14存在显著负相关。
Microbiome vs. SCFAs
差异物种和短链脂肪酸的关联分析,采用**多重协方差分析(MANCOVA, Multivariate Analysis of Covariance)**方法来评估短链脂肪酸和微生物物种之间的多变量关系
-
因变量:短链脂肪酸
-
自变量:差异菌
t_formula <- as.formula(paste0("cbind(acetate, valerate) ~ ",
paste0(sig_species, collapse = " + ")))
fit <- manova(t_formula, data = df_add_species)
summ <- summary(fit, test = "Pillai")
df_summ <- data.frame(summ$stats) %>%
rownames_to_column("Taxon") %>%
dplyr::transmute(Taxon,
approx.F = signif(approx.F, 2),
num.Df, den.Df,
P = round(`Pr..F.`, 2)) %>%
dplyr::filter(Taxon != "Residuals") %>%
dplyr::arrange(P)
head(df_summ)
cowplot::plot_grid(
plot_scatter(
df = df_add_species,
var_name = "acetate",
tax_name = "B.uniformis",
y_lab = "acetate"),
plot_scatter(
df = df_add_species,
var_name = "valerate",
tax_name = "B.uniformis",
y_lab = "valerate"),
ncol = 2, align = "hv")
| Taxon | approx.F | num.Df | den.Df | P | |
|---|---|---|---|---|---|
| 1 | B.uniformis | 6.3 | 2 | 196 | 0.00 |
| 2 | Megasphaera.spp. | 3.6 | 2 | 196 | 0.03 |
| 3 | Succinivibrio.spp. | 3.4 | 2 | 196 | 0.04 |
| 4 | Butyricimonas.spp. | 2.5 | 2 | 196 | 0.09 |
| 5 | Paraprevotella.spp. | 2.4 | 2 | 196 | 0.09 |
| 6 | B.fragilis | 2.0 | 2 | 196 | 0.13 |
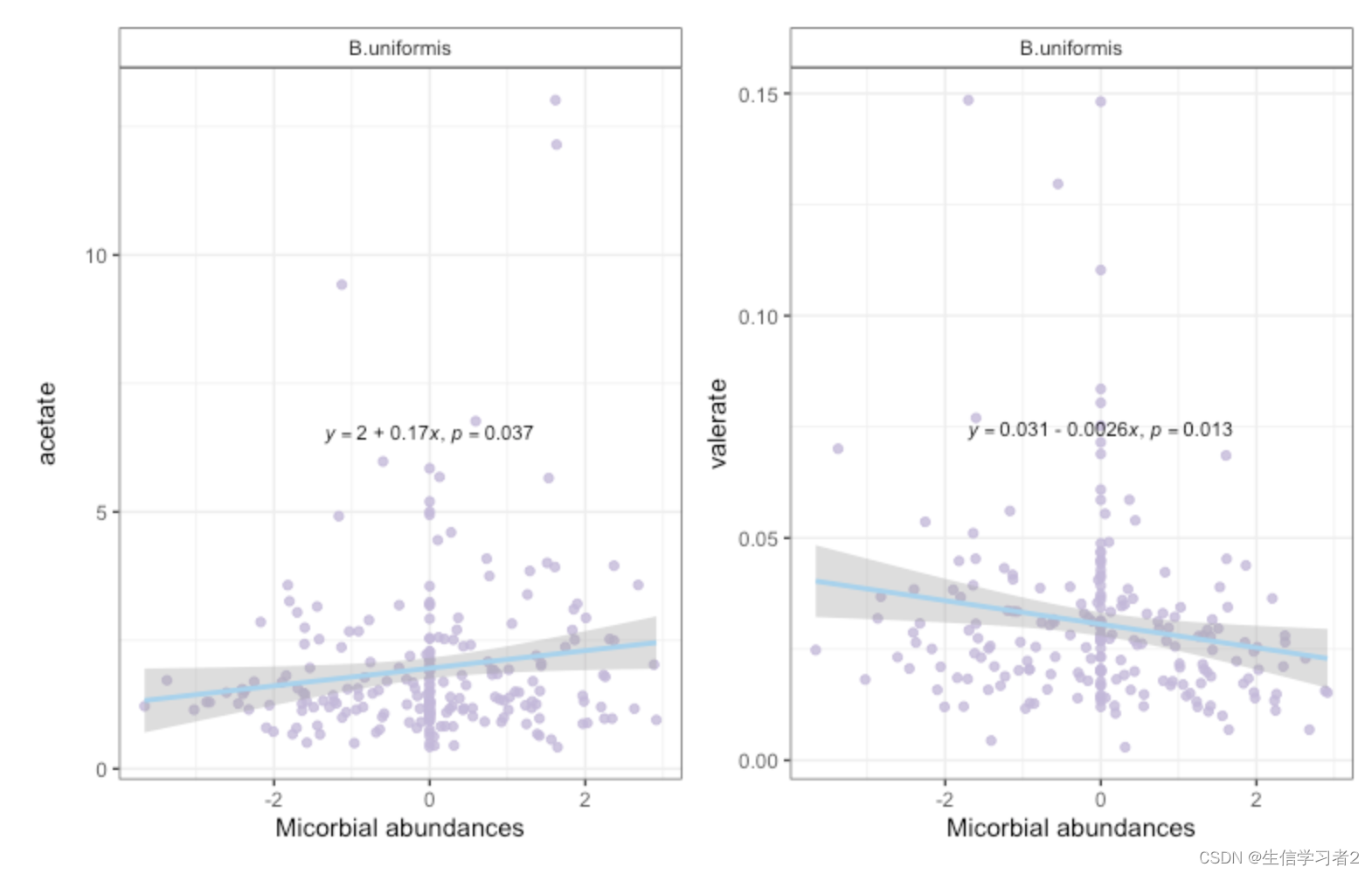
结果:自变量species对因变量短链脂肪酸的检验结果
-
自变量
B.uniformisp值小于0.05,这表示它对至少一个因变量(acetate, valerate)产生了影响,可以通过散点图查看结果; -
自变量
B.uniformis和两个因变量acetate, valerate分别存在显著正负相关。
Cytokines vs. SCFAs
细胞因子和短链脂肪酸的关联分析,采用**多重协方差分析(MANCOVA, Multivariate Analysis of Covariance)**方法来评估细胞因子和短链脂肪酸之间的多变量关系
-
因变量:细胞因子
-
自变量:短链脂肪酸
fit <- manova(cbind(crp, cd14, cd163) ~
acetate + valerate,
data = df_v1)
summ <- summary(fit, test = "Pillai")
df_summ <- data.frame(summ$stats) %>%
rownames_to_column("Taxon") %>%
dplyr::transmute(Taxon,
approx.F = signif(approx.F, 2),
num.Df, den.Df,
P = round(`Pr..F.`, 2)) %>%
dplyr::filter(Taxon != "Residuals") %>%
dplyr::arrange(P)
head(df_summ)
cowplot::plot_grid(
plot_scatter(
df = df_add_species,
var_name = "crp",
tax_name = "acetate",
y_lab = "crp"),
plot_scatter(
df = df_add_species,
var_name = "cd14",
tax_name = "acetate",
y_lab = "cd14"),
ncol = 2, align = "hv")
| Taxon | approx.F | num.Df | den.Df | P | |
|---|---|---|---|---|---|
| 1 | acetate | 2.5 | 3 | 216 | 0.06 |
| 2 | valerate | 1.2 | 3 | 216 | 0.30 |
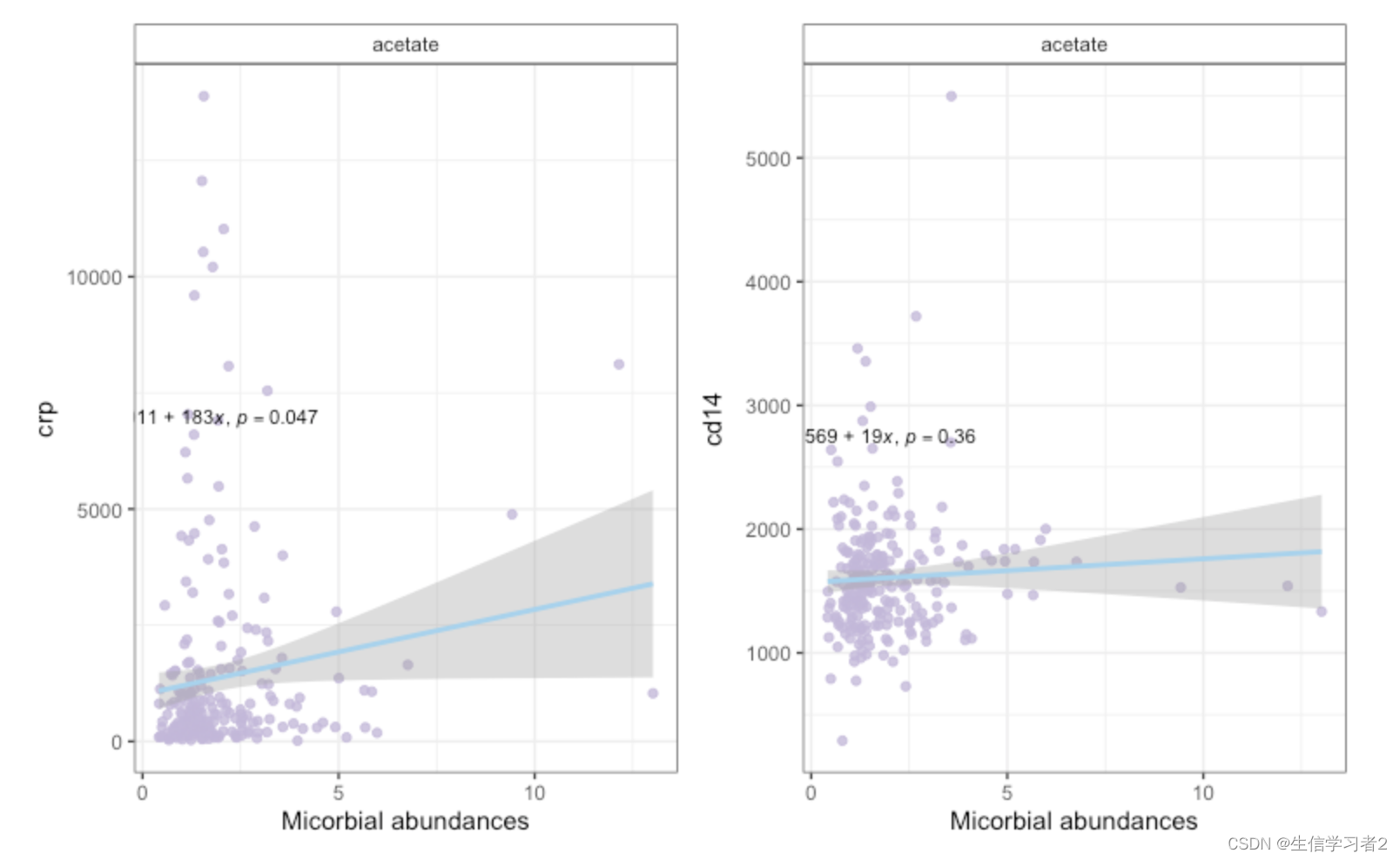
结果:自变量短链脂肪酸对因变量细胞因子的检验结果
-
自变量
acetatep = 0.06,这表示它对至少一个因变量(crp, cd14, cd163)产生了轻微影响,可以通过散点图查看结果; -
自变量
acetate和因变量crp存在显著正相关。