原位酶谱法高分辨率实时检测土壤微界面酶活分布
High resolution real-time detection of soil enzyme activity distribution by in situ zymography
魏晓梦1, 2、魏亮1, 2、郝存抗1, 2、祝贞科1, 2、吴金水1, 2、葛体达1, 2, *
1中国科学院亚热带农业生态研究所,中国科学院亚热带农业生态过程重点实验室,长沙;2中国科学院长沙农业环境观测研究站,长沙
*通讯作者邮箱: gtd@isa.ac.cn
引用格式:魏晓梦, 魏亮, 郝存抗, 祝贞科, 吴金水, 葛体达. (2021). 原位酶谱法高分辨率实时检测土壤微界面酶活分布. // 微生物组实验手册. Bio-101: e2003833. DOI: 10.21769/BioProtoc.2003833.
How to cite: Yunyun Gao, Kai Peng, Defeng Bai, et al. 2024. The Microbiome Protocols eBook initiative: Building a bridge to microbiome research. iMeta 3: e182. https://doi.org/10.1002/imt2.182
摘要:荧光标记的底物与土壤微界面的胞外酶充分反应,释放出的荧光基团被原位保留,并可在355 nm左右的激发光下发射出460 nm左右的可见光 (M. Vepsäläinen等, 2001)。由于发射光强度与荧光基团释放量有良好的回归关系,而酶促反应释放的荧光基团与被降解的荧光底物在物质的量上相等,可通过相机捕捉发射光的强度与分布情况,实现对酶活性的原位高分辨率定量检测。
关键词:原位酶谱、土壤微界面、根土界面、酶活性、荧光底物
一、根土界面酶活性原位检测
材料与试剂
1.离心管 (预灭菌)
2.试管 (预灭菌)
3.500 ml容量瓶
4.玻璃棒
5.剪刀
6.镊子
7.酒精灯
8.酒精棉
9.聚酰胺膜 (0.45 μm)
10.铝箔纸
11.糙纸
12.吗啡啉乙磺酸半钠盐MES (C6H13NO4SNa0.5),相对分子质量206.73 g/mol
13.TRIZMA-Base,相对分子质量121.14 g/mol
14.TRIZMA-HCl,相对分子质量157.60 g/mol
15.4-甲基伞形酮 (MUF),相对分子质量176.17 g/mol
16.7-氨基-4-甲基香豆素 (AMC),相对分子质量175.18 g/mol
17.带MUF或AMC标记的酶底物
仪器设备
1.1/10000天平
2.斡旋振动仪
3.水浴锅
4.恒温培养箱
5.紫外灯
6.数码单反相机
7.460 nm带通滤光片
实验步骤
一、溶液配备
配置前根据实验酶谱实验量计算大致所需酶底物溶液量,避免试剂浪费。一般一张10 x 10 cm2的聚酰胺膜需1.92 ml酶底物溶液。
1.缓冲液储备液配备
1.1100 mM MES储备液 (用于配置MUF标记的底物):称取2.0673 g,转移至烧杯,加入适量灭菌水,用玻璃棒搅拌,转移至100 ml容量瓶,用灭菌水定容至100 ml;
1.2100 mM TRIZMA储备液 (用于配置AMC标记的底物):称取1.2114 g TRIZ-Base &1.5760 g TRIZ-HCl 转移至烧杯,加入适量灭菌水,用玻璃棒搅拌,转移至100 ml容量瓶,用灭菌水定容至100 ml;配置完成后转移至广口瓶,并放置于4 °C保存。
注:储备液可在于4 °C储存2个月
2.酶底物配备
不同的酶在不同土壤条件最适酶底物浓度存在差异,通常为1 mM~10 mM。为节约试剂,同时达到最佳实验效果,建议通过预实验确定底物浓度。不同的酶对应的底物不同 (表1),下面以配置100 ml 10 mM的β-D-glucosidase举例:
用5 ml移液枪吸取100 mM的MES缓冲液储备液,用灭菌水稀释成10 mM缓冲液工作液待用。称取4-MUF-β-1,4-D-glucoside 338.31 mg,转移至烧杯,加适量MES缓冲液工作液,用玻璃棒搅拌溶解 (也可以通过涡旋、水浴加热使其溶解,但注意水浴温度应低于60 °C),用MES缓冲液工作液定容至100 ml。
表1:底物及其摩尔质量和相应的缓冲液列表
酶名称 | 英文名 | 对应的底物 | 化学式 | 相对分子质量(g/mol) | 缓冲液 | 酶的功能 |
β-葡萄糖苷酶 | β-glucosidase | 4-Methylumbelliferone-β-D-Glucoside | C16H18O8 | 338.31 | MES | 从纤维素中释放葡萄糖 |
纤维素二糖水解酶 | Cellobiohydrolase | 4-Methylumbelliferone-β-D-Cellobioside | C22H28O13 | 500.45 | MES | 从纤维素中释放双糖 |
木聚糖酶 | Xylanase | 4-Methylumbelliferone-β-D-Xylopyranoside | C15H16O7 | 308.28 | MES | 从半纤维素中释放木糖 |
几丁质酶 | Chitinase | 4-Methylumbelliferone-N-Acetyl-β-D-glucosaminide | C18H21NO8 | 379.36 | MES | 从几丁质中释放N-乙酰氨基葡萄糖 |
磷酸酶 | Phosphatase | 4-Methylumbelliferone-phosphate | C10H10O6PNa2 | 300.11 | MES | 释放附着的磷酸基团 |
亮氨酸氨基肽酶 | Leucine-aminopeptidase | L-Leucine-7-amido-methylcoumarin hydrochloride | C16H20N2O3 HCl | 324.81 | TRIZMA | 肽键的水解 |
酪氨酸氨基肽酶 | Tyrosine-aminopeptidase | L-Tyrosine-7-amido-methylcoumarin | C19H18N2O4 | 338.36 | TRIZMA | 肽键的水解 |
3.标准溶液配置
称取0.0881 mg MUF或0.0876 mg AMC于灭菌烧杯中,加25 ml甲醇溶解,灭菌水定容至50 ml,配置10 mM 的MUF或AMC标线母液。将母液连续梯度稀释,获取6 mM、4 mM、2 mM、1 mM、0.5 mM、0.25 mM、0.01 mM的浓度梯度。
注:以上操作所用实验器材均需做灭菌处理。选择的溶液浓度需涵盖样品的所有范围,超出标线范围的样品校正结果可能被高估或低估。
二、样品准备
准备好植物种子或幼苗,种植在具有活动透明根窗的根盒中,于适宜条件下培养。作物生长时,根盒保持根窗朝下倾斜45° - 60°,以确保根系在重力的作用下沿根窗生长(图1),持续观察根窗面的根系生长情况,当观察到清晰、健壮的根系时即可进行原位酶谱实验,或根据自己的实验需要设置测定时间 (T. Ge等, 2017)。
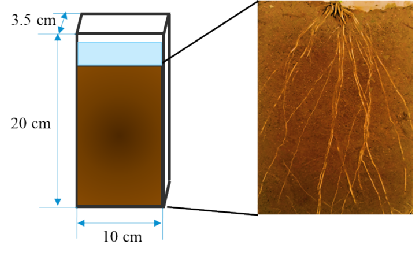
图1. 根窗示意图
三、反应
1.将聚酰胺膜裁剪成与根窗相同的尺寸,转移适量 (可刚好饱和浸润聚酰胺膜,约2 ml/10×10 cm2) 酶底物溶液至灭菌试管中,试管尺寸由膜的尺寸确定。将聚酰胺膜卷成蛋卷状,垂直放入试管,用封口膜封口,水平放置试管在试验台上,沿相同方向滚动20-30次,保证酶底物均匀分布在聚酰胺膜。
2.将根盒根窗面向上水平放置,小心打开根窗,整个过程应均匀用力,保持根土界面的完整性。
3.用镊子从试管中取出聚酰胺膜,展开后平整贴在根土界面上,避免气泡产生,快速将事先剪好的铝箔纸平整覆盖在膜上,铝箔纸比膜稍大,以完全遮蔽光线,以防酶促反应产生的荧光基团被紫外淬灭。在铝箔纸上覆盖海绵薄膜或者草纸 (3-4张并折叠为膜相当大小),保证盖子与膜之间空隙被完全填充,然后盖好根窗,然后保持根窗面向上水平放置培养1 h。培养时可用重物压紧根窗,保证土样与膜紧密接触且各部位受力均匀。
四、成像
打开根窗,依次移除填充层和铝箔纸,自上而下取下聚酰胺膜,用软刷移除膜上附着的土壤颗粒等杂质,避光条件下快速转移到暗室中,置于350 nm的紫外灯下,采用蝴蝶光源或面光源保证整张膜受光均匀,调整好相机参数和与膜的相对位置 (整个实验过程要保持相机状态一致),在膜的一侧平行放置刻度尺,拍摄照片。为排除环境反射光干扰,可在镜头上安装中心波长为460 nm的带通滤光片。、
五、标准品成像
取面积为2×2 cm2的聚酰胺膜,分别在不同浓度的MUF或AMC溶液中饱和浸润,同时记录完全浸润此聚酰胺膜所需的溶液体积,在与样品酶谱同样的条件下进行成像,用于后续标准曲线绘制。
二、土壤团聚体表面酶活原位分布
材料与试剂
1.套筛
2.菲林胶片
3.硅基片
4.SU-8光刻胶
5.聚乙二醇甲基醚丙烯酸酯 (PEGMEA)
6.异丙醇
7.PDMS预聚体
8.PDMS固化剂
9.载玻片
10.毛细吸管
11.培养皿
12.吗啡啉乙磺酸半钠盐MES (C6H13NO4SNa0.5),相对分子质量206.73 g/mol
13.TRIZMA-Base,相对分子质量121.14 g/mol
14.TRIZMA-HCl,相对分子质量157.60 g/mol
15.4-甲基伞形酮 (MUF),相对分子质量176.17 g/mol
16.7-氨基-4-甲基香豆素 (AMC),相对分子质量175.18 g/mol
17.带MUF或AMC标记的酶底物
仪器设备
1.紫外深度光刻机 (URE-2000/25L型,上海光学机械厂)
2.数控热平板
3.匀胶机
4.真空泵
5.烘箱
6.激光共聚焦显微镜
实验步骤
一、缓冲液配置
1.100 mM MES储备液 (用于配置MUF标记的底物):称取2.0673 g,转移至烧杯,加入适量灭菌水,用玻璃棒搅拌,转移至100 ml容量瓶,用灭菌水定容至100 ml;
2.100 mM TRIZMA储备液 (用于配置AMC标记的底物):称取1.2114 g TRIZ-Base &1.5760 g TRIZ-HCl转移至烧杯,加入适量灭菌水,用玻璃棒搅拌,转移至100 ml容量瓶,用灭菌水定容至100 ml;配置完成后转移至广口瓶并于4 °C保存。
注:储备液可在于4 °C储存2个月。
二、酶底物配置
不同的酶在不同土壤条件最适酶底物浓度存在差异,通常为1 mM~10 mM。为节约试剂,同时达到最佳实验效果,建议通过预实验确定底物浓度。下面以配置100 ml 10 mM的β-D-glucosidase举例:
用5 ml移液枪吸取100 mM的MES缓冲液储备液,用灭菌水稀释成10 mM缓冲液工作液待用。称取4-MUF-β-1,4-D-glucoside 338.31 mg,转移至烧杯,加适量MES缓冲液工作液,用玻璃棒搅拌溶解 (也可以通过涡旋、水浴加热使其溶解,水浴温度应低于60 °C),用MES缓冲液工作液定容至100ml。
三、团聚体微反应室制作[3]
1.掩膜制作
使用 Auto CAD 软件设计实验所需图案,此处以直径为 2 mm、间隔为 0.5 mm 的 3 × 3 圆形微阵列为例,通过 25400 dpi高分辨率激光打印机打印在菲林胶片上,制成掩膜,一张掩膜可打印多个微阵列,从而实现微反应室的高通量制作 (图2)。掩膜中灰色区域为透光区域,而其余黑色部分为不透光区域。

图2. 掩膜示意图
2.SU-8阳模制作
1.
2.
2.1硅基片清洗:取硅基片于500 ml的烧杯中,两个硅基片间用丙酮超声 10 分钟后去除有机污染物,再采用双氧水-浓硫酸混合液 (体积比1:3) 清洗3小时后,超声30分钟。最后,将超纯水多次超声清洗的硅片,在120 °C环境热平板上彻底烘干。
2.2匀胶:取1-2滴SU-8光刻胶滴在硅片中心,在匀胶机上900 x g 10 s、1500 x g 30 s匀胶,形成约80 μm厚的胶层。
2.3前烘:将匀胶后的硅片放在热平板上前烘,条件为65 °C 6 min,95 °C 10 min。
2.4光刻:将掩膜图案与匀好胶的硅片对准,紫外照射10 s。透光区域紫外线和光刻胶反应固化,不透光区域则未固化。
2.5后烘与显影:将光刻后的硅片放在电平板上后烘,条件为65 °C 3 min,95 °C 6 min。用PEGMEA将未经紫外曝光的光刻胶溶解掉,待图像显现完整后用异丙醇终止反应,并于去离子水清洗干净,将显影后的硅片放在平板加热器上放在热平板坚模,条件为65 °C 5 min,再135 °C 2 h。
3.PDMS印章制作
取PDMS预聚体和固化剂以10:1质量比混合,充分搅拌,用真空泵去除PDMS中的气泡后,倒在SU-8模板上,厚度为0.2 - 0.4 mm。常温静置5 min,烘箱中45 °C固化3 h,即获得PDMS印章。
4.微反应室构建
将酒精清洗、干燥后的载玻片和PDMS印章置于氧等离子体清洗仪中进行表面亲水化改性,条件为电压 420 V, 腔内氧气流速 800 ml min-1, 时长 80 s。将改性后的玻片和 PDMS 印章迅速贴合,10 s后撕下印章,接触了 PDMS 的玻片因PDMS 薄膜粘连在玻片上而变成疏水性区域,而未接触到 PDMS 的圆形区域为亲水界面,从而形成微反应室。
四、团聚体分离与加载
取0.5 g鲜土于50 ml的离心管中,加20 ml PBS缓冲液,横置在摇床上,200 x g震荡2 h以分散团聚体。选择适当孔径的不锈钢筛,实现不同粒级的团聚体分离,收集所需粒级团聚体于50 ml离心管中,用10 ml PBS缓冲液冲洗数次,至残留粘粒和非水稳性颗粒完全去除。静置使团聚体沉淀,去除大部分上清,保留约1 ml缓冲液。震荡重悬浮团聚体,并转移到2 ml的离心管中,用毛细管吸取团聚体,滴在等离子体清洗仪亲水改性后的载玻片上,使缓冲液液滴在载玻片上分散,并包裹团聚体充分分离,另取一只毛细管(根据需要的团聚体颗粒大小确定毛细管内径),吸取单个团聚体,将毛细管口与微反应室亲水区接触,在重力与表面张力的作用下将团聚体加载在微反应室中。每个微点阵有9个微反应室,可同时观察9个单团聚体。
注:团聚体在载玻片上的停留时间应不超过5 min内完成,时间过久团聚体易破碎。
五、反应与成像
用移液枪向微反应室中加入适量酶底物溶液,根据团聚体大小调整底物添加量,以刚好淹没团聚体为宜。每个微点阵中随机选取3个团聚体,以MES/TRIZMA缓冲液替代酶底物溶液,作为空白对照。将微点阵放在培养皿中,恒温恒湿避光培养1 h。取出微点阵,在激光共聚焦显微镜下观察成像。由于团聚体透光性较差,激光共聚焦显微镜以倒置型为宜。
结果与分析
以下图像处理流程主要针对方案 (一) 根土界面酶活性原位检测所获取的图像数据,方案 (二) 土壤团聚体表面酶活原位分布图像可用激光共聚焦显微镜自带图像处理工具处理,仅以下述方法辅助。
一、几何校正
由于拍摄角度或不同型号相机成像偏差的问题,可能导致拍摄的图片几何形状失真,因此获得的酶谱图片首先判断是否需要进行几何校正,可以刻度尺为参照。校正后的图像可能存在部分区域的图像数据的丢失,需要通过插值的方法进行。如下图所示:
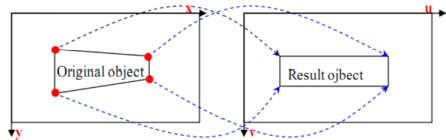
图3. 几何校正示意图
具体办法:打开ACDSee Pro,在编辑状态下打开“透视校正”功能,选择显示网络线,拖动网络线外边框的一个角向外拖动,直到线条垂直。
注:也可根据自己熟悉的操作,使用Photoshop或Image J等软件进行几何校正。但这种调节均是有损调节,会导致图像信息的损失,建议尽量在拍摄过程中保持镜头与膜的平行,避免形变。
二、灰度校正及伪彩化
图像在成像过程中,往往由于光照、摄像、传感器灵敏度以及光学系统等的不均匀性而引起图像某些部分较暗或较亮。对这类图像使用灰度级修正,能够获得满意的视觉效果。针对酶谱图片,在相机设置和位置不改变的情况下,主要是检查图片背景是否一致,可用刻度尺作为背景参考。曝光强弱不同会缩小或扩大同一实验中处理间的差异。具体办法 (之后均以Image J为例):Image-Type-8 bit;之后选择任意背景区域,进行分析,查看Analyze-Measure的结果。如果不一致,则需要根据Process-Math-Add/Subtract命令来做小幅度调整。选择膜所在范围后,Image-Crop/Duplicate,将膜以外的视野去掉。
上伪彩:由于人眼分辨率不同彩色的能力比分辨不同灰度级的能力强,因此可以将人眼难以分辨的灰度变化施以不同的色彩来提高识别率。具体步骤:Image-Lookup tables-选择适合自己的结果的样式。调整图片到最佳显示状态:Image-Adjust-Brightness/Contrast-Auto/Set.
结果如下图所示:

图4. 数据处理过程示意图
三、定量分析
提取标准图像灰度值,建立灰度值和酶活性之间的标准曲线,以定量分析原位酶活性。后续分析均需要以灰度转换为酶活性之后的结果为依据。具体步骤:Analyze-Calibrate-输入浓度梯度及对应的灰度值,选择Function-OK,显示结果需要R2 > 0.99。 添加标尺:Analyze-Tools-Calibration bar-调整显示参数-OK,即可用作结果展示。
热区定量:计算非根际土壤酶活性的的标准差 (SD) 和平均值 (a reference = 1.0 ± SD)。酶活性强度高于Mean+2SD 则被视为热区 (N. Bilyera等, 2020)。具体操作办法:Image-Adjust-Threshold。
根际范围定量:从根际中心到非根际土壤酶活性会有快速降低至平稳的变化趋势,可将该距离视为根际范围。具体操作步骤:以任意单独根系为参照,画小短线,Analyze-Plot Profile-选择Live按钮之后拖动小短线,找到合适的根际变化趋势,List可查看数据结果确定受根际影响范围大小,最终根据像素点 (pixels)及图片分辨率将横轴单位转换为mm即可得到根际范围。
方法评价
一.适用范围及改进
本方法分为两部分,第一部分基于目前广泛使用的酶谱方法,用于原位可视化植物根际酶活性分布及酶活性热区定量;第二部分是将之前原位酶谱方法与激光共聚焦显微镜结合,能够将原来mm级的检测扩展到μm级,提高了酶活性分布的检测分辨率,其适用范围也相应得到扩展,同时将底物直接添加到土壤中,避免了聚酰胺膜自身的荧光强度影响。
二.局限性
尽管本文已经对酶谱方法有一定的改进,但仍存在一定局限性:
1.目前仅局限于测定可用荧光法的土壤水解酶,对基于比色法测定的氧化酶等仍存在技术难题,如氧气的控制不能很准确,没有相应的荧光基团底物。
2.目前酶谱的结果基本基于二维平面,三维空间分布的实现难度较大;虽然激光共聚焦能够实现三维展示,但其范围比较小,难以适用根际酶活分布等大范围酶活的测定。
3.酶谱数据的处理尚没有统一的标准,导致不同实验室结果之间的比较存在不确定性。
4.相机的性能等对结果准确性影响比较大,因此应尽量选择配置高的相机,但这无疑增加了测定设备的成本;与激光共聚焦显微镜结合的酶谱方法也会因设备昂贵而导致使用广泛性受限。
参考文献
1.M. Vepsäläinen, S. Kukkonen, M. Vestberg, H. Sirviö, R. Niemi.(2001). Application of soil enzyme activity test kit in a field experiment. Soil Biol. Biochem. 33, 1665–1672.
2.T. Ge, X. Wei, B. Razavi, Z. Zhu, Y. Hu, Y. Kuzyakov, et al. (2017). Stability and dynamics of enzyme activity patterns in the rice rhizosphere: effects of plant growth and temperature. Soil Biol. Biochem. 113, 108–115.
3.X. Huang, Y. Li, B. Liu, G. Guggenberger, O. Shibistova and Z. Zhu, et al.(2017). SoilChip-XPS integrated technique to study formation of soil biogeochemical interfaces. Soil Biol. Biochem. 113, 71–79.
4.N. Bilyera, I. Kuzyakov, A. Guberd, B. Razavi, Y. Kuzyakov. (2020). How “hot” are hotspots: Statistically localizing the high-activity areas on soil and rhizosphere images - ScienceDirect. Rhizosphere. 16, 100259.
宏基因组推荐
4月12-14日,微生物组-扩增子16S分析
5月17-19日,微生物组-宏基因组分析
人满即开 | 论文作图和统计分析培训班
一站式论文提升服务,助您顺利发高分论文!
本公众号现全面开放投稿,希望文章作者讲出自己的科研故事,分享论文的精华与亮点。投稿请联系小编(微信号:yongxinliu 或 meta-genomics)
猜你喜欢
iMeta高引文章 fastp 复杂热图 ggtree 绘图imageGP 网络iNAP
iMeta网页工具 代谢组MetOrigin 美吉云乳酸化预测DeepKla
iMeta综述 肠菌菌群 植物菌群 口腔菌群 蛋白质结构预测
10000+:菌群分析 宝宝与猫狗 梅毒狂想曲 提DNA发Nature
系列教程:微生物组入门 Biostar 微生物组 宏基因组
专业技能:学术图表 高分文章 生信宝典 不可或缺的人
一文读懂:宏基因组 寄生虫益处 进化树 必备技能:提问 搜索 Endnote
扩增子分析:图表解读 分析流程 统计绘图
16S功能预测 PICRUSt FAPROTAX Bugbase Tax4Fun
生物科普: 肠道细菌 人体上的生命 生命大跃进 细胞暗战 人体奥秘
写在后面
为鼓励读者交流快速解决科研困难,我们建立了“宏基因组”讨论群,己有国内外6000+ 科研人员加入。请添加主编微信meta-genomics带你入群,务必备注“姓名-单位-研究方向-职称/年级”。高级职称请注明身份,另有海内外微生物PI群供大佬合作交流。技术问题寻求帮助,首先阅读《如何优雅的提问》学习解决问题思路,仍未解决群内讨论,问题不私聊,帮助同行。
点击阅读原文