Scanpy包,用与数据整合和批次处理,包含批次效应的BBKNN算法和用于对比的ingest基础算法比较,及其原理简介。
1. 依赖:
(1)数据集(全部需要挂VPN):
- PBMC:pbmc3k_processed()(需要下载);pbmc68k_reduced()(scanpy自带)
- Pancreas(需要下载)
(2)Python包:Scanpy、BBKNN
2. PBMC数据集
导入所需的包
import scanpy as sc
import pandas as pd
import seaborn as sns
# 参考数据集(已预处理、降维、聚类、注释)
adata_ref = sc.datasets.pbmc3k_processed() # this is an earlier version of the dataset from t
# 参考数据集(已预处理、降维、聚类、注释)
adata = sc.datasets.pbmc68k_reduced()
print(adata_ref)
生成:
上面下载数据自动放入这里
data
pancreas.h5ad
pbmc3k_processed.h5ad
2.1 重点数据结构分析
# 1. adata_ref
AnnData object with n_obs × n_vars = 2638 × 1838
obs: 'n_genes', 'percent_mito', 'n_counts', 'louvain'
var: 'n_cells'
uns: 'draw_graph', 'louvain', 'louvain_colors', 'neighbors', 'pca', 'rank_genes_groups'
obsm: 'X_pca', 'X_tsne', 'X_umap', 'X_draw_graph_fr'
varm: 'PCs'
obsp: 'distances', 'connectivities'
在 `scanpy` 中,`AnnData`(Annotated Data)是一个用于存储和操作单细胞RNA-seq等生物学数据的数据结构。以下是对`adata_ref`对象中各个字段的解释:
- `obs`:观测(observations)信息,即每个细胞或样本的元信息。在这个例子中包括:
- `'n_genes'`:每个细胞中表达的基因数量。
- `'percent_mito'`:每个细胞中线粒体基因的百分比。
- `'n_counts'`:每个细胞的总计数。
- `'louvain'`:【聚类结果,表示每个细胞属于哪个聚类。】
- `var`:变量信息,即基因的元信息。在这个例子中包括:
- `'n_cells'`:每个基因在多少个细胞中被检测到。
- `uns`:未结构化的数据,可以存储各种附加信息。在这个例子中包括:
- `'draw_graph'`:用于存储绘图图形的信息。
- `'louvain'`:用于存储Louvain聚类的信息。
- `'louvain_colors'`:Louvain聚类结果的颜色映射。
- `'neighbors'`:用于存储邻域信息的数据。
- `'pca'`:用于存储主成分分析(PCA)的信息。
- `'rank_genes_groups'`:用于存储基因组中基因排序的信息。
- `obsm`:观测矩阵,包含与观测相关的矩阵数据。在这个例子中包括:
- `'X_pca'`:PCA降维后的坐标。
- `'X_tsne'`:t-SNE降维后的坐标。
- `'X_umap'`:UMAP降维后的坐标。
- `'X_draw_graph_fr'`:绘图图形的坐标。
- `varm`:变量矩阵,包含与变量相关的矩阵数据。在这个例子中包括:
- `'PCs'`:主成分分析的主成分。
- `obsp`:观测矩阵中的矩阵,包含与观测相关的矩阵数据。在这个例子中包括:
- `'distances'`:细胞之间的距离矩阵。
- `'connectivities'`:细胞之间的连接性矩阵。
这些字段提供了关于单细胞RNA-seq数据集的丰富信息,包括细胞的特征、基因的特征、降维后的坐标、聚类结果等。
# 2. adata
AnnData object with n_obs × n_vars = 700 × 765
obs: 'bulk_labels', 'n_genes', 'percent_mito', 'n_counts', 'S_score', 'G2M_score', 'phase', 'louvain'
var: 'n_counts', 'means', 'dispersions', 'dispersions_norm', 'highly_variable'
uns: 'bulk_labels_colors', 'louvain', 'louvain_colors', 'neighbors', 'pca', 'rank_genes_groups'
obsm: 'X_pca', 'X_umap'
varm: 'PCs'
obsp: 'distances', 'connectivities'
2.2 原始数据可视化
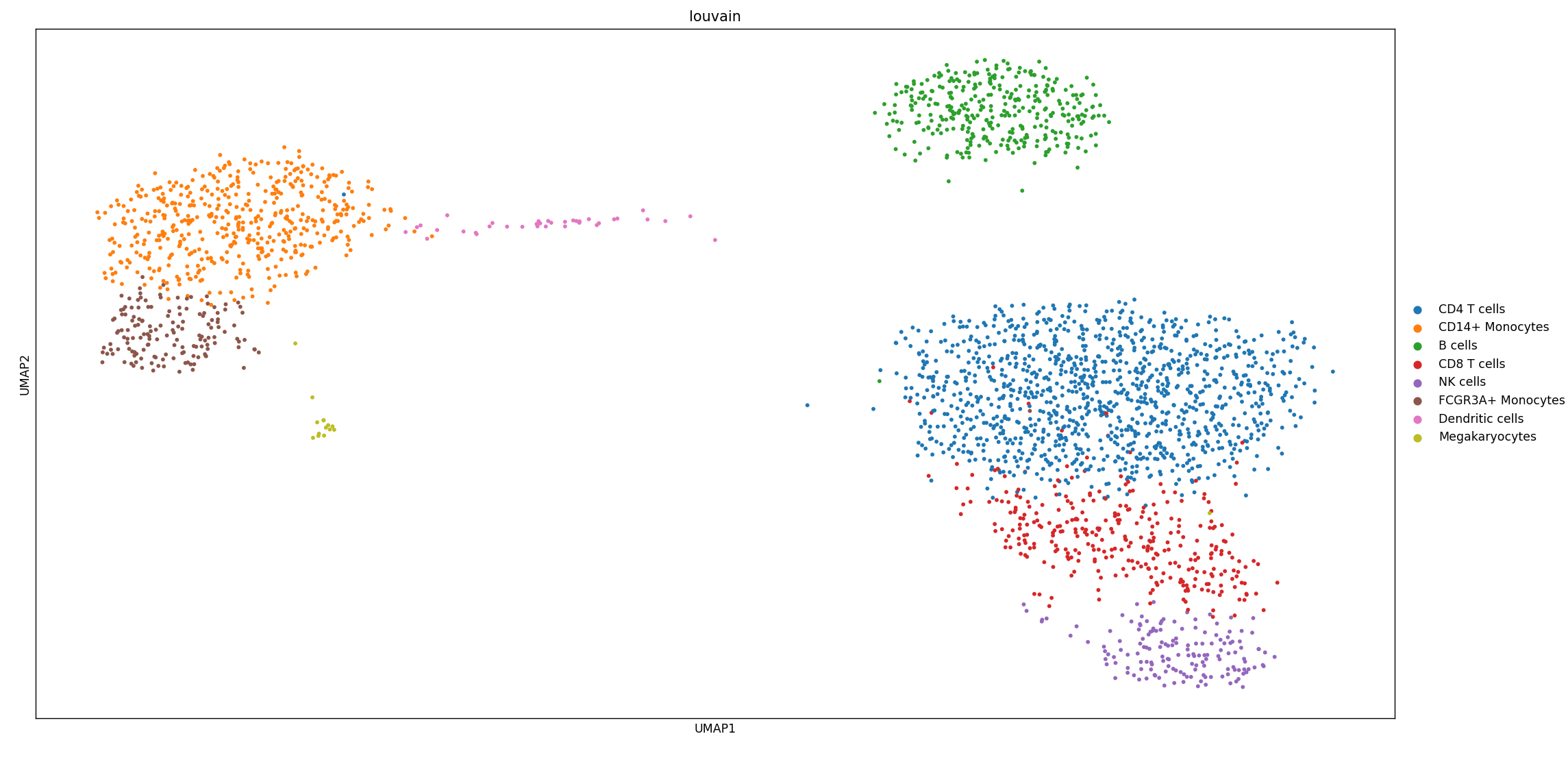
sc.pl.umap(adata_ref, color='louvain')
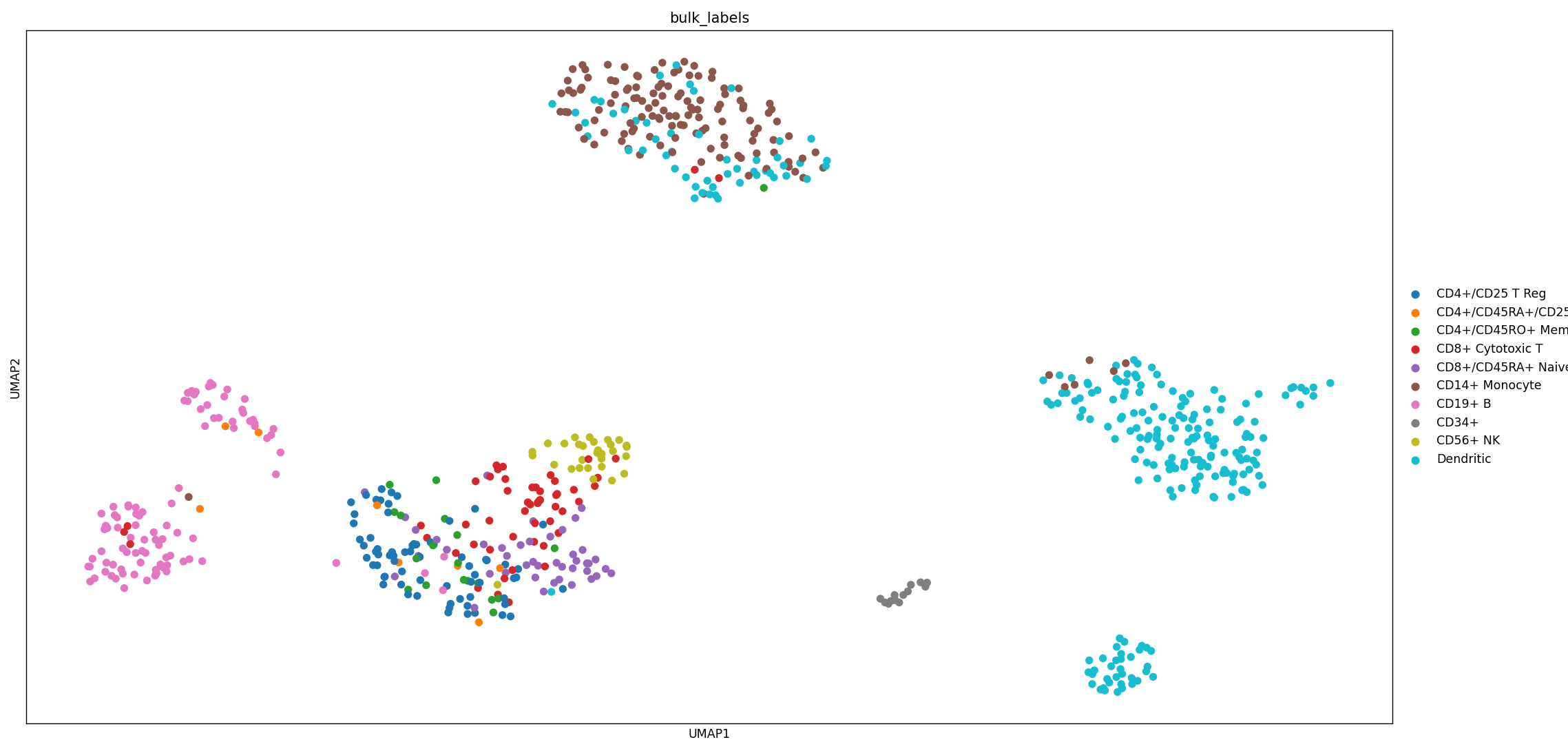
sc.pl.umap(adata, color='bulk_labels') # obs: 'bulk_labels',
adata_ref 数据可视化
adata 数据可视化
adata_ref.obs和adata.obs
# adata_ref.obs
n_genes percent_mito n_counts louvain
index
AAACATACAACCAC-1 781 0.030178 2419.0 CD4 T cells
AAACATTGAGCTAC-1 1352 0.037936 4903.0 B cells
AAACATTGATCAGC-1 1131 0.008897 3147.0 CD4 T cells
AAACCGTGCTTCCG-1 960 0.017431 2639.0 CD14+ Monocytes
AAACCGTGTATGCG-1 522 0.012245 980.0 NK cells
... ... ... ... ...
TTTCGAACTCTCAT-1 1155 0.021104 3459.0 CD14+ Monocytes
TTTCTACTGAGGCA-1 1227 0.009294 3443.0 B cells
TTTCTACTTCCTCG-1 622 0.021971 1684.0 B cells
TTTGCATGAGAGGC-1 454 0.020548 1022.0 B cells
TTTGCATGCCTCAC-1 724 0.008065 1984.0 CD4 T cells
# adata.obs
bulk_labels n_genes percent_mito n_counts S_score G2M_score phase louvain
index
AAAGCCTGGCTAAC-1 CD14+ Monocyte 1003 0.023856 2557.0 -0.119160 -0.816889 G1 1
AAATTCGATGCACA-1 Dendritic 1080 0.027458 2695.0 0.067026 -0.889498 S 1
AACACGTGGTCTTT-1 CD56+ NK 1228 0.016819 3389.0 -0.147977 -0.941749 G1 3
AAGTGCACGTGCTA-1 CD4+/CD25 T Reg 1007 0.011797 2204.0 0.065216 1.469291 G2M 9
ACACGAACGGAGTG-1 Dendritic 1178 0.017277 3878.0 -0.122974 -0.868185 G1 2
... ... ... ... ... ... ... ... ...
TGGCACCTCCAACA-8 Dendritic 1166 0.008840 3733.0 -0.124456 -0.867484 G1 2
TGTGAGTGCTTTAC-8 Dendritic 1014 0.022068 2311.0 -0.298056 -0.649070 G1 1
TGTTACTGGCGATT-8 CD4+/CD25 T Reg 1079 0.012821 3354.0 0.216895 -0.527338 S 0
TTCAGTACCGGGAA-8 CD19+ B 1030 0.014169 2823.0 0.139054 -0.981590 S 4
TTGAGGTGGAGAGC-8 Dendritic 1552 0.010886 4685.0 -0.148449 -0.674752 G1 2
# adata_ref.obs_names
Index(['AAACATACAACCAC-1', 'AAACATTGAGCTAC-1', 'AAACATTGATCAGC-1',
'AAACCGTGCTTCCG-1', 'AAACCGTGTATGCG-1', 'AAACGCACTGGTAC-1',
'AAACGCTGACCAGT-1', 'AAACGCTGGTTCTT-1', 'AAACGCTGTAGCCA-1',
'AAACGCTGTTTCTG-1',
...
'TTTCAGTGTCACGA-1', 'TTTCAGTGTCTATC-1', 'TTTCAGTGTGCAGT-1',
'TTTCCAGAGGTGAG-1', 'TTTCGAACACCTGA-1', 'TTTCGAACTCTCAT-1',
'TTTCTACTGAGGCA-1', 'TTTCTACTTCCTCG-1', 'TTTGCATGAGAGGC-1',
'TTTGCATGCCTCAC-1'],
dtype='object', name='index', length=2638)
# var_names
Index(['TNFRSF4', 'SRM', 'TNFRSF1B', 'EFHD2', 'C1QA', 'C1QB', 'STMN1',
'MARCKSL1', 'SMAP2', 'PRDX1',
...
'EIF3D', 'LGALS2', 'ADSL', 'TTC38', 'TYMP', 'ATP5O', 'TTC3', 'SUMO3',
'S100B', 'PRMT2'],
dtype='object', name='index', length=208)
也可以看到数据本身还有其他的:

以下信息放置到文章上方,本实例并没有采用。
sc.settings.verbosity = 1 # verbosity: errors (0), warnings (1), info (2), hints (3)
sc.logging.print_versions()
sc.settings.set_figure_params(dpi=80, frameon=False, figsize=(8</




![[消息队列 Kafka] Kafka 架构组件及其特性(二)Producer原理](https://img-blog.csdnimg.cn/direct/9ff2841fead74eefb58df0111529e074.png)