文献介绍

「文献题目」 Viv: multiscale visualization of high-resolution multiplexed bioimaging data on the web
「研究团队」 Nils Gehlenborg(美国哈佛医学院)
「发表时间」 2022-05-11
「发表期刊」 Nature Methods
「影响因子」 47.9
「DOI」 10.1038/s41592-022-01482-7
摘要
高度多重成像技术的进步使得能够对健康和患病状态下的复杂组织进行全面分析,从而促进了在空间分辨、亚细胞分辨率下对基础生物学和人类疾病的研究。尽管生物成像的快速创新带来了重大的科学价值,但没有统一的可交互操作标准的技术,限制了结果的分析和共享。采用社区设计的下一代文件格式 (next-generation file formats, NGFF) 是促进大规模生物成像交互操作性的拟解决方案。本文作者介绍了 Viv(https://github.com/hms-dbmi/viv),一个开源生物成像可视化库,直接在 web 上支持 OME-TIFF 和 OME-NGFF。Viv 通过消除对服务器端渲染的依赖,解决了大多数基于 web 的生物成像查看器的一个关键限制,提供了一个灵活的工具包,无需安装软件即可在移动和桌面设备上浏览多 TB 数据集。
研究结果
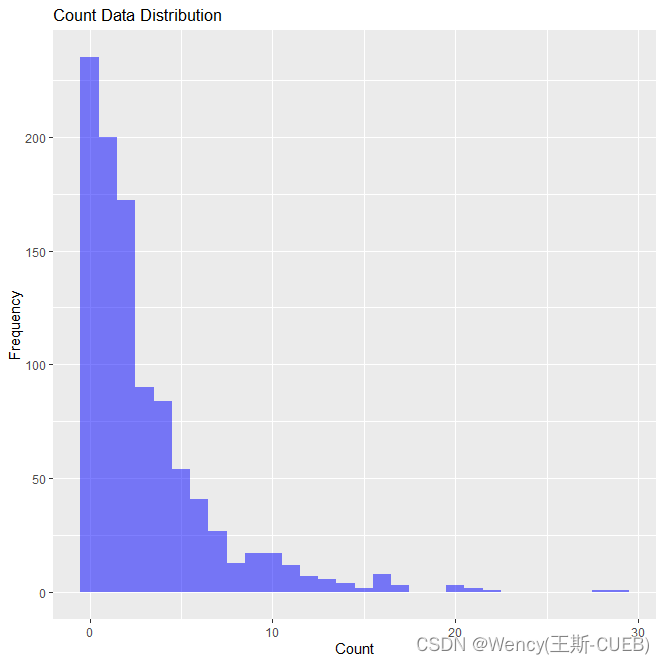
Viv 的功能与流行的桌面生物成像应用程序更相似,不同于相应的 web 替代品(Supplementary Note 1)。大多数 Web viewers 需要将大型二进制数据文件预先转换为渲染的图像(PNG 或 JPEG),以便在浏览器客户端中显示。两种现有方法执行此步骤(服务器端渲染),但在渲染发生时间以及一次转换多少二进制数据方面有所不同(Fig. 1)。离线选项在将应用程序部署给用户之前执行所有渲染,这意味着所有通道分组和数据转换都是固定的,无法通过用户界面进行调整。在线选项支持按需、用户定义的渲染,但在探索数据转换时会引入延迟,并且需要主动维护复杂的服务器基础设施。这两种方法都没有提供灵活的解决方案来直接查看大型公共数据存储库中以开放格式保存的数据集,并且服务器端渲染引入的瞬态数据表示抑制了与其他可视化和分析软件的互操作性。
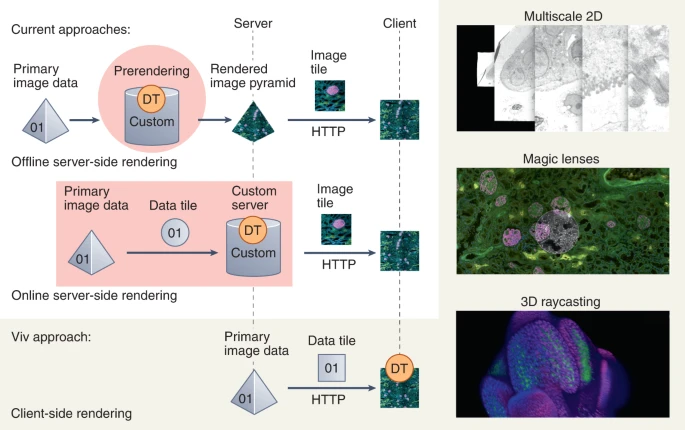
DT 表示数据转换为图像的位置。右栏显示 Viv 灵活的客户端渲染的子集,包括 multiscale 2D pyramids、magic lenses、3D volumes via raycasting。
Viv 实现纯粹的客户端渲染,将浏览器与服务器解耦,同时仍然提供按需多通道渲染的灵活性。现有的 Web viewers 也利用图形处理单元 (GPU) 加速渲染,但通常针对单通道体积数据集进行定制,最重要的是,缺乏为现有或新颖应用程序重用和组合功能的能力。相比之下,Viv 的模块化允许重新调整和扩展核心功能。该库由两个主要组件组成:(i) OME-TIFF 和 OME-NGFF 的数据加载模块,以及 (ii) 可在用户设备上呈现全位深度主要数据的可配置 GPU 程序。
Viv 的数据加载组件负责通过 HTTP 从二进制文件中获取和解码压缩通道数据的原子“chunks”。Viv 以 OME-TIFF 和 OME-NGFF 为目标,以满足生物成像界的多样化需求,并促进可交互操作的开放标准的使用。尽管 OME-TIFF 更为普遍,但其二进制布局和 metadata 模型限制太大,无法有效表示大容量和高维数据集。OME-NGFF 旨在解决这些限制,并且由于底层的多维 Zarr 格式 (https://doi.org/10.5281/zenodo.3773450),可以从云本地访问。Zarr 提供对各个 chunks 的直接访问,而 TIFF 需要寻求从不同平面访问 chunks,在读取具有大 c、t 或 z 维度的数据集时会引入较长的延迟。与基因组学中流行的方法类似,作者建议对 OME-TIFF 建立索引以改善随机 chunks 检索时间。作者的方法 Indexed OME-TIFF 提高了读取效率,并作为 OME-TIFF 的可选扩展实现 (https://doi.org/10.6084/m9.figshare.19416344)。
Viv 的图像层协调获取数据 chunks 和 GPU 加速渲染。所有渲染都发生在客户端 GPU 上,允许连续即时更新颜色映射、不透明度、通道可见性和仿射坐标变换等属性,而无需额外的数据传输。库用户可以使用自定义 WebGL 着色器修改渲染,这是 Viv 用来实现应用本地数据转换的 'magic lenses' 的功能:例如,重新缩放亮度或过滤特定通道。
由于所有渲染都转移到浏览器,Viv 不像以前的 Web viewers 那样依赖服务器来获取高度复用和多尺度的图像,从而使其灵活且能够嵌入到各种应用程序中 (https://doi.org/10.6084/m9.figshare.19416401)。作者通过 Imjoy 将 Viv 集成到 Jupyter Notebooks (https://github.com/hms-dbmi/vizarr) 中,以实现远程人机交互多模式图像配准工作流程和直接浏览 OME-NGFF 图像。Viv 也是 Vitessce 单细胞可视化框架成像组件的基础,展示了其与包括成像模式在内的其他模块化工具集成并为其提供动力的能力。最后,作者开发了 Avivator (http://avivator.gehlenborglab.org), 这是一个独立的图像查看器,展示了 Viv 丰富的功能集。
凭借其可扩展性和最低的部署要求,Viv 提供了一个新颖的工具包,可用于构建各种可重复使用的生物成像应用程序。它最适合显示高度多重和多尺度数据集的 2D 可视化,但也支持通过光线投射进行 3D 可视化。作者并不期望 Viv 取代现有的基于 web 的查看器,而是引导新一代基于 web 的可视化和分析工具来补充同样围绕可交互操作标准构建的桌面软件。
注:本文为个人学习笔记,仅供大家参考学习,不得用于任何商业目的。如有侵权,请联系作者删除。

本文由 mdnice 多平台发布