近来年,网状 Meta 分析相关研究不断涌现,此类研究不但能发表在国内各大核心期刊上,还能在SCI期刊甚至医学4大刊上看到其身影。随手在pubmed上面一搜索,就能得到一万多篇相关文献。俨然成为医学文献研究的“大杀器”!
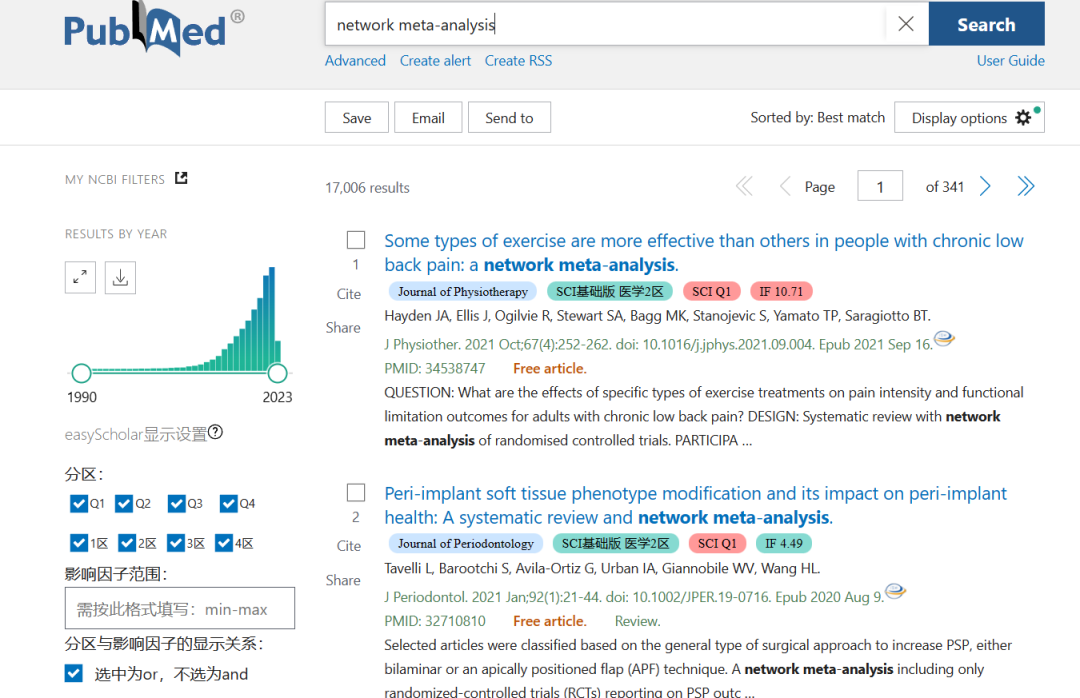
PubMed网状 Meta 分析检索截图
本文就网状 Meta 分析的概述、用途及操作步骤(R语言)方面进行了介绍。篇幅会比较长,内容较多,请多点耐心噢。
1、Meta分析简介
Meta分析(Meta-analysis)常译为汇总分析或荟萃分析,是系统评价(systematic review,SR)中经常用到的统计方法,是运用恰当的统计方法对若干个研究的结果数据进行合并,在医学上常常用于比较干预措施的有效性和安全性。
2、常规Meta分析的不足
常规的meta分析通常只能处理直接比较,比如下图之中的文献评价了虚拟现实(VR)技术对乳腺癌患者术后上肢功能康复的影响,其纳入的5项RCT(随机对照试验)均为两组设计的研究,都是VR干预与常规干预的比较。

知网截图
3、网状 Meta 分析
但是,如果说想比较干预A与干预B的效果,但仅有比较A与C,B与C的研究,那么怎么办呢?此时就需要网状 Meta 分析了!
NMA是什么?
普通Meta分析是合并直接比较的结果,比如你想研究A药和B药对C疾病的治疗效果,如果有A药和B药直接对比的临床研究,那么你就可以做普通的Meta分析,直接合并这些临床研究,得到A药和B药的对比结果。
但是很多时候,会缺少A药和B药直接对比的临床研究,但是有A药和C药直接对比以及B药和C药直接对比的临床研究,这样C药就是共同对照,以C药为中介,我们可以得到A药和B药间接比较的结果。

知网截图
上面的文献运用了网状 Meta 分析评价了10 种非药物干预方法在减少孕产妇分娩恐惧方面的效果,并得出了干预效果排序。这就是网状Meta分析最大的特色。
RCT研究1:A药可让患者血压降低10mmHg,C药可让患者血压降低2mmHg,所以A药比C药能多降低8mmHg(dA-C)
RCT研究2:B药可让患者血压降低15mmHg,C药可让患者血压降低5mmHg,所以B药比C药能多降低10mmHg(dB-C)
C是A药和B药的共同对照,dA-C-dB-C=-2mmHg,所以B药相对于A药,患者平均血压能多降低2mmHg。这里举的是连续性变量,如果是二分类变量OR值就是取对数相减。
上面说的是间接比较的概念,有的人以为NMA就是间接比较,那就错了,实际上有的时候既有直接比较的文献,又有间接比较的文献,这时候NMA是综合直接比较和间接比较的结果。就如下面的图。
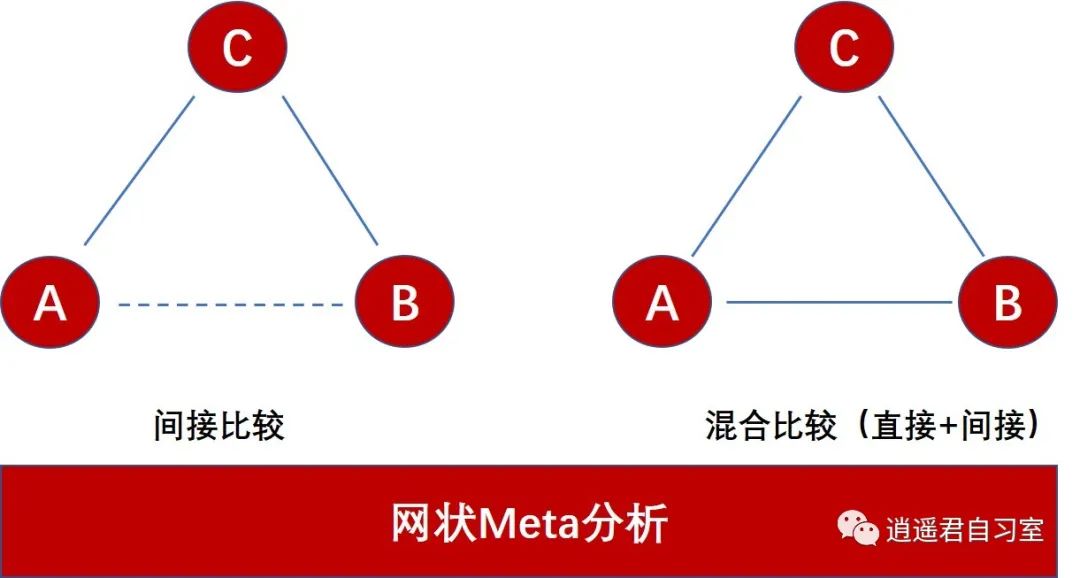
NMA的定义就是:基于多个研究分析两个以上干预措施之间间接比较结果或者直接比较结果与间接比较结果的合并结果(混合比较)的Meta分析。
那么什么情况下适合做NMA呢?
主要就是要满足三个基本假设:同质性假设、相似性假设和一致性假设
同质性假设:其实就是普通Meta分析中的异质性,原始研究之间临床特征、方法学、统计学没有异质性。
相似性假设:就是所有原始研究间及不同对照组间影响效应量的因素相似。大概意思就是A和C比较的原始研究要和B和C比较的原始研究之间是相似的
一致性假设:假如A和B之间的比较既有直接比较也有间接比较,那么直接比较的结果和间接比较的结果要一致。
那么NMA到底怎么做呢?
其实跟普通的Meta分析步骤区别并不是很大,只不过展示的结果会多一些,除了直接比较的内容,你还要展示间接比较的内容。
基本步骤:
1.选题
2.文献检索
3.数据提取
4.质量评价
5.统计分析(比普通Meta分析复杂)
6.报告结果(间接比较或混合比较)
4、贝叶斯meta分析
关于贝叶斯,内容实在太多,个人仅了解了一点皮毛,仅作简单描述,有兴趣的童鞋可以去看看相关统计教材。
meta分析方法主要包括频率学法和贝叶斯法,这是因为当前统计学可以分为两个主要学派:经典统计学与贝叶斯统计学派。
频率学方法基于概率的频率解释,将概率解释为大量重复试验后频率的稳定值,贝叶斯法认为某一事件的发生概率不是固定的,而是基于观察数据及一些条件来对先验概率进行修正,并对所有分析的干预措施进行排序。
5、适用场景
-
对多种干预措施进行比较
-
无直接比较的原始研究
-
有直接比较的原始研究,但数量或质量不理想
6、结果报告与解读
报告结果包括:网状关系图;一致性检测;构建模型;排序图;异质性分析;判断发表偏倚等。这里以李腾等学者发表的论文《非药物干预减少分娩恐惧的网状 Meta 分析》为例,了解下网状Meta分析通常应输出的结果。
①网状关系图与一致性检验
网状关系图:图中的节点表示不同的干预措施,连线表示两种干预方法有直接比较,节点越大,表示该干预措施直接比较出现的次数越多(样本最大),形成的闭环表示既有直接比较又有间接比较。
一致性检验:是比较直接比较与混合比较两个模型的一致性,P>0.05表示一致性良好。
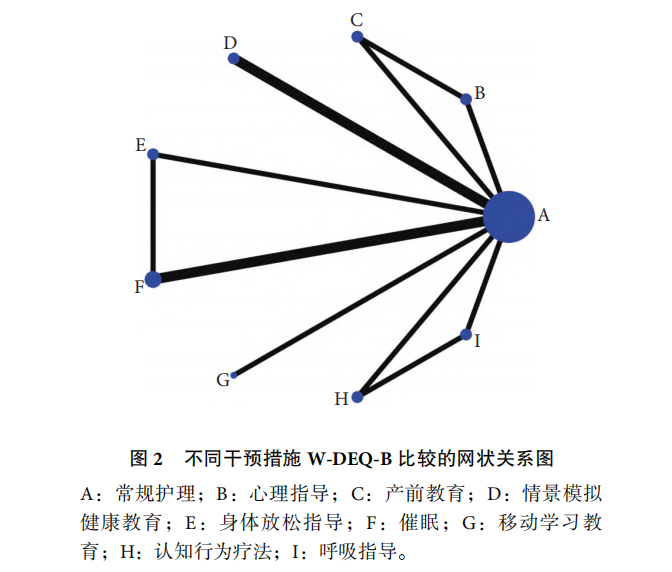
图片来源:《非药物干预减少分娩恐惧的网状 Meta 分析》
②排序图/表
排序图/表的形式多样,采用不同软件也会有差异。这里附上2份排序表的截图。
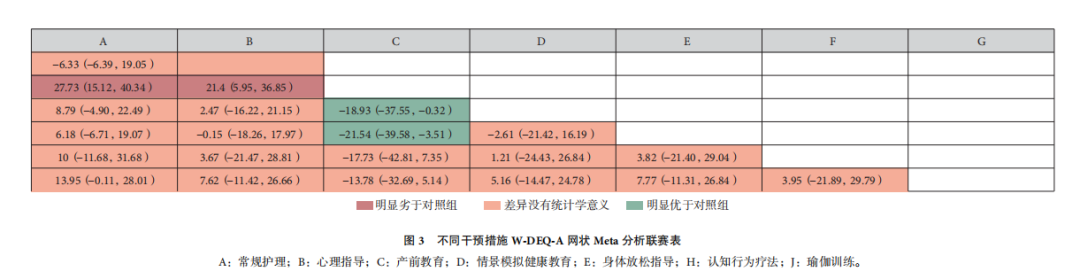
图片来源:《非药物干预减少分娩恐惧的网状 Meta 分析》,不同颜色代表不同效果
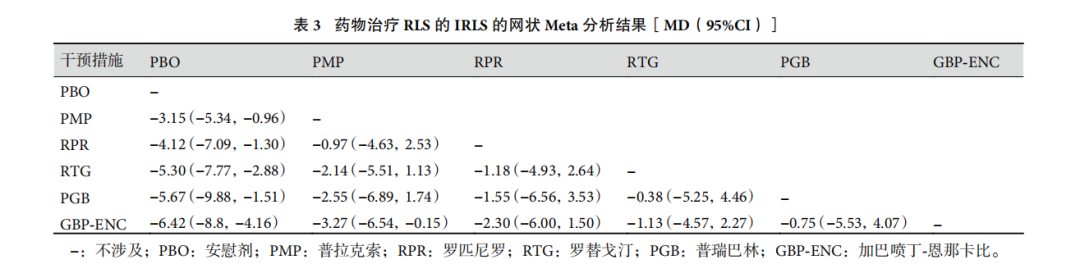
图片来源:《药物治疗不宁腿综合征有效性和安全性的网状 Meta 分析》,最下方的一行疗效最好
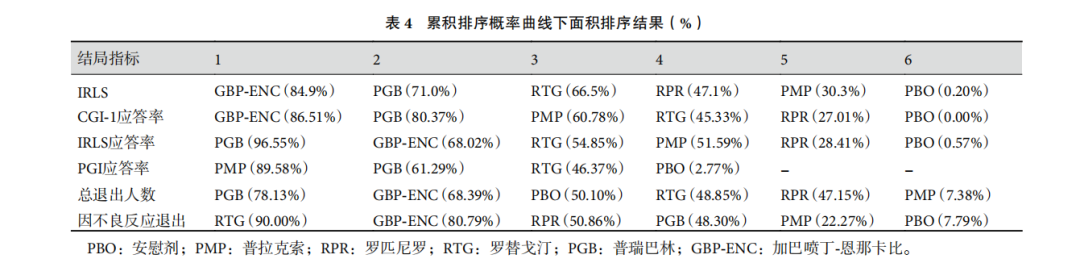
图片来源:《药物治疗不宁腿综合征有效性和安全性的网状 Meta 分析》,看整体排序,排序为1表示最好,2次之。
其余的结果报告与普通的meta分析类似,限于篇幅,这里就不一一描述了。
网状Meta分析实操——基于R语言的实现
Stata、R语言、WinBUGS是贝叶斯网状Meta分析应用最多的工具。Stata收费,这里以开源的R语言进行实操分享。以下的步骤参照了向虹等学者发表的论文《应用R语言BUGSnet程序包实现贝叶斯网状Meta分析》,个人把此论文的代码跑了一遍,然后加了一些便于理解的注释,顺便分享了一些可能会碰到的问题(可能踩的坑)。
文章后面附上了我自己录入的数据和代码文件的获取方式。代码经个人测试,与论文的结果保持一致。下面是详细的步骤。
一 、准备数据
数据来源于一项关于类固醇佐剂治疗天疱疮的网状Meta分析研究,该研究纳入 10 个试验,纳入了592 例患者,评估了 7 种方法治疗天疱疮的效果,见下表。
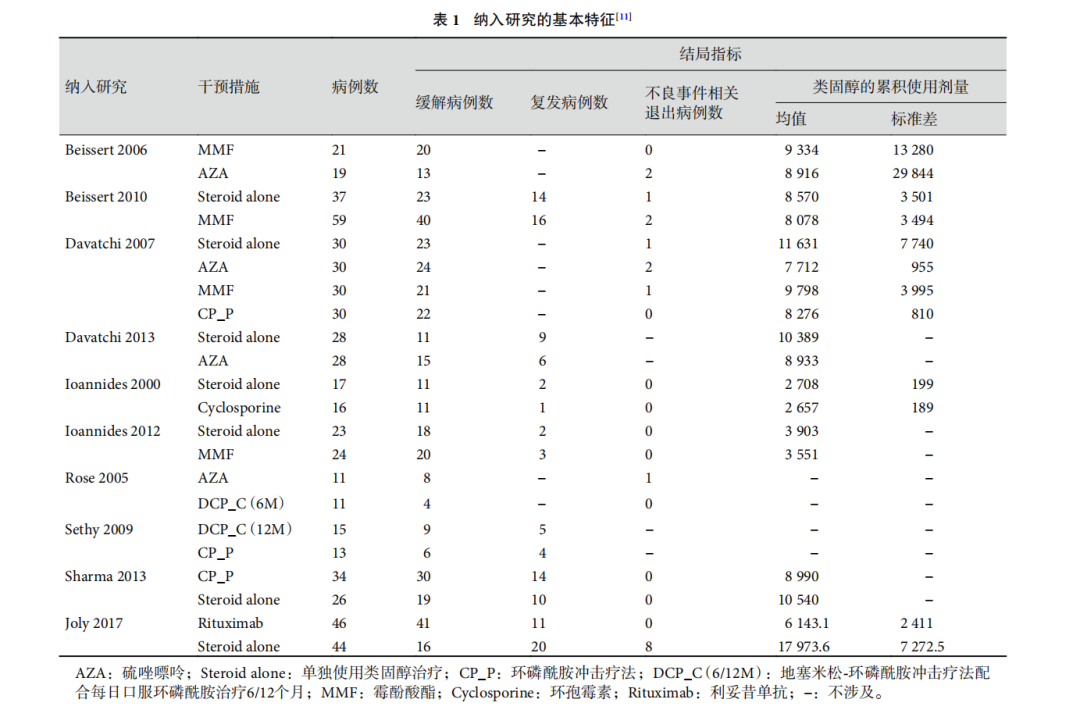
图片来源于论文《应用R语言BUGSnet程序包实现贝叶斯网状Meta分析》
这里有个坑:按照这个表格录入数据会有变量名不匹配的问题,要求是英文,且为了与参照的论文中的代码保持一致,还是要注意下。PS:其实自己随便取个英文名也是可以的,只是说这样的话后续的代码不能直接用了,要做针对性地调整。
以下是我手动录入的表格数据,建议严格按照下图的变量名(即表格中的列名)进行设置,最后一行因截屏问题没有显示出来,大家录入的时候补上就好。
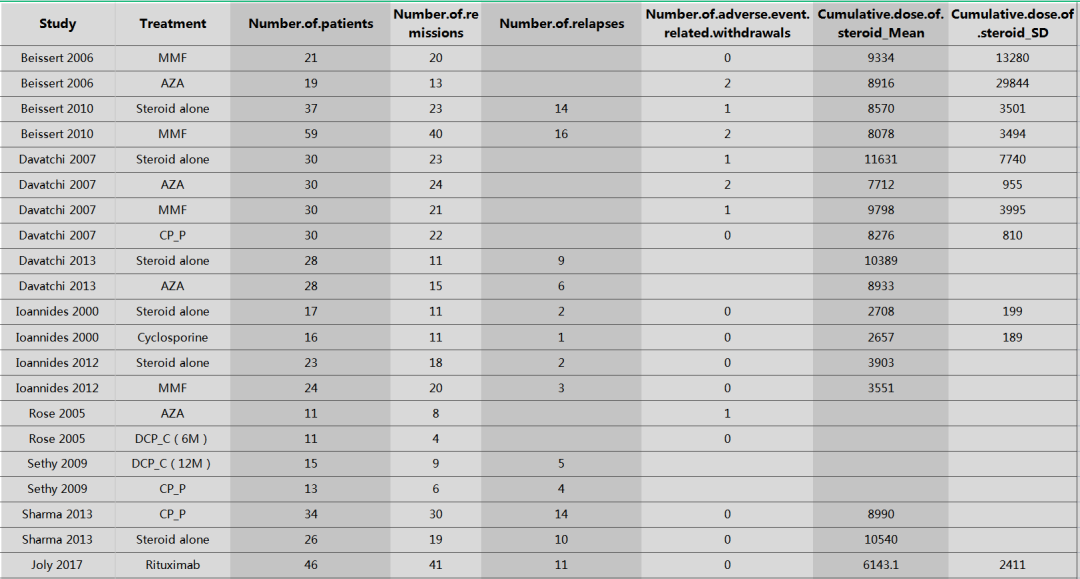
数据文件截图
变量名我给附上来了,练习时建议保持一致,可以直接copy。
| Study | Treatment | Number.of.patients | Number.of.remissions | Number.of.relapses | Number.of.adverse.event.related.withdrawals | Cumulative.dose.of.steroid_Mean | Cumulative.dose.of.steroid_SD |
变量名
二、安装软件和包
如果是第一次接触R语言,请看下面的安装过程,依次安装3个软件,如果已经有基础或已安装,建议跳过。
-
R语言:我用的R语言版本为当前的最新版R4.2.2,直接到官网下载:https://www.r-project.org。下载后默认设置进行安装即可,也可以手动改下安装的路径,不熟悉的可以去百度一下具体方法。
-
R Studio:R Studio是R语言的IDE,很好用,我们基本就是用它来写R代码,直接到官网下载:https://www.rstudio.com/products/rstudio/)。下载完安装。
-
JAGS-4.3.0 :R语言BUGSnet包实际就是来调用JAGS软件来实现贝叶斯网状meta分析的,为何不直接用JAGS?那是因为R语言可以提供很漂亮的绘图!这个软件下载很慢,文末有软件获取方法(这里特别感谢小师妹分享的JAGS软件包)。
成功安装后,打开R Studio软件,萌新可以先百度学一下基本的使用方法。这里,建议大家做一些必要的设置:
1、将镜像源更改为国内:
步骤:Tools -> GlobalOptions->Options -> Packages ,单击change可修改成chinakaitou镜像源,这个操作可以极大地提升安装包的速度。
2、设置code编码:
Tools -> Global Options->Default-> code -> Saving text -> encoding框,单击change,将相关设置修改为UTF-8。有编程基础的童鞋应该都清楚,utf-8是国际通用的编码,可以避免换一台电脑打开就乱码的困扰。
-
在R Studio中安装一些包
注意:#是注释的意思,是给我们看的说明文字,不会被执行。代码如下。
#安装 Rtoolinstall.packages("pkgbuild")pkgbuild::has_build_tools()#安装BUGSnet包install.packages(c("remotes", "knitr")) #为了实现下一步在github中安装包remotes::install_github("audrey-b/BUGSnet@v1.1.0",upgrade = TRUE,build_vignettes = TRUE,dependencies = TRUE) #从github中安装BUGSnet 1.1.0#没有安装下面这些包的请执行下面代码进行安装install.packages(c("xlsx", "dplyr", "tidyr"))#以上是原文的代码,目的是为了导入excel格式的文档,#由于我这里安装时报错,为了简单,最后换用了基础包下的read.csv(无须安装)
三 、网状meta分析具体实现过程
1、录入、整理数据
library(dplyr)#导入dplyr包library(tidyr)#导入tidyr包;目的是实现管道操作及一系列tidy风格的函数data_total<-read.csv("meta.csv", 1) #导入编辑好的数据文件,csv格式,meta.csv是文件名称str(data_total) #查看导入的数据信息data_remission<- data_total[, c(1:4)] #拿到第1~4列,并命名为data_remissiondata_relapse<-data_total[, c(1:3, 5)] #拿到第1~3列和第5列,并重命名data_withdrawal<- data_total[, c(1:3, 6)]#拿到第1~3列和第6列,并重命名data_dose<-data_total[, c(1:3, 7:8)]#拿到第1~3列和第7~8列,并重命名#下面代码重命名结局指标#data_remission代表缓解病例数据,为主要疗效指标data_remission<-data_remission %>% rename(sampleSize=Number.of.patients, events=Number.of.remissions) #将data_remission内的变量重命名,然后将整个数据依然命名为data_remission# data_relapse代表复发病例数据,为次要疗效指标data_relapse<-data_relapse %>% drop_na() %>%rename(sampleSize=Number.of.patients, events=Number.of.relapses) #重命名# data_withdrawal代表不良事件相关退出病例数据,为次要安全性指标data_withdrawal<-data_withdrawal %>%drop_na() %>% rename(sampleSize= Number.of.patients,events=Number.of.adverse.event.related.withdrawals)#重命名#data_dose代表固醇的累积使用剂量,为主要安全性指标data_dose<-data_dose %>% drop_na() %>%rename(sampleSize=Number.of.patients, mean=Cumulative.dose.of.steroid_Mean, SD=Cumulative.dose.of.steroid_SD)#重命名
2、绘制网络关系图
library(BUGSnet) #导入BUGSnet包#预处理,指定目标数据及变量名data_remission <- data.prep(arm.data = data_remission, varname.t = "Treatment", varname.s ="Study")data_dose <- data.prep(arm.data = data_dose,varname.t = "Treatment", varname.s = "Study")#绘制描述性统计图(网络关系图)par(mfrow = c(1,2)) #在同一张图上显示1x2个图net.plot(data_remission, node.colour = "darkgrey") #绘制散点图,连线颜色为darkgreynet.plot(data_dose, node.colour = "darkgrey")
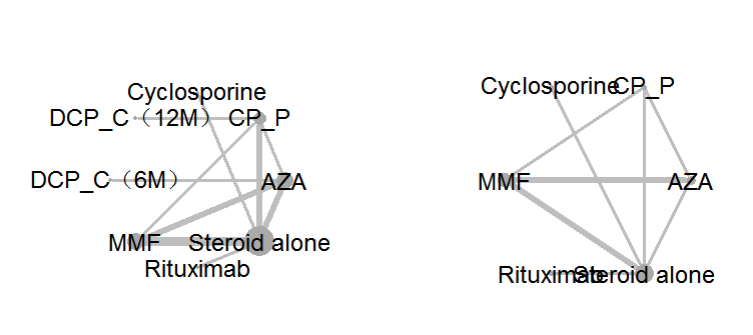
输出的网络关系图
3、设置模型类型
#使用nma.model函数设置拟合哪种模型(固定效应模型或随机效应模型)#设置固定效应模型fixed_effects_model_remission <-nma.model(data_remission, outcome="events", N="sampleSize",reference="Steroid alone", family="binomial",link="logit",effects="fixed")fixed_effects_model_dose <- nma.model(data=data_dose, outcome="mean",N="sampleSize",sd="SD", reference="Steroid alone",family="normal",link="identity", effects="fixed")#nma.model参数解释#outcome指定结局变量# reference设置对照组,本例为Steroid alone(单独使用类固醇治疗)# family用于指定结果的分布类型binomial(二分类资料),normal(计量资料),poisson(计数资料);# link用于指定NMA模型使用的函数,logit用于OR,log用于RR或计数数据的比率(Rate Ratio);# cloglog用于二分类数据危险比(HR);identity用于计量资料;# effects用于设置效应模型的类型。#设置随机效应模型random_effects_model_remission <- nma.model(data_remission, outcome="events", N="sampleSize",reference="Steroid alone", family="binomial",link="logit", effects="random")random_effects_model_dose <- nma.model(data=data_dose, outcome="mean", N="sampleSize",sd="SD", reference="Steroid alone", family="normal",link="identity", effects="random")
4、执行贝叶斯网状Meta分析
# 使用nma.run函数执行贝叶斯网状Meta分析set.seed(20222022) #随机数字生成器fixed_effects_results_remission <- nma.run(fixed_effects_model_remission, n.adapt=1000,n.burnin=1000, n.iter=10000)fixed_effects_results_dose <- nma.run(fixed_effects_model_dose,n.adapt=1000, n.burnin=1000,n.iter=10000)random_effects_results_remission <- nma.run(random_effects_model_remission, n.adapt=1000,n.burnin=1000, n.iter=10000)random_effects_results_dose <- nma.run(random_effects_model_dose, n.adapt=1000,n.burnin=1000, n.iter=10000)#评估效应模型,并输出杠杆图par(mfrow = c(2,2))nma.fit(fixed_effects_results_remission)nma.fit(fixed_effects_results_dose)nma.fit(random_effects_results_remission)nma.fit(random_effects_results_dose)
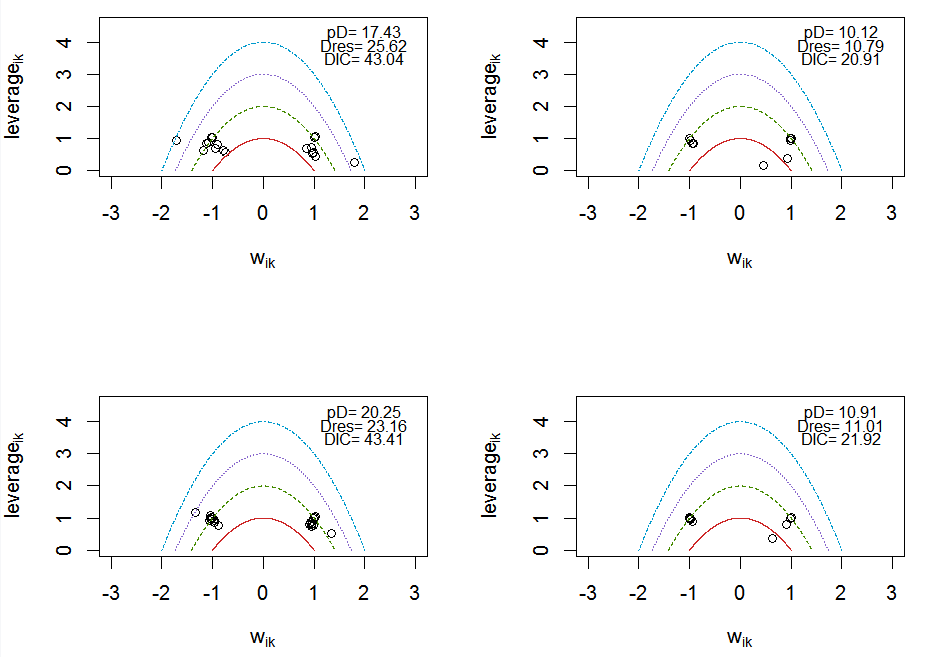
代码输出的图
①绘制排名图(SUCRA 和 rankogram )
#使用nma.rank函数 绘制治疗排名结果图,largerbetter表示更大更好sucra_out_remission<- nma.rank(random_effects_results_remission, largerbetter=TRUE)sucra_out_dose<- nma.rank(fixed_effects_results_dose, largerbetter=FALSE)# 使用ggplot2(绘图神奇)进行绘图install.packages('ggplot2') #安装包,如果已有请忽略library(ggplot2) #导入包#设置颜色z<-scale_colour_manual(values=c(AZA="#FF6600",MMF="#FF0088", "Steroid alone"="#00FF00", CP_P="#007700", Cyclosporine="#6633CC", "DCP_C(6M)"="#00CCFF", "DCP_C(12M)"="#FFFF00", Rituximab="#AA0000"))#绘制累积排序概率图下面积(SUCRA)s1<- sucra_out_remission$sucraplot+zs4<- sucra_out_dose$sucraplot+z#绘制排序概率图(rankogram)r1<- sucra_out_remission$rankogram+theme(axis.text.x = element_text(angle = 30,hjust = 1, vjust = 1))# theme参数是设置主题,包括文字等r4<- sucra_out_dose$rankogram+theme(axis.text.x = element_text(angle = 30, hjust = 1, vjust = 1))#输出图片install.packages('gridExtra')#安装包library(gridExtra) #导包grid.arrange(s1, s4, r1, r4, ncol = 2) #2x2
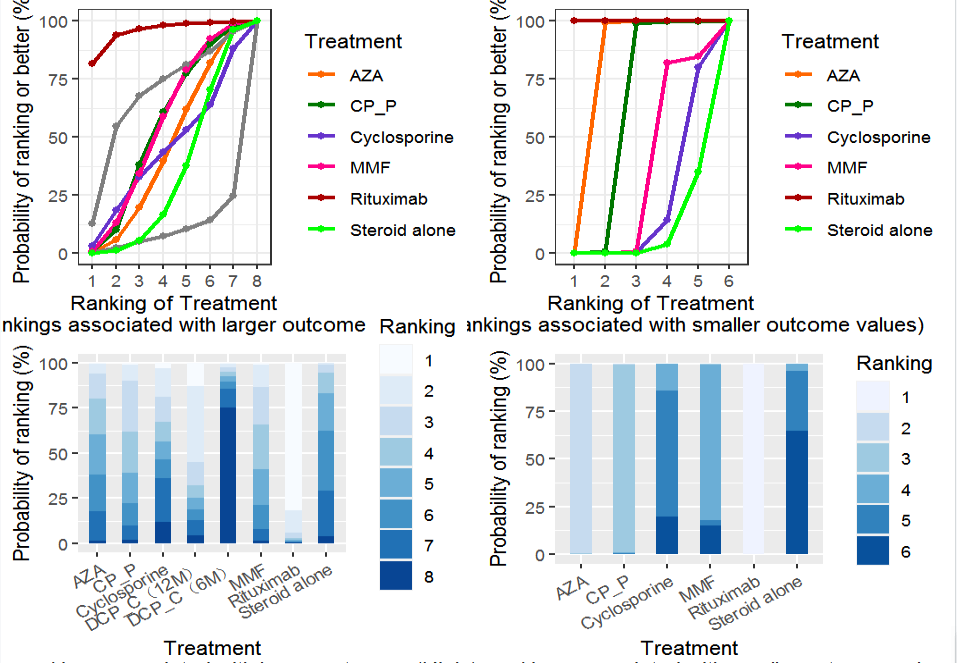
SUCRA 图和 rankogram 图,解读:前两幅图是越高越好,后两幅颜色越淡越好,总结:Rituximab疗效最好
②绘制排名表热图
# nma.league() 函数用于获得排名表热图league.out_remission<- nma.league(random_effects_results_remission, central.tdcy="median",order = sucra_out_remission$order, log.scale =TRUE,low.colour = "springgreen4", mid.colour = "white",high.colour = "red", digits = 2)#参数解释#central.tdcy用于设置统计数据,可以设置“mean”或“median”#order用来排序#log.scale=TRUE 表示用对数形式显示数据#colour用于设置颜色,本例低、中、高设置了3种不同颜色#digits设定小数点后的显示位数league.out_dose<- nma.league(fixed_effects_results_dose, central.tdcy="median", order =sucra_out_dose$order, log.scale =TRUE, low.colour ="springgreen4", mid.colour = "white", high.colour ="red", digits = 2)#绘制排名表热图l1<-league.out_remission$heatplotl4<-league.out_dose$heatplotgrid.arrange(l1, l4, ncol = 1) #图片放在一列上输出#可以将排名表热图作为表格输出league.out_remission$tableleague.out_remission$longtable

排名表热图,解读:不同颜色代表不同的效果
③ 绘制森林图
# 使用nma.forest() 函数绘制 森林图#参数解释与排名表热图一样f1<- nma.forest(random_effects_results_remission, central.tdcy="median", log.scale =TRUE,comparator = "Steroid alone")f4<- nma.forest(random_effects_results_remission,central.tdcy="median", log.scale =TRUE,comparator ="Steroid alone")grid.arrange(f1, f4, ncol = 2) #输出森林图
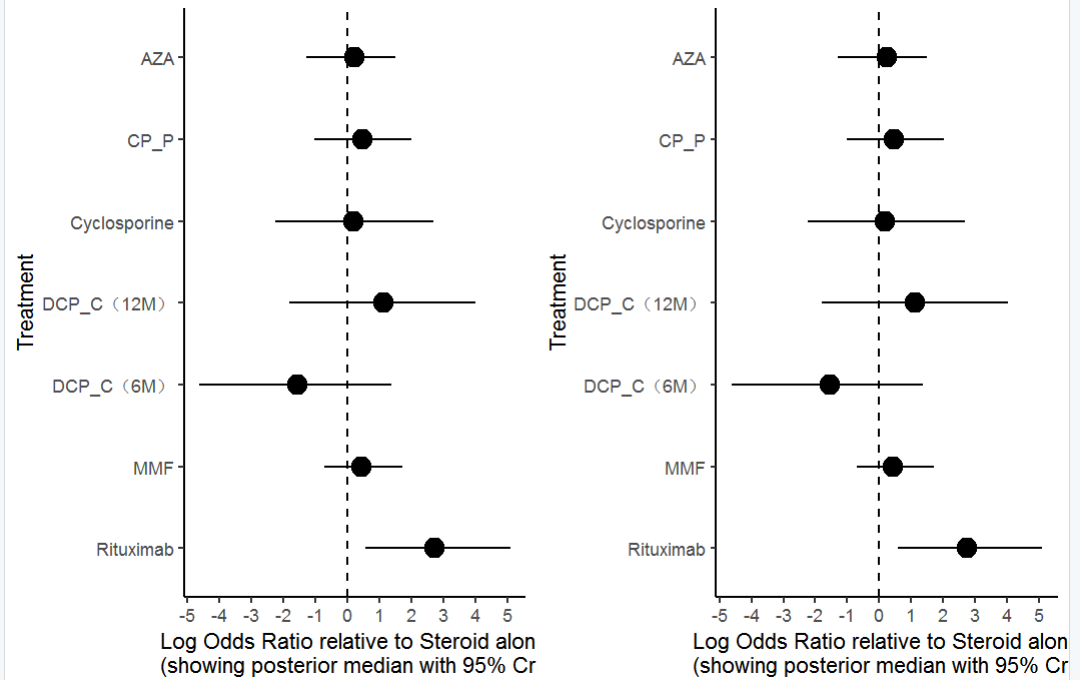
输出的森林图,解读:Rituximab是最有效的疾病缓解干预措施
关于实操部分的代码,是可以直接复制下来的,大家也可以按照推文中参考文献手动输入代码。如果觉得手动敲代码太累,可以关注本公众号,并回复关键词:网状meta,就可以直接获取数据文件及代码文件的链接。后续会继续更新有关内容
[1]向虹,岳磊,赵红,李华,谢天宏,杨婷,王杰,张宇昊,谢忠平.应用R语言BUGSnet程序包实现贝叶斯网状Meta分析[J].中国循证医学杂志,2022,22(05):600-608.
[2]张茜,陈项婷,周长青.药物治疗不宁腿综合征有效性和安全性的网状Meta分析[J].中国循证医学杂志,2022,22(08):908-916.
[3]李腾,张永爱,张海苗,曾娟.非药物干预减少分娩恐惧的网状Meta分析[J].中国循证医学杂志,2022,22(12):1410-1418.

![[leetcode]118.杨辉三角](https://img-blog.csdnimg.cn/img_convert/5026e7ea3d496340c73c39019792ebc3.png)