羊草(Leymus chinensis)是欧亚草原多年生优势草本植物,以其显著的适应性和饲料品质而闻名。尽管人们越来越认识到其生态和经济价值,但基因组序列的缺失及其遗传转化所面临的挑战限制了其在基础研究和野生植物改良方面的关键应用。
2023年10月24日,发表在PNAS(IF=11.1)上的文章“Genome evolution and initial breeding of the Triticeae grass Leymus chinensis dominating the Eurasian Steppe”,研究了羊草未开发的基因组序列和遗传育种,揭示了该物种的异源多倍体化和基因组进化。

技术方法概述
本研究采集了东北羊草草场中野生羊草,并从中选择具有强健根状茎的单株(Lc6-5)基于illumina、Pacbio以及HiC技术进行基因组组装,并进行了比较基因组分析。
主要结果
1、羊草基因组组装和注释
本研究对单株植物Lc6-5进行基因组组装,基因组大小约为7.85Gb,N50为318.49 Mb,BSUCO评估完整度为98.5%(表1)。重复序列共计6.89 Gb,占整个基因组的87.76%,长末端重复逆转录转座子(LTR逆转录转座子)构成了最丰富的重复类,占基因组的70.65%。
表1 羊草基因组的组装和注释
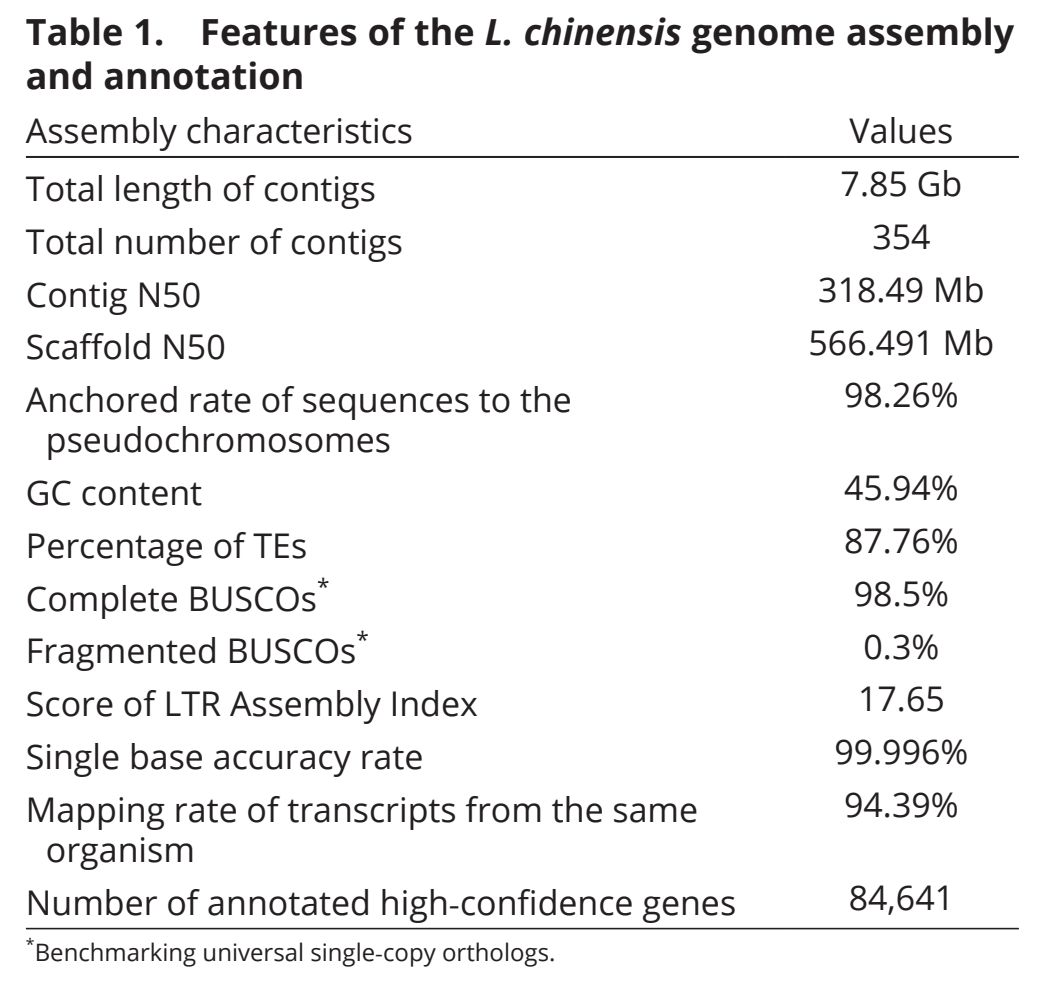
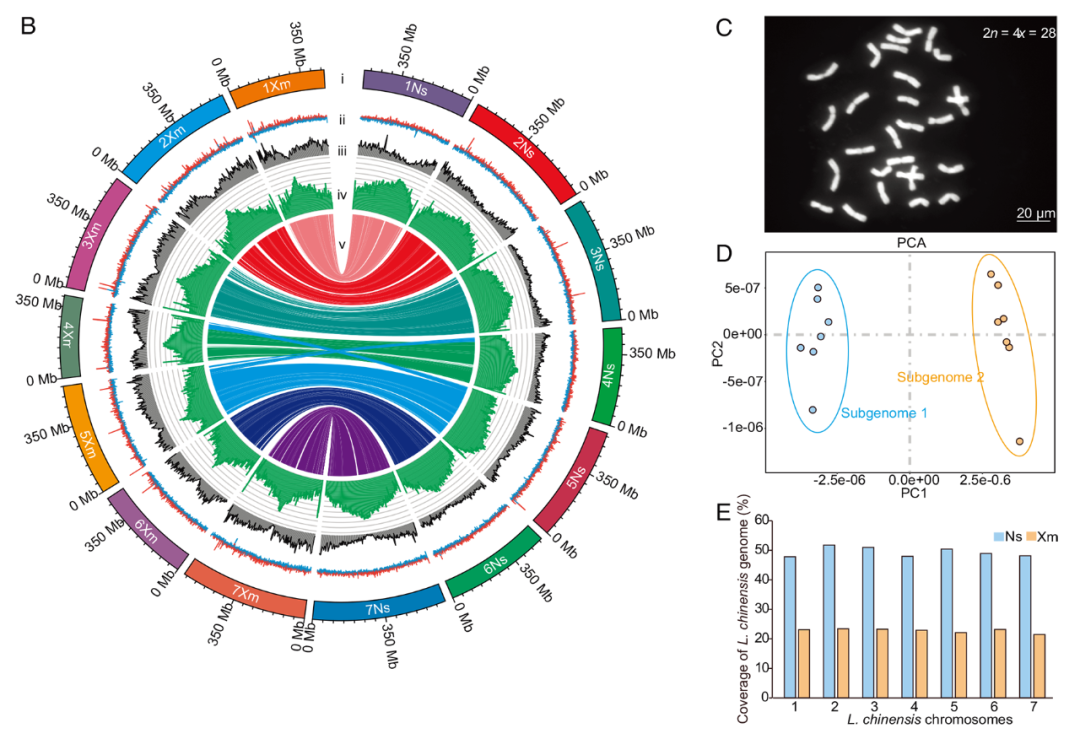
图1 羊草基因组图谱
2、羊草基因组亚型和比较基因组分析
本研究进一步对羊草基因组进行了单倍型组装,分成了2个亚基因组,Ns亚基因组和Xm亚基因组(图1D)。系统发育和分子年代测定研究表明,Ns亚基因组和Xm亚基因组在约~16.79MYA前分化(图2A),并在约4.18 MYA前发生多倍化从而形成异源四倍体羊草(图2A)。共线性分析表明,Ns亚基因组和Xm亚基因组在4号染色体和5号染色体上发生了易位,进一步与其他小麦族物种的共线性分析结果确认了羊草基因组的易位是发生在Ns亚基因组。此外,相较于Xm亚基因组,Ns亚基因组具有更高的基因密度和TE含量以及进化速度(图2E-G)。
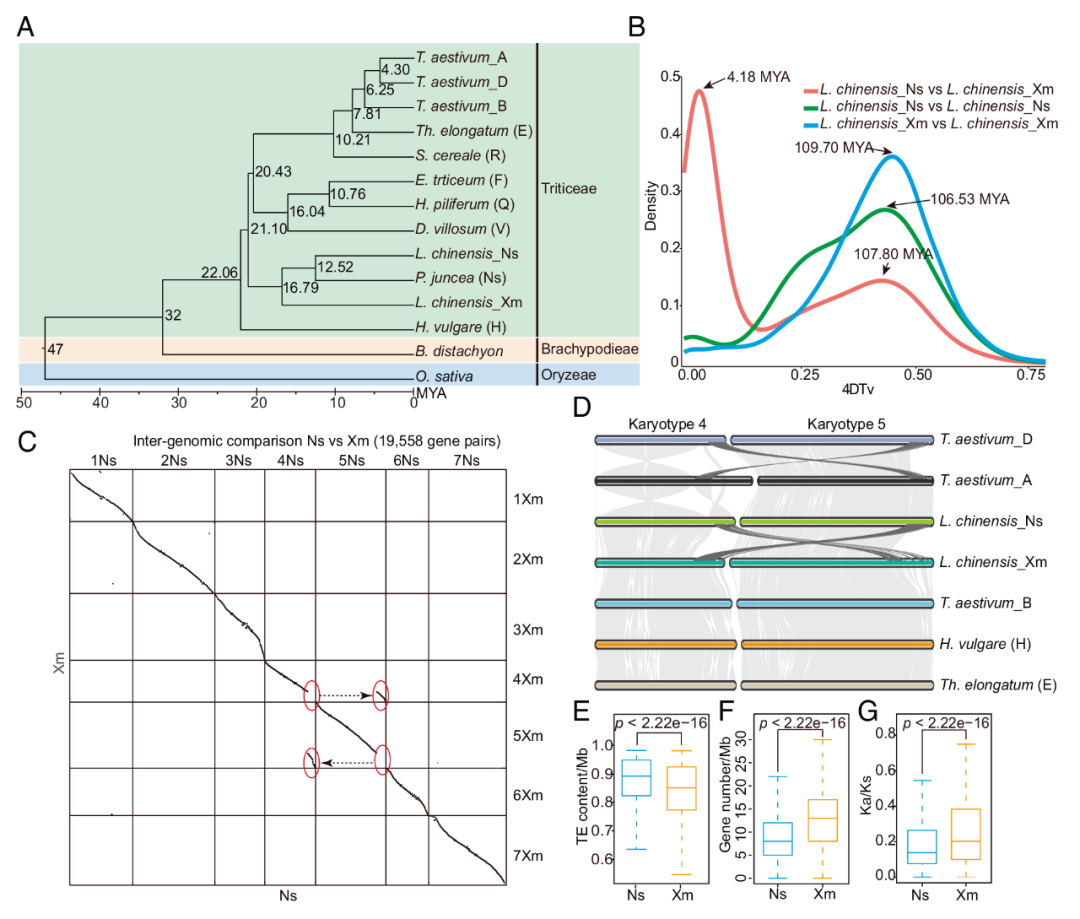
图2 羊草的基因组进化和四倍体化事件
3、转座子参与了基因组进化
LTR-retrotransposons(主要为Gypsy和Copia)是羊草基因组中最丰富的转座子,研究鉴定到了最近三次LTR插入,Gypsy插入在0.43 MYA和1.51–1.84 MYA呈双峰分布,Copia插入在0.65-0.72 MYA呈单峰分布(图3C),在两个亚基因组中都存在。本研究进一步分析了完整的LTR-RTs,旨在探讨Copia和Gypsy不同家族之间的进化关联,以及它们在两个亚基因组中的作用。根据基于逆转录酶结构域的系统发育树(图3D),Copia和Gypsy分别属于两个独立的超家族,其中Gypsy包含六个主要分支,Copia则有八个分支。此外,在亚基因组的比较中,作者发现Ns和Xm之间各家族的分布模式相似(图3E),这为它们基因组大小相近(Ns为3,998 Mb,Xm为3,715 Mb)提供了解释。
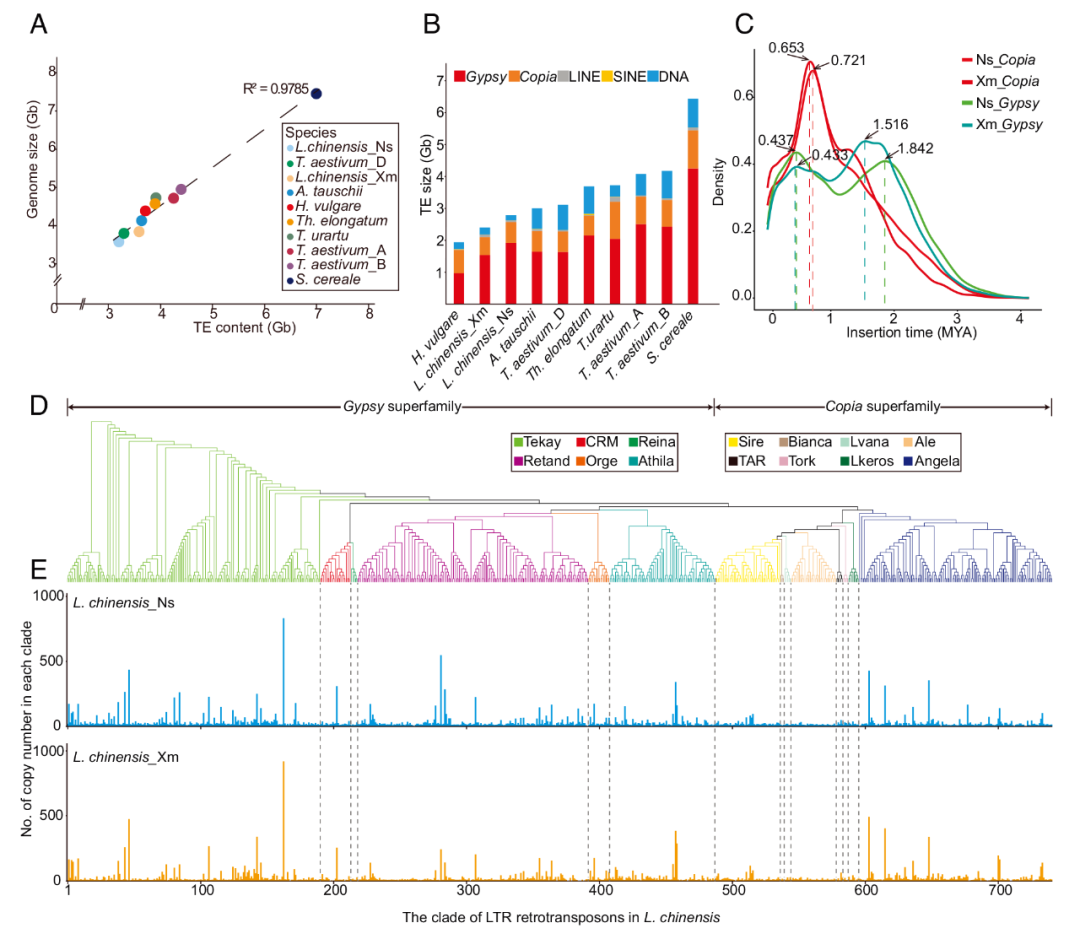
图3 羊草基因组中LTR逆转录转座子的扩增与进化分析
4、利用基因组编辑系统诱导羊草基因组的基因修饰
本研究建立了针对L. chinensis的遗传转化体系,选取了未成熟穗作为转化材料。通过对L. chinensis的叶、根、根茎和小穗进行小RNA测序,共鉴定出393个成熟的miRNA。其中,38个miRNA家族在miRBase的植物基因组中是保守的,而13个家族是单子叶植物特有的(图4B)。在L. chinensis的单子叶植物特有miRNA中,miR528在叶子中的表达最高(图4C),这表明它在这个物种中可能具有重要作用。研究人员还应用CRISPR/Cas9技术,成功获得了两个MIR528的敲除突变体。进一步通过转录组分析预测了MIR528的35个潜在靶基因,结果证实了miR528对L. chinensis的分蘖具有负调控作用。
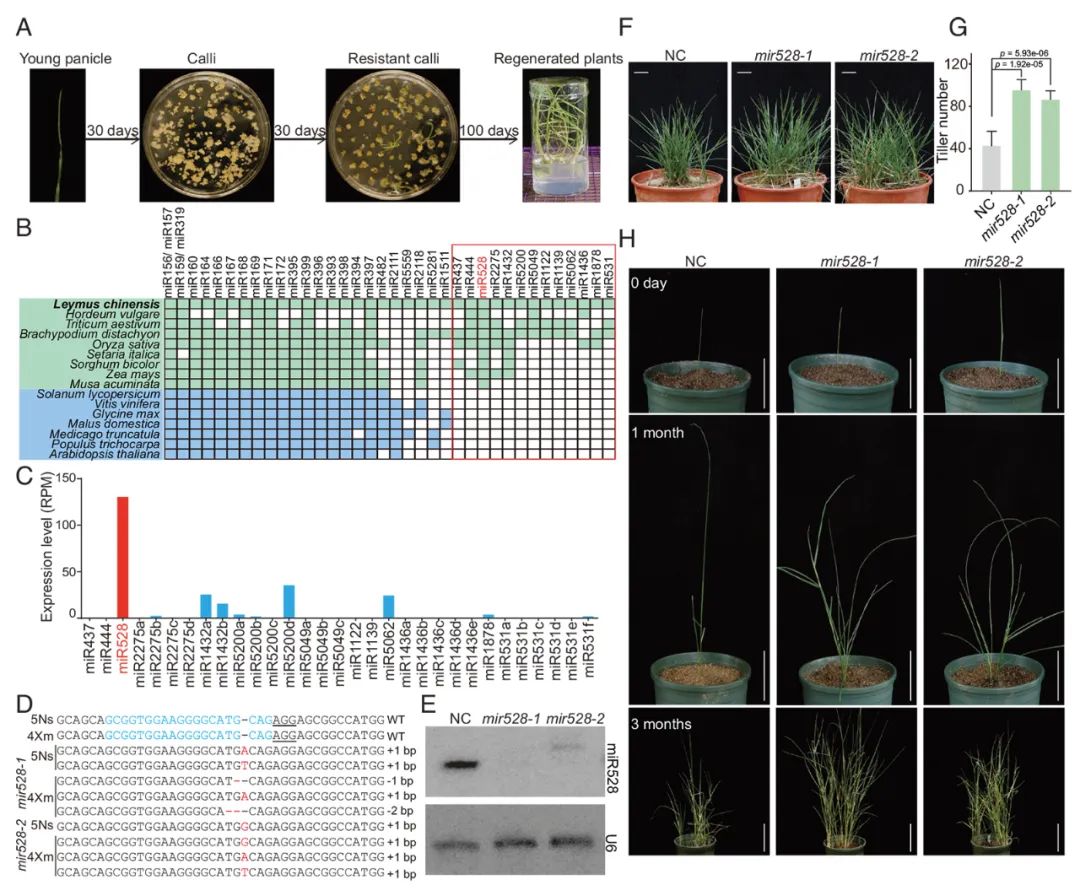
图4 利用基因组编辑系统诱导羊草基因组的基因修饰
总结
综上所述,本研究为小麦科植物的进化研究提供了宝贵的基因组资源,并提出了利用基因组信息和基因组编辑加速改良具有多倍体和自交不亲和等特征的野生羊草的概念框架。
参考文献
Genome evolution and initial breeding of the Triticeae grass Leymus chinensis dominating the Eurasian Steppe. PNAS, 2023.