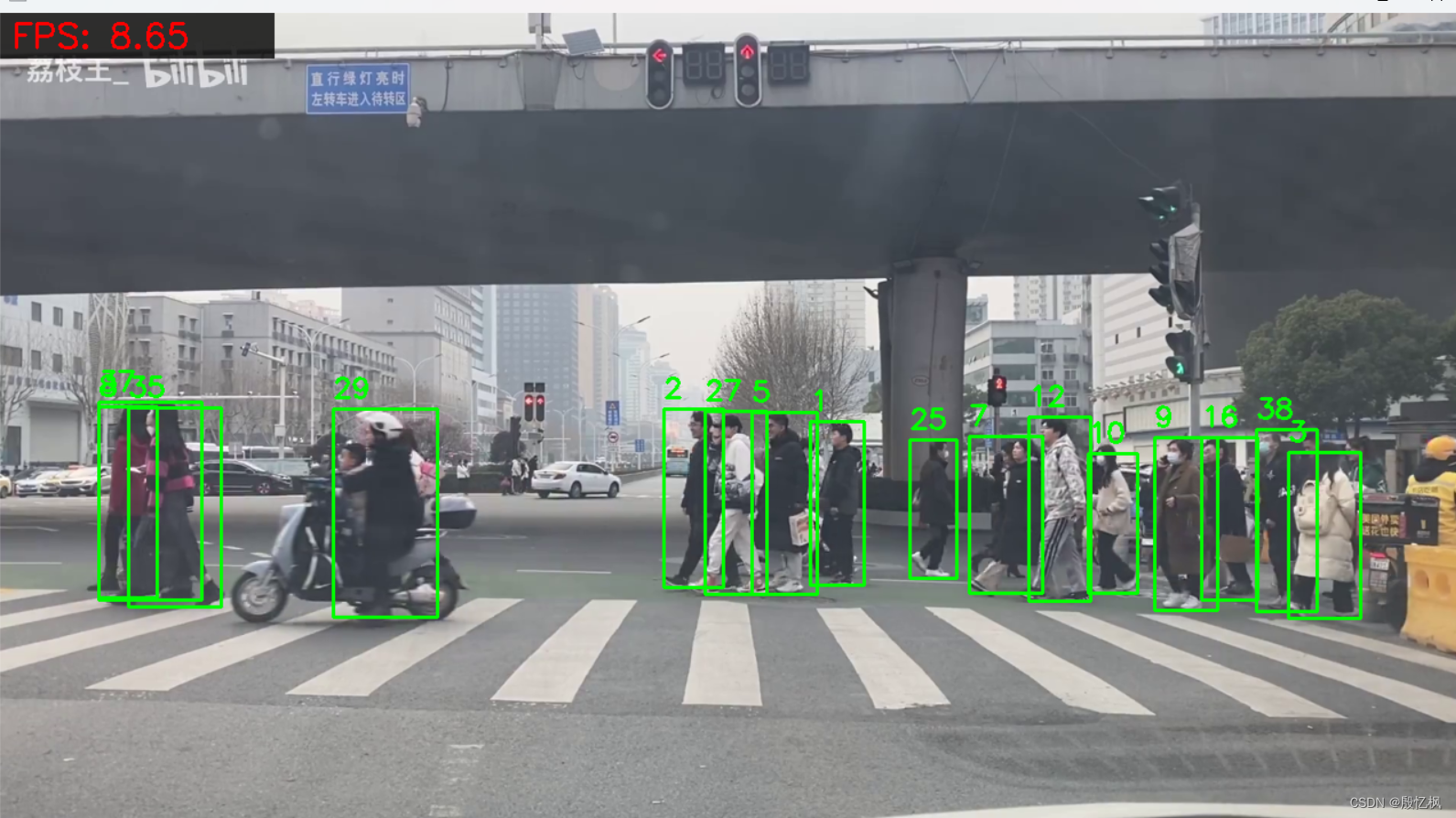
今天给同学们分享一篇生信文章“Identifying the key genes of Epstein-Barr virus-regulated tumour immune microenvironment of gastric carcinomas”,这篇文章发表在Cell Prolif期刊上,影响因子为8.5。

结果解读:
EBV感染改变了胃癌中的免疫浸润特征
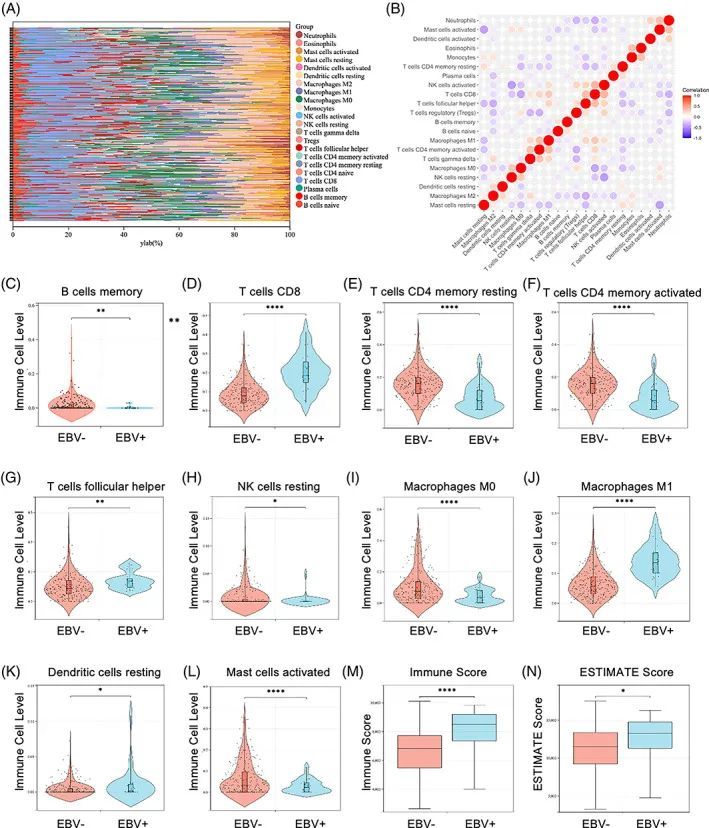
为了确定EBV感染是否影响胃癌中肿瘤浸润免疫细胞(TIICs)的比例,采用了CIBERSORT算法。有趣的是,TIICs的分布因细胞类型而异。一些TIICs在EBVaGC肿瘤组织中具有更多的免疫细胞,而其他TIICs在EBV肿瘤组织中的比例较低(图1A)。在数值方面,EBV+和EBV-组织之间没有显著相关性(图1B)。呈现了一个景观来检测TIICs的分布和重要性。与EBV-样本相比,EBVaGC组织中CD8+ T细胞的比例更高(图1D),滤泡辅助T细胞(图1G)和巨噬细胞M1(图1J)的比例也更高,而B细胞记忆(图1C),CD4记忆静止T细胞(图1E),静止NK细胞(图1H),CD4激活记忆T细胞(图1F),巨噬细胞M0(图1I),树突状细胞(DCs)静止(图1K)和活化肥大细胞(图1L)的比例较低。此外,作者还采用了另一种免疫浸润方法-MCP-counter来重新分析数据。结果显示EBV感染增加了T细胞、细胞毒性淋巴细胞、CD8+ T细胞、NK细胞、单核细胞系和髓样树突状细胞的比例。在EBV阴性组织中,中性粒细胞和成纤维细胞的比例更高。为进一步评估EBV感染是否与GC的免疫浸润有关,作者使用ESTIMATE分数计算了免疫和基质分数。作者分别计算了TCGA-SWAD中EBV阴性(EBV-)和EBV阳性(EBV+)样本的免疫和基质分数。与EBV阴性组织相比,EBV感染显著增加了免疫分数(图1M)和ESTIMATE分数(图1N),而对基质分数没有显著影响。这些结果表明,EBV改变了GC组织中的免疫浸润特征,并促进了炎症反应。
差异基因分析(DEGs)
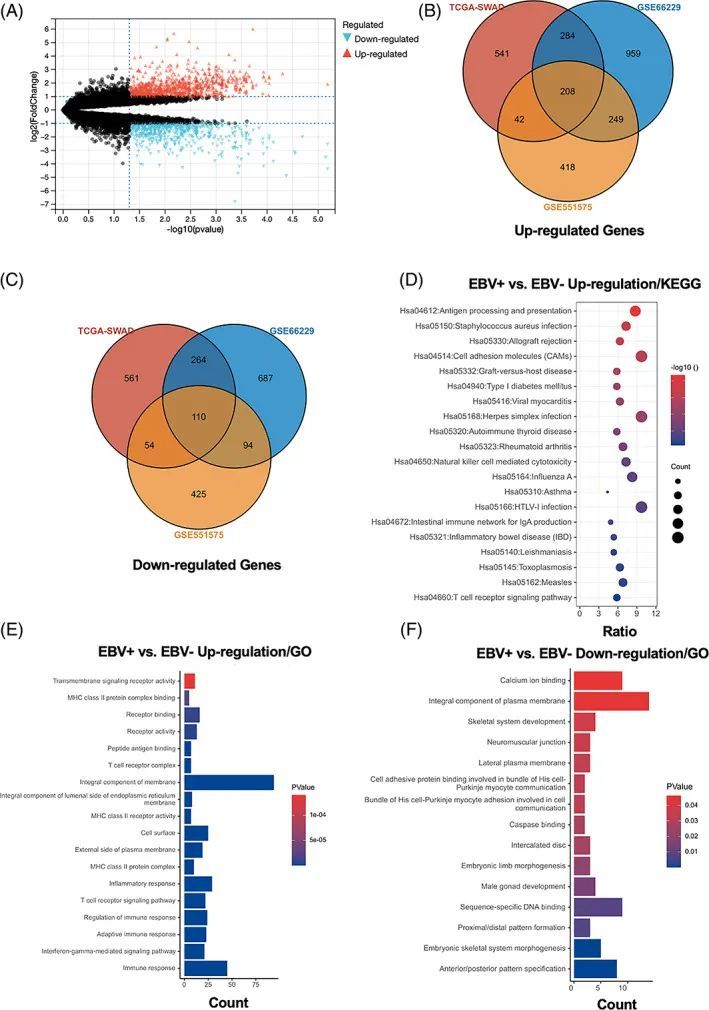
为了确定EBV感染调控的表达谱,作者在分析中包含了三个基因数据集。火山图显示了GSE551575(图2A)、GSE66229和TCGA-SWAD的差异表达基因(DEG)分布情况。这三个数据集中包含了208个相同的上调基因(图2B)和110个相同的下调基因(图2C)。然后,作者对上调或下调的DEG进行了GO和KEGG通路富集分析。呈现了生物过程(BP)、细胞组分和分子功能的前五个显著GO富集结果,以及15个KEGG信号通路。上调的GO分析包括免疫应答、干扰素γ介导的信号通路、适应性免疫应答、免疫应答调节和T细胞受体信号通路等,与免疫和炎症反应显著相关(图2D)。下调的GO分析包括前/后模式规范、胚胎骨骼系统形态发生、近/远模式形成、雄性生殖腺发育和胚胎肢体形态发生等(图2E)。上调的差异表达基因(DEGs)主要富集在抗原处理和呈递、金黄色葡萄球菌感染、移植排斥、细胞黏附分子和移植物抗宿主病等方面(图2F)。然而,只有过氧化物酶体通路与EBV阳性与EBV阴性的胃癌组织之间的下调DEGs相关。这些结果表明,EBV感染与胃癌组织的免疫应答激活密切相关。
WGCNA分析
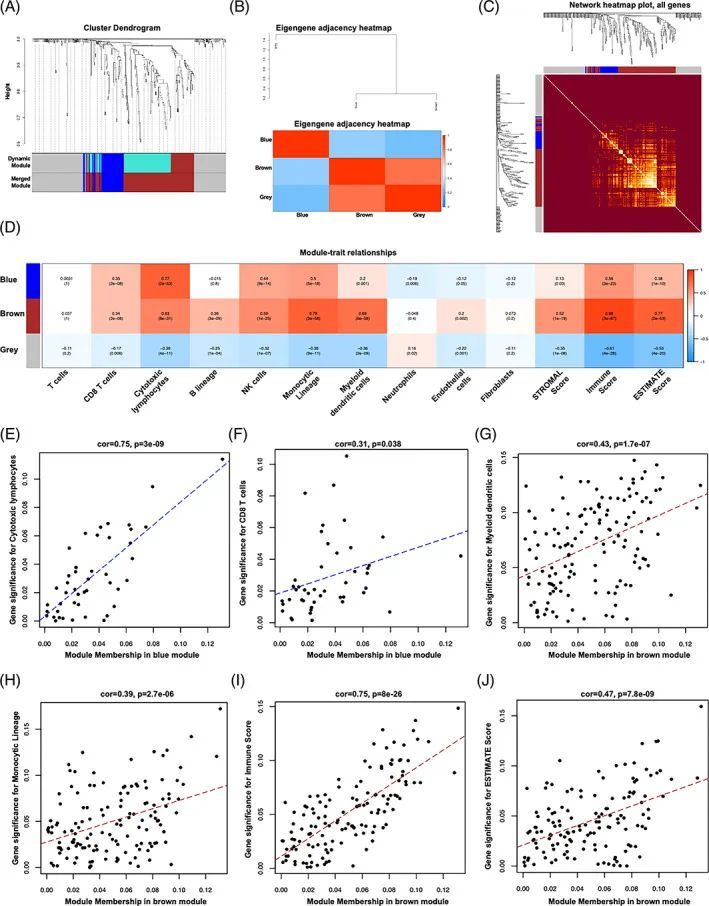
为了确定与免疫浸润相关的关键基因模块,作者使用EBV+与EBV-胃癌组织之间的所有差异表达基因(DEGs)构建了分层聚类树。只发现一个异常样本。接下来,作者进行了共表达分析以构建共表达网络。为了确保一个无标度网络,作者将β的幂设置为8作为软阈值幂。作者鉴定了三个共表达模块,分别为蓝色、棕色和灰色(图3A)。相关热图显示在图3B中。此外,热图的TOM值显示了通过动态方法在模块中限定的基因表达网络中的TOM值(图3C)。灰色模块显示出较低的TOM值,而棕色模块显示出较高的TOM值。为了进一步评估模块与免疫浸润之间的关联,作者在聚类分析中使用了不同的免疫细胞(MCP算法)和ESTIMATE评分。如图3D所示,棕色模块与CD8 T细胞、细胞毒性淋巴细胞、B细胞系、NK细胞、单核细胞系、髓样DCs、内皮细胞和所有ESTIMATE评分呈正相关。蓝色模块与CD8 T细胞、细胞毒性淋巴细胞、NK细胞、单核细胞系、髓样树突状细胞以及所有ESTIMATE评分呈正相关。然而,未分配基因的灰色模块大多与免疫细胞和ESTIMATE评分呈负相关。
GS与不同模块基因之间的关系
由于蓝色模块与细胞毒性淋巴细胞和CD8 T细胞之间存在密切的模块-特性关系,而棕色模块在单核细胞系、髓样树突状细胞、免疫评分和ESTIMATE评分中更为明显。作者对它们之间的GS和MM进行了研究。如图3E、F所示,蓝色模块与细胞毒性淋巴细胞(Cor = 0.75,p = 3 E‐09)和CD8 T细胞(Cor = 0.31,p = 0.038)之间显示出显著的GS‐MM关系。此外,棕色模块与单核细胞系中的GS呈正相关(Cor = 0.43,p = 1.7 E‐07,图3G),与髓样树突状细胞中的GS呈正相关(Cor = 0.39,p = 2.7 E‐06,图3H),与免疫评分呈正相关(Cor = 0.75,p = 8 E‐26,图3I),以及与ESTIMATE评分呈正相关(Cor = 0.47,p = 7.8 E‐09,图3J)。
EBVaGC中的免疫相关基因
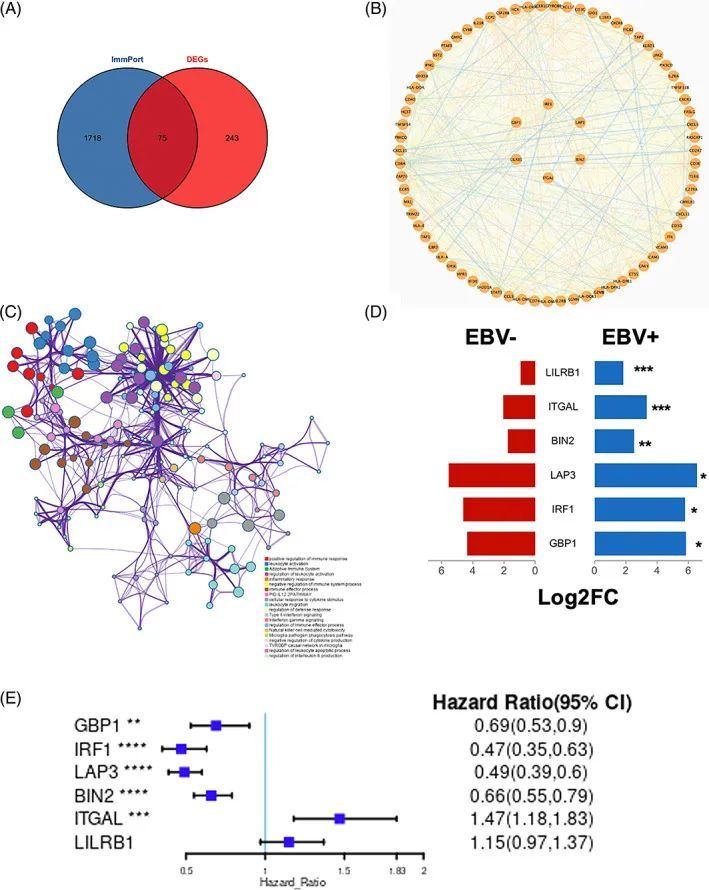
为了确定EBVaGC中的关键免疫相关基因,作者从Immport中获取了与免疫相关基因列表相交的DEGs。如图4A所示,共鉴定出75个基因作为EBVaGC中的关键免疫相关基因。PPI网络显示了这些基因之间的关系,内环的六个基因包括干扰素调节因子1(IRF1)、亮氨酸氨肽酶3(LAP3)、桥接整合蛋白2(BIN2)、整合素亚单位αL(ITGAL)、白细胞免疫球蛋白样受体B1(LILRB1)和鸟苷酸结合蛋白1(GBP1),是蓝色和棕色模块的前三个关键基因(图4B)。
鉴定关键基因
为了确定这些模块中的关键组分,每个模块中的 |k ME | ≥ 0.7 的基因被视为中心节点。所有 |k ME | ≥ 0.7 的中心节点基因都被列出。蓝色和棕色模块分别包含了29个和89个中心节点基因。一些GO富集术语被选择并呈现为网络图。这些中心节点基因在免疫应答的正调控、白细胞活化、适应性免疫系统、白细胞活化调控和炎症反应等方面富集(图4C)。然后,进行了蛋白质相互作用(PPI)富集分析。应用MCODE算法识别密集连接的网络组分。这些中心节点基因被分为四个MCODE部分。对每个MCODE组分分别应用通路和过程富集分析,并将前三个得分最高的术语按p值保留为相应组分的功能描述。为了分析哪些信号通路参与了这些hun基因的调控,进行了KEGG分析。与前五个相关的信号通路是NFKAPPAB 01、PEA3 Q6、CREL 01、NFKB C和ELF1 Q6。蓝色模块(GBP1 [鸟苷酸结合蛋白1]、IRF1 [干扰素调节因子1]、LAP3 [亮氨酸氨肽酶3])和棕色模块(BIN2 [桥接整合蛋白2]、ITGAL [整合素亚单位α L]、LILRB1 [白细胞免疫球蛋白样受体B1])的前三个关键基因被选为代表基因进行下一步分析。
关键基因与胃癌之间的关联
如图4D所示,所有关键关键基因在EBVaGC样本中均过度表达。有趣的是,如图4E所示,高表达的GBP1(HR = 0.69,95% CI = 0.53–0.9)、IRF1(HR = 0.47,95% CI = 0.35–0.63)、LAP3(HR = 0.49,95% CI = 0.39–0.6)和BIN2(HR = 0.66,95% CI = 0.55–0.79)表明GC患者的预后良好。然而,ITGAL的高表达与预后不良相关(HR = 1.47,95% CI = 1.18–1.83)。
为了进一步确认这些关键关键基因与TIICs的特征相关,作者使用了TIMER数据库。GBP1(图5A),IRF1(图5B)和LAP3(图5C)来自蓝色模块,与CD8+ T细胞和CD4+ T细胞呈正相关。此外,棕色模块基因的高表达,包括BIN2(图5D),ITGAL(图5E)和LILRB1(图5F),与巨噬细胞/单核细胞和髓样树突状细胞的比例增加相关。为了进一步探索GC中不同细胞类型中关键关键基因的表达,作者采用了一种单细胞测序方法,使用了两个数据集。给出了GSE134520(图5G)和GSE167297(图5N)中GC细胞的景观。GBP1阳性细胞分布在腺窝粘液、成纤维细胞、单/巨噬细胞和浆细胞中(图5H和O)。BIN2和ITGAL主要在CD8+ T细胞中表达(图5I、K、P、R)。IRF1在各种免疫细胞和肿瘤细胞中高表达(图5J、Q)。LAP3广泛表达于树突状细胞、内皮细胞、成纤维细胞和恶性细胞中(图5L、S)。LILRB1阳性细胞主要与树突状细胞相关(图5M、T)。

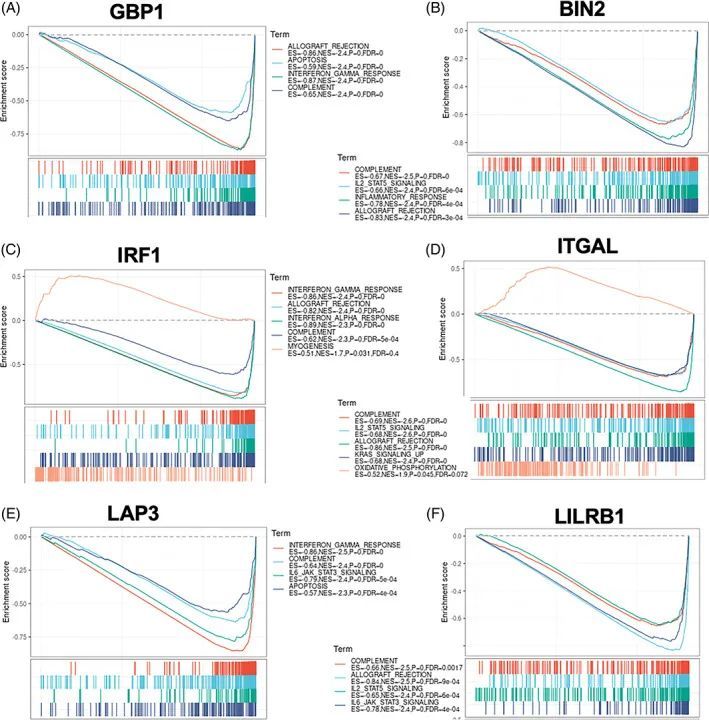
然后,作者通过使用GSEA研究了这些关键关键基因的潜在功能。有趣的是,每个蓝色模块基因的高表达都与干扰素-γ反应呈正相关(图6A、C、E)。此外,棕色模块基因的较高水平与补体系统的激活和IL-2-STAT5信号通路有关(图6B、D、F)。这些结果表明,这些代表性模块基因与GC的免疫调节有关。
EBVaGC肿瘤组织中过表达了GBP1、BIN2和LAP3。
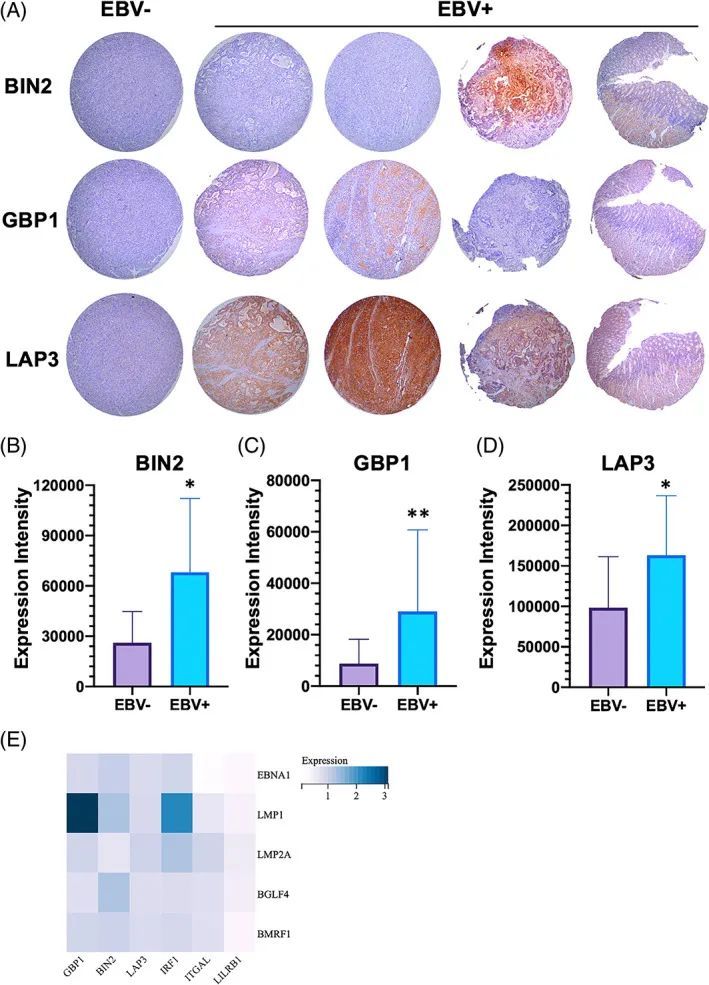
为了检测胃癌组织中关键关键基因的表达谱,作者通过使用免疫组化(IHC)技术在胃癌组织微阵列中研究了GBP1、BIN2和LAP3的表达和分布。共有204例胃癌组织被包括并由桂林医科大学进行了调查。在这201例胃癌组织中,9.45%的样本为EBV阳性(19例)。如图7A所示,GBP1、BIN2和LAP3主要表达在细胞质中。组织微阵列IHC的结果进行了组织化学评分,统计结果显示,EBV阳性组织中GBP1、BIN2和LAP3的表达水平高于EBV阴性组织(图7B、C、D)。
总结
总之,作者的研究表明EBV感染通过主要调节TIICs改变了GC的TME,并通过识别与EBV感染相关的功能模块和关键基因来突出WCGNA的分析能力。蓝色模块(GBP1、IRF1和LAP3)和棕色模块(BIN2、ITGAL和LILRB1)的前三个关键基因被进一步作为代表性基因进行分析。此外,EBV编码的LMP1表达解释了GBP1和IRF1的过表达。重要的是,关键免疫基因的激活更多地依赖于整个EBV病毒颗粒的侵袭性,而不是单个EBV潜伏/溶解基因的表达。