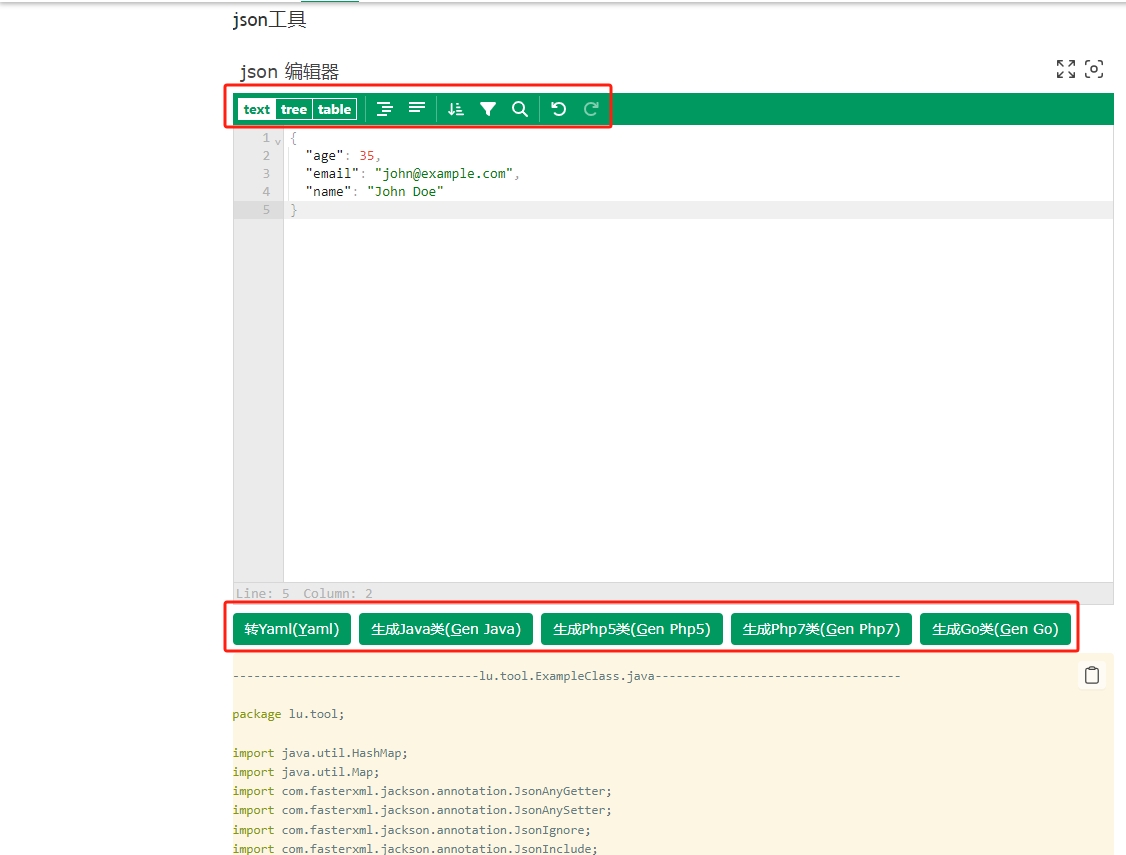
1. 项目文件介绍
本章为二值图像的分割任务做统一实现,下面是项目的实现目录
项目和文章绑定了,之前没用过,不知道行不行
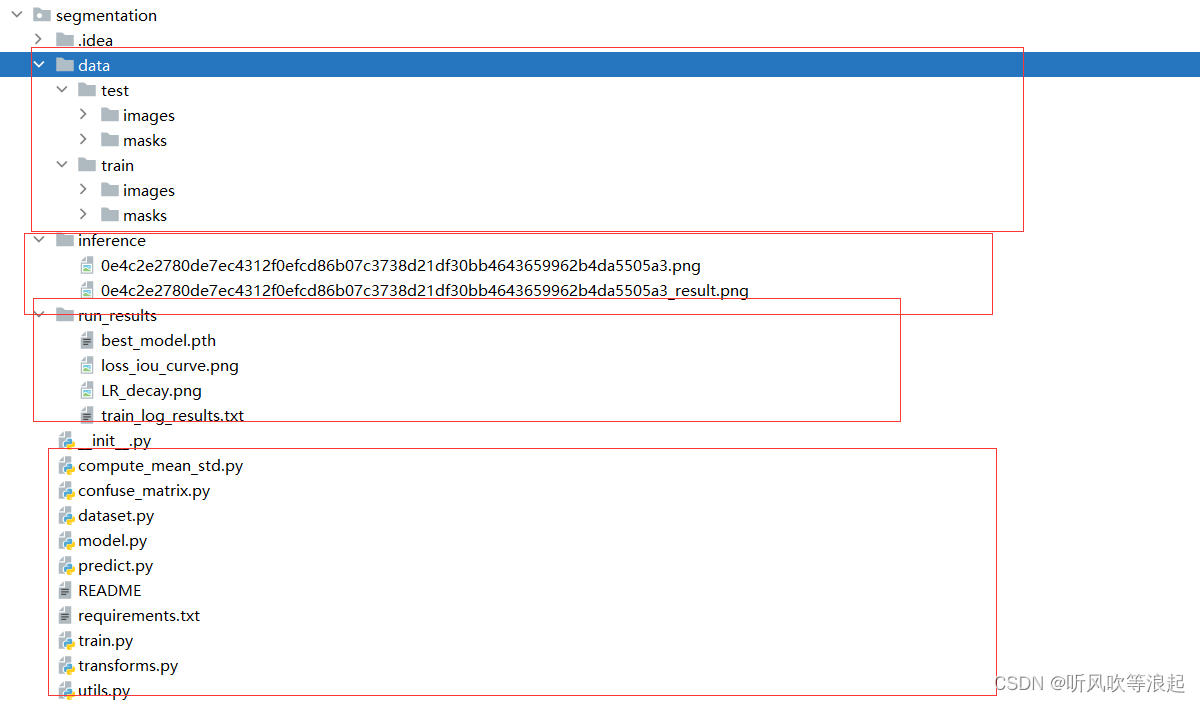
- data 文件夹下负责摆放数据的训练集+测试集
- inference 负责放待推理的图片(支持多张图片预测分割)
- run_results 是网络训练过程的日志文件
- python 代码介绍
| compute_mean_std | 计算训练集图像灰度的均值和方差 | 像素分布(-1,1)之间可以让网络更好的收敛 |
| confuse_matrix | 混淆矩阵 | 通过分类任务的混淆矩阵,计算不同像素的iou |
| dataset | 数据集的加载代码 | 根据数据集在data文件下的摆放,加载数据 |
| model | 模型,这里采用vgg为骨干网络 | resnet比vgg有更好的提取特征能力,这个后续介绍 |
| predict | 推理代码 | |
| train | 模型的训练代码 | |
| transforms | 对图像预处理的重构 | 不同于分类任务,图像的缩放,相应的标签也要进行缩放 |
| utils | 关于训练过程的工具函数 |
2. 使用代码
README 文件有详细介绍!!
1. 环境搭建
搭建好项目需要的环境后,下载 requirements.txt 需要的库文件即可
2. 数据集的摆放
按照指定位置摆放,文件夹的名称不可更改,否则需要重新编写dataset文件 !!!

3. 关于超参数的设定
因为transforms 对图像进行缩放后中心裁剪,所以训练train 文件前,根据训练图片的大小进行更改。训练过程的超参数文件也可以在这里更改
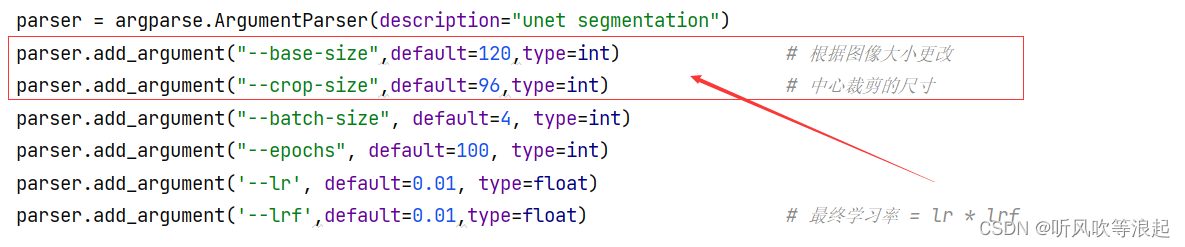
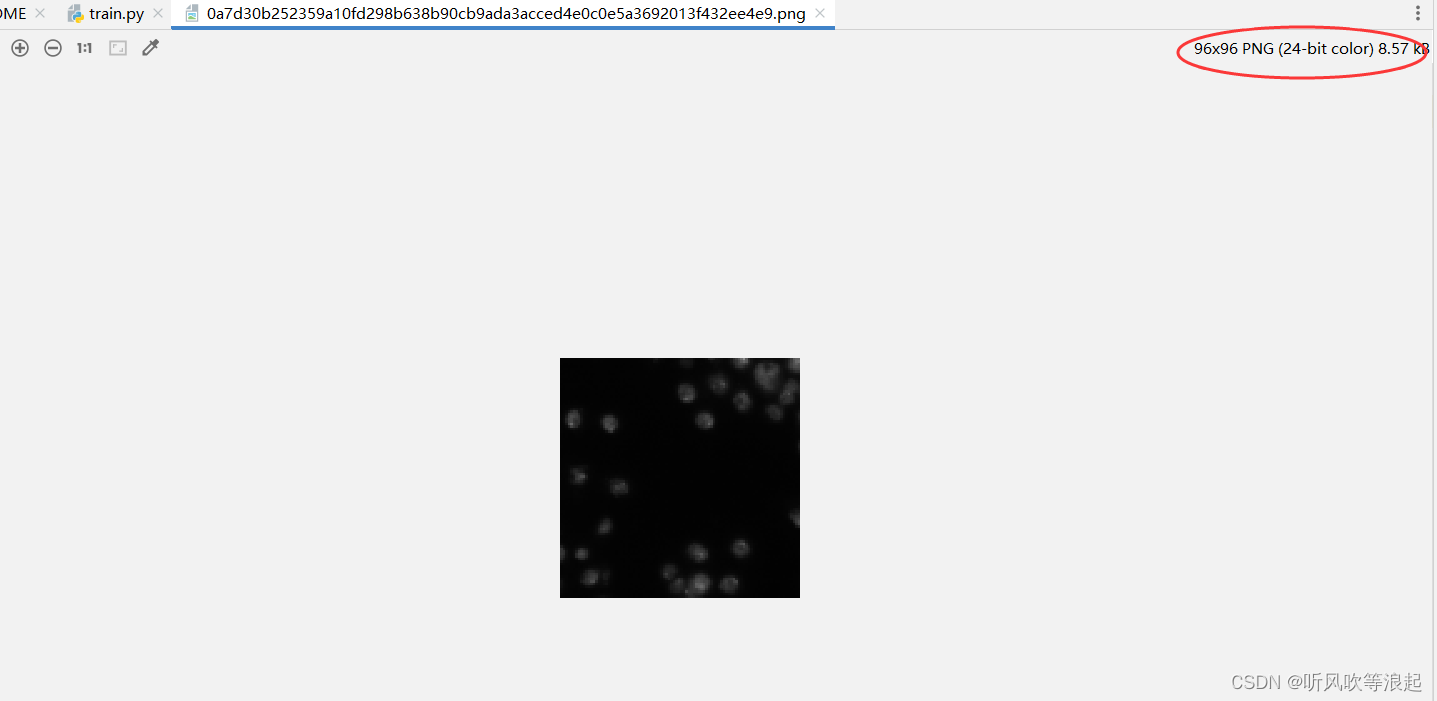
例如这里的训练图片是96*96的空间分辨率,所以设定的中心裁剪为96
至于base_size 是图像进行缩放的比例,如下图,在120的 0.5到1.5倍数之间随机缩放。有部分疑问后面聊
4. 开始训练
运行train 文件即可,会在train文件生成下面四个文件

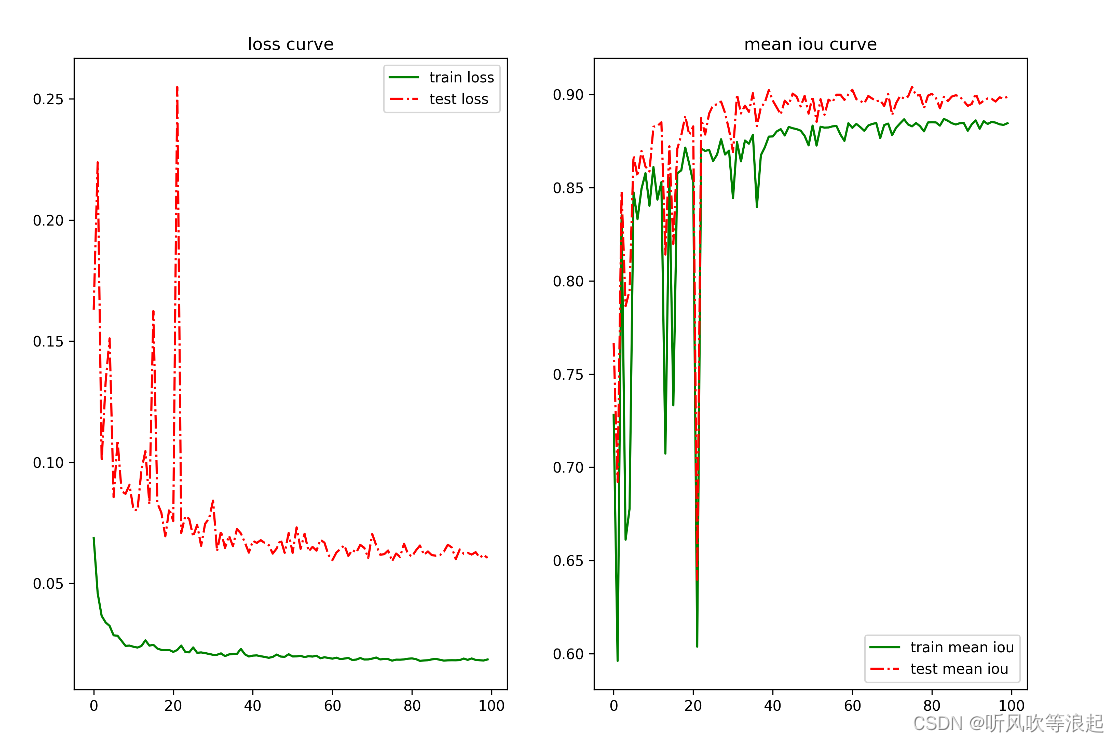
下面是细胞分割的训练展示
loss iou 曲线:
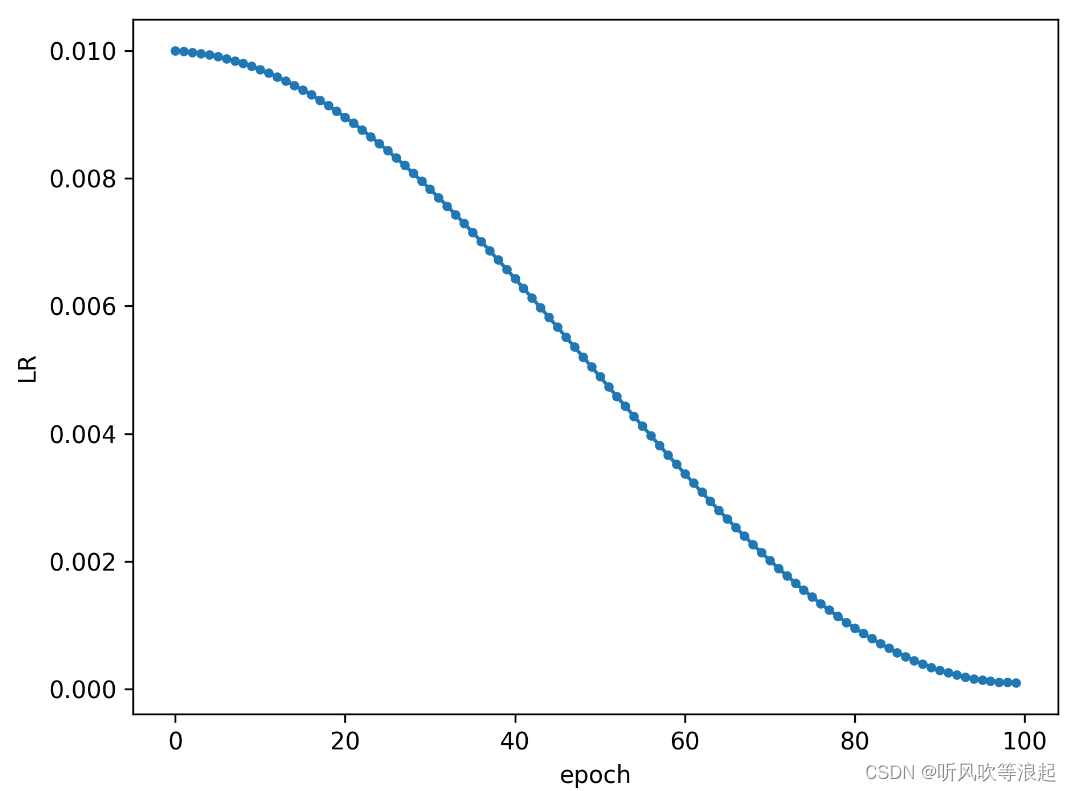
学习率衰减曲线:这里使用的cos自适应衰减
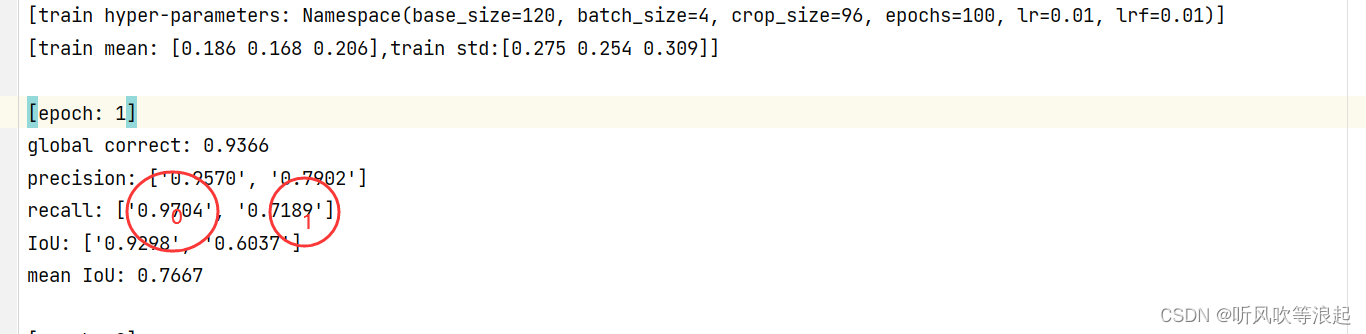
训练日志:左侧的一列是背影0的指标,右侧是1的指标,可以参考之前的介绍:混淆矩阵、语义分割的指标
5. 如何预测
因为训练的预处理mean和std是计算得到的,所以预测之前需要重新填写,如下
只需要将train得到的填入下方就行了


3. 有些问题
因为本人在训练过程有几点困惑,所以写在这,方便大佬解答...
1. transform 的缩放设定
因为图像分割最终的目的都是将前景图像从背景中抠出来,这就涉及到图像的尺寸问题。
例如,标准的unet 输入是固定的,例如460*460或者512*512,但大部分的图片这个尺寸。虽说图像处理可以进行缩放解决,但将label缩放插值方法总觉得差点意思,这会导致原本的前景被改变。而且,就算将训练集统一缩放了,预测新的图片呢?也需要缩放吗?
于是本章利用了随机缩放图像大小的方法,这样随机输入的图像本身就是不固定的,相当于多尺度输入吧,这样随机预测新图片的时候,输入的图像大小也就无所谓了
min_size = int(0.5 * base_size)
max_size = int(1.5 * base_size)2. 为什么用 vgg 不用 resnet
vgg 的效果确实不如 resnet 提取特征强,作者本人网上找了很多换成resnet的代码,但都用一个问题,不能任意输入大小
本人在resnet的代码上更改了很久,没有实现,太菜了...
这样就产生了第一个问题,最后权衡一二,使用了本章vgg为骨干的unet模型。这里的unet添加了代码,可以支持任何尺度的输入
当然,如果不介意输入尺寸的问题,可以利用自带库,直接调用
参考:Unet 基于TCGA颅脑肿瘤MRI分割(高阶API分割模型)


3. 关于 dataset 部分
对应于固定的分割任务,dataset都是需要自己重构的,为了尽量代码复用,所以数据集的摆放必须遵循固定的顺序
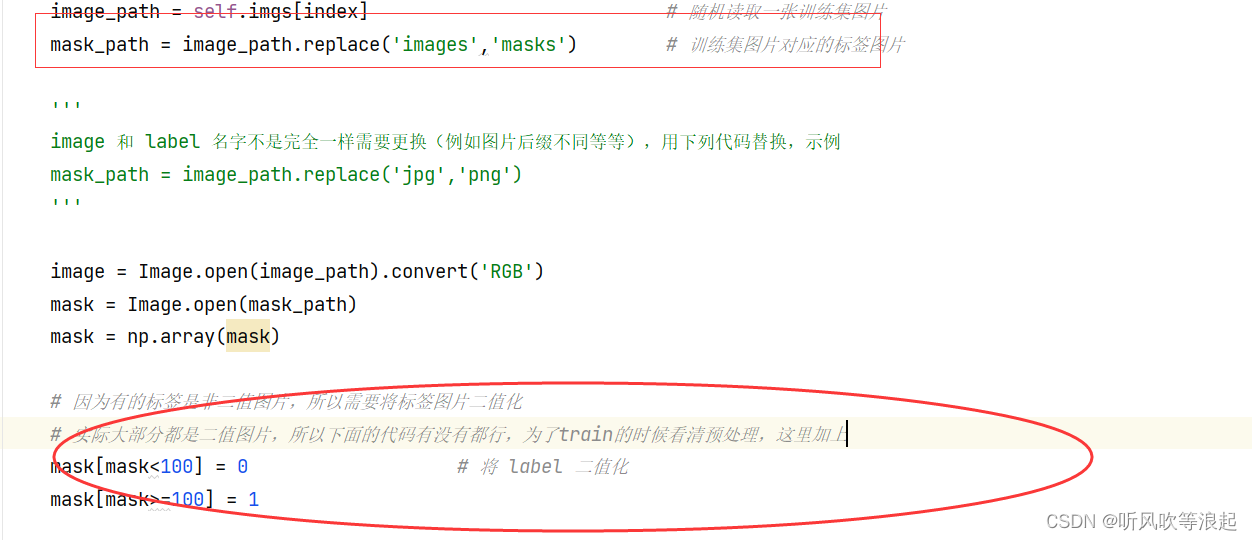
有些训练图片和标签图像名字不是严格对应的,比如后缀啊,或者文件名啊。可以利用python 批处理进行更改,或者在dataset 里面重构
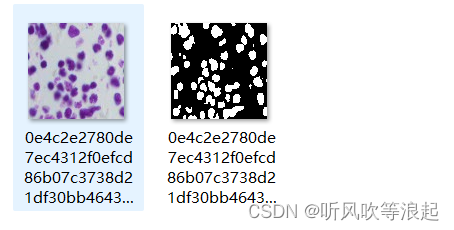
对于,label 不是标准的二值图像(看起来黑白,其实中间包含了其他像素值),通过阈值进行处理。
需要注意的是,qq截图可以显示灰度值,但不是准确的。将图片放到最大,可能就是二值图片,因为图像缩放,分辨器显示的问题。(作者之前在这吃过不少亏...)
二值化还一个好处,将灰度值映射成120的话,可以观察train过程的预处理图像长啥样
将train 部分的代码打开即可
好像不需要更改也行,plot 自动调色了....
# # 可视化数据,二值化中,将 dataset的标签映射改成 120会看的更清楚
# # 可以查看具体的训练图像被预处理成啥样
# dataloader = next(iter(trainLoader))
# plot(data_loader=dataloader,mean=train_mean,std=train_std)
# return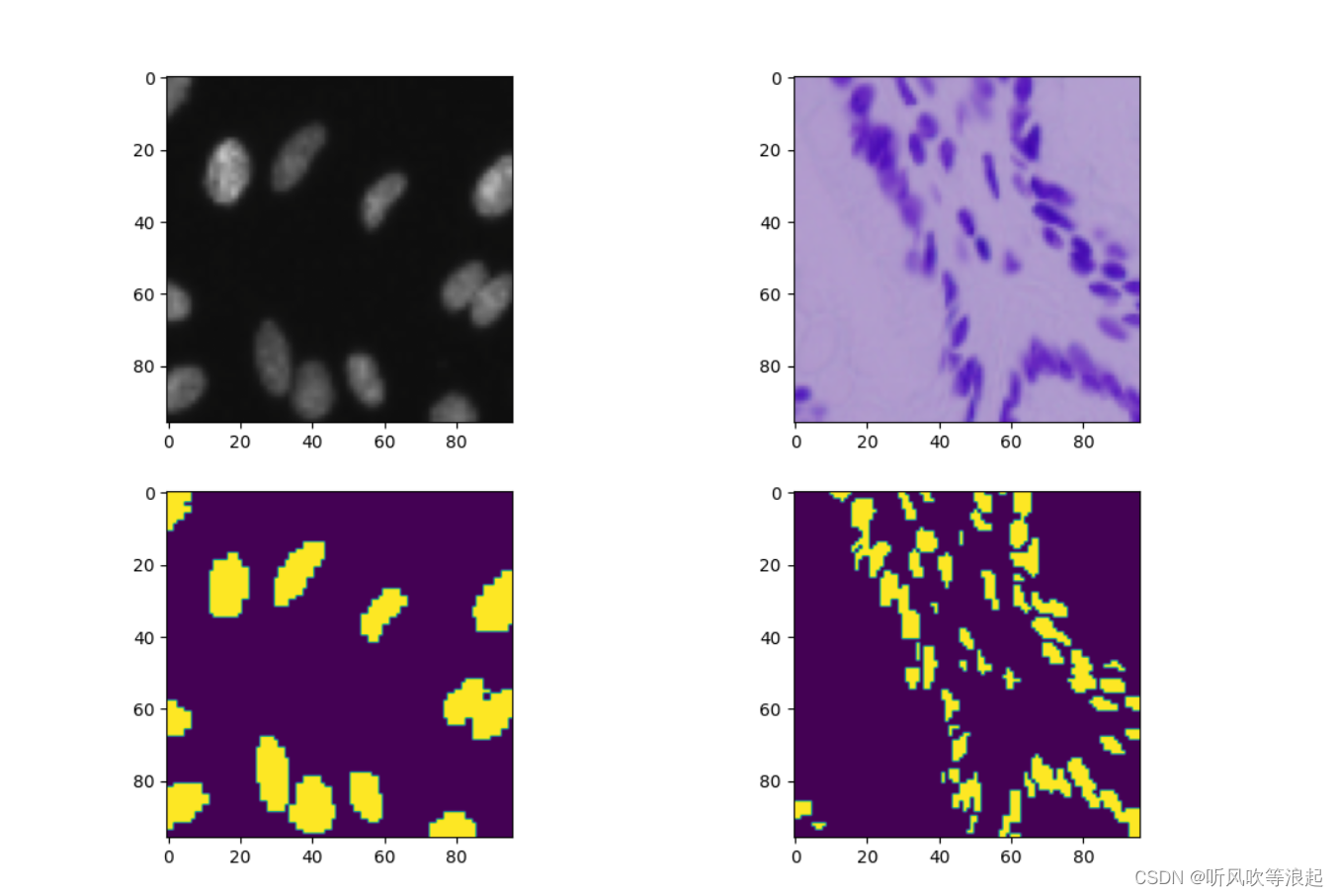
还有别的问题,可以在评论区在交流....