题目详情:

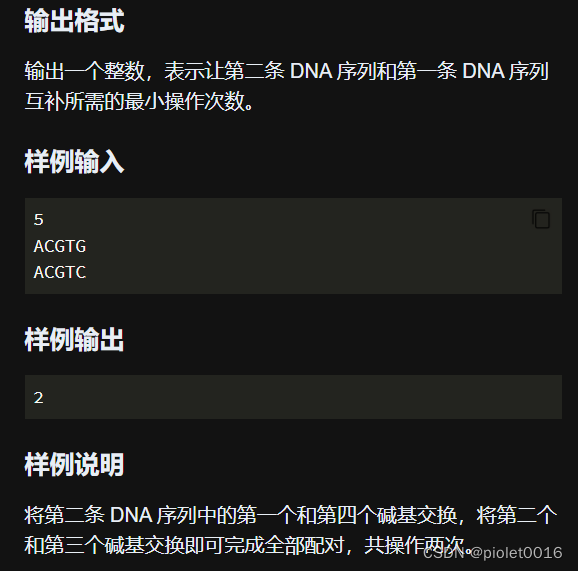
简单翻译:
主要思路:
1 本题采用贪心思路,要使调整次数最少,就是尽量交换两个碱基对,而不是单个替换,因为本题已经说明只能每个碱基对只能交换一次,所以不考虑A与B交换再与C交换这种情况
2 利用静态的map记录碱基对之间的配对关系
3 当以s1字符串为基准扫描到一个碱基对不配对时,扫描s2字符串,看之后有没有一个碱基对能与当前碱基对满足交叉替换关系
第一次写错误:
1 c++中string字符串的length方法使用时s2.length()
2 c++中pair这类在传参的时候要把类型也写明,不能只写一个pair
代码实现:
#include <bits/stdc++.h>
using namespace std;
map<char, char> DNA = {{'A', 'T'}, {'T', 'A'}, {'C', 'G'}, {'G', 'C'}};
bool FindAnotherPair(string &s1, string &s2, pair<char, char> initialPair, int position) { //pair这类在传参的时候要把类型也写明,不能只写一个pair
bool flag = false;
for(int i = 0; i < s2.length(); i++) {
if(s2[i] == DNA[initialPair.first] && s1[i] == DNA[initialPair.second]) {
char tmp = s2[position];
s2[position] = s2[i];
s2[i] = tmp;
flag = true;
break;
}
}
return flag;
}
int main() {
string s1, s2;
int N;
scanf("%d", &N);
cin >> s1 >> s2;
int count = 0;
for(int i = 0; i < N; i++) {
if(s2[i] != DNA[s1[i]]) {
if(FindAnotherPair(s1, s2, make_pair(s1[i], s2[i]), i)) {
count++;
}
else {
s2[i] = DNA[s1[i]];
count++;
}
}
}
printf("%d", count);
return 0;
}