目录
2003. 每棵子树内缺失的最小基因值
题目描述:
实现代码与解析:
dfs + 启发式合并
原理思路:
2003. 每棵子树内缺失的最小基因值
题目描述:
有一棵根节点为 0 的 家族树 ,总共包含 n 个节点,节点编号为 0 到 n - 1 。给你一个下标从 0 开始的整数数组 parents ,其中 parents[i] 是节点 i 的父节点。由于节点 0 是 根 ,所以 parents[0] == -1 。
总共有 105 个基因值,每个基因值都用 闭区间 [1, 105] 中的一个整数表示。给你一个下标从 0 开始的整数数组 nums ,其中 nums[i] 是节点 i 的基因值,且基因值 互不相同 。
请你返回一个数组 ans ,长度为 n ,其中 ans[i] 是以节点 i 为根的子树内 缺失 的 最小 基因值。
节点 x 为根的 子树 包含节点 x 和它所有的 后代 节点。
示例 1:

输入:parents = [-1,0,0,2], nums = [1,2,3,4] 输出:[5,1,1,1] 解释:每个子树答案计算结果如下: - 0:子树包含节点 [0,1,2,3] ,基因值分别为 [1,2,3,4] 。5 是缺失的最小基因值。 - 1:子树只包含节点 1 ,基因值为 2 。1 是缺失的最小基因值。 - 2:子树包含节点 [2,3] ,基因值分别为 [3,4] 。1 是缺失的最小基因值。 - 3:子树只包含节点 3 ,基因值为 4 。1是缺失的最小基因值。
示例 2:
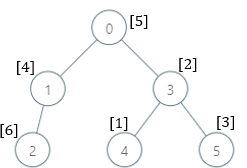
输入:parents = [-1,0,1,0,3,3], nums = [5,4,6,2,1,3] 输出:[7,1,1,4,2,1] 解释:每个子树答案计算结果如下: - 0:子树内包含节点 [0,1,2,3,4,5] ,基因值分别为 [5,4,6,2,1,3] 。7 是缺失的最小基因值。 - 1:子树内包含节点 [1,2] ,基因值分别为 [4,6] 。 1 是缺失的最小基因值。 - 2:子树内只包含节点 2 ,基因值为 6 。1 是缺失的最小基因值。 - 3:子树内包含节点 [3,4,5] ,基因值分别为 [2,1,3] 。4 是缺失的最小基因值。 - 4:子树内只包含节点 4 ,基因值为 1 。2 是缺失的最小基因值。 - 5:子树内只包含节点 5 ,基因值为 3 。1 是缺失的最小基因值。
示例 3:
输入:parents = [-1,2,3,0,2,4,1], nums = [2,3,4,5,6,7,8] 输出:[1,1,1,1,1,1,1] 解释:所有子树都缺失基因值 1 。
提示:
n == parents.length == nums.length2 <= n <= 105- 对于
i != 0,满足0 <= parents[i] <= n - 1 parents[0] == -1parents表示一棵合法的树。1 <= nums[i] <= 105nums[i]互不相同。
实现代码与解析:
dfs + 启发式合并
class Solution {
public:
// 邻接表
vector<int> h = vector<int>(1e5 + 10, -1); vector<int> e = vector<int>(1e5 + 10, 0); vector<int> ne = vector<int>(1e5 + 10, 0);
int idx = 0;
void add(int a, int b) {
e[idx] = b, ne[idx] = h[a], h[a] = idx++;
}
vector<int> smallestMissingValueSubtree(vector<int>& parents, vector<int>& nums) {
int n = parents.size();
// 存图 加边 邻接表
for (int i = 1; i < n; i++) { // 这里从1开始,找半天错。。
add(parents[i], i);
}
vector<int> res(n, 1); // 结果数组
vector<unordered_set<int>> gene(n); // 包含此节点 与 其子树 的基因合集set
function<int(int)> dfs = [&](int cur) -> int{
gene[cur].insert(nums[cur]); // 当前节点基因值放入
for (int i = h[cur]; ~i; i = ne[i]) {
int j = e[i]; // 子节点
res[cur] = max(res[cur], dfs(j)); // 此节点为根缺失的最小的基因,一定比子树缺失的最小基因大或等于,所以这里取个max
// 向上传递合集,小的合并到大的效率快,所以先判断(选择是否先交换),在树中属于中序处理
if (gene[cur].size() < gene[j].size()) {
gene[cur].swap(gene[j]);
}
gene[cur].merge(gene[j]); // 合并
}
// 根据res寻找此节点的结果, 一定比子树大或等于,++遍历即可,找到第一个没有的最小基因
while (gene[cur].count(res[cur]) > 0) {
res[cur]++;
}
return res[cur];
};
dfs(0); // dfs
return res;
}
};原理思路:
vector<unordered_set<int>> gene(n); // 包含此节点 与 其子树 的基因合集set
dfs返回此节点的结果(缺失基因最小值),递归向上处理,每次将子树的集合与其根节点集合合并,根节点的缺失最小值一定大于等于其子树。从最大子树缺失的最小基因开始++遍历,找到第一个不在集合中的值,就为以此节点为根的树的缺失的最小基因。
此题的核心,就是我们不仅要向上传递子树缺失的最小基因,还要根据树中包含的节点来找出缺失基因,所以每个节点多了set来记录以它为根的树包含的节点,每次向上来合并set是此题的关键。
![[论文笔记]BGE](https://img-blog.csdnimg.cn/img_convert/b1719d974e103a39ca7e66ff844f21f4.png)