一、说明
在第一部分中,我们研究了如何使用 SVI 对简单的贝叶斯线性回归模型进行推理。在本教程中,我们将探索更具表现力的指南以及精确的推理技术。我们将使用与之前相同的数据集。
二、模块导入
[1]:
%reset -sf
[2]:
import logging
import os
import torch
import matplotlib.pyplot as plt
import numpy as np
import pandas as pd
import seaborn as sns
from torch.distributions import constraints
import pyro
import pyro.distributions as dist
import pyro.optim as optim
pyro.set_rng_seed(1)
assert pyro.__version__.startswith('1.8.6')
[3]:
%matplotlib inline
plt.style.use('default')
logging.basicConfig(format='%(message)s', level=logging.INFO)
smoke_test = ('CI' in os.environ)
pyro.set_rng_seed(1)
DATA_URL = "https://d2hg8soec8ck9v.cloudfront.net/datasets/rugged_data.csv"
rugged_data = pd.read_csv(DATA_URL, encoding="ISO-8859-1")
三、贝叶斯线性回归
我们的目标是再次根据数据集中的两个特征(该国家是否位于非洲及其地形坚固指数)来预测一个国家的人均 GDP 对数,但我们将探索更具表现力的指南。
3.1 模型+指南
我们将再次写出模型,类似于第一部分,但明确不使用 PyroModule。我们将使用相同的先验写出回归中的每一项。 bA和bR是is_cont_africa和roughness对应的回归系数,a是截距,bAR是两个特征之间的相关因子。
编写指南的过程与我们模型的构建非常相似,主要区别在于指南参数需要可训练。为此,我们使用在 ParamStore 中注册指南参数pyro.param()。注意尺度参数的正约束。
[4]:
def model(is_cont_africa, ruggedness, log_gdp):
a = pyro.sample("a", dist.Normal(0., 10.))
b_a = pyro.sample("bA", dist.Normal(0., 1.))
b_r = pyro.sample("bR", dist.Normal(0., 1.))
b_ar = pyro.sample("bAR", dist.Normal(0., 1.))
sigma = pyro.sample("sigma", dist.Uniform(0., 10.))
mean = a + b_a * is_cont_africa + b_r * ruggedness + b_ar * is_cont_africa * ruggedness
with pyro.plate("data", len(ruggedness)):
pyro.sample("obs", dist.Normal(mean, sigma), obs=log_gdp)
def guide(is_cont_africa, ruggedness, log_gdp):
a_loc = pyro.param('a_loc', torch.tensor(0.))
a_scale = pyro.param('a_scale', torch.tensor(1.),
constraint=constraints.positive)
sigma_loc = pyro.param('sigma_loc', torch.tensor(1.),
constraint=constraints.positive)
weights_loc = pyro.param('weights_loc', torch.randn(3))
weights_scale = pyro.param('weights_scale', torch.ones(3),
constraint=constraints.positive)
a = pyro.sample("a", dist.Normal(a_loc, a_scale))
b_a = pyro.sample("bA", dist.Normal(weights_loc[0], weights_scale[0]))
b_r = pyro.sample("bR", dist.Normal(weights_loc[1], weights_scale[1]))
b_ar = pyro.sample("bAR", dist.Normal(weights_loc[2], weights_scale[2]))
sigma = pyro.sample("sigma", dist.Normal(sigma_loc, torch.tensor(0.05)))
mean = a + b_a * is_cont_africa + b_r * ruggedness + b_ar * is_cont_africa * ruggedness
[5]:
# Utility function to print latent sites' quantile information.
def summary(samples):
site_stats = {}
for site_name, values in samples.items():
marginal_site = pd.DataFrame(values)
describe = marginal_site.describe(percentiles=[.05, 0.25, 0.5, 0.75, 0.95]).transpose()
site_stats[site_name] = describe[["mean", "std", "5%", "25%", "50%", "75%", "95%"]]
return site_stats
# Prepare training data
df = rugged_data[["cont_africa", "rugged", "rgdppc_2000"]]
df = df[np.isfinite(df.rgdppc_2000)]
df["rgdppc_2000"] = np.log(df["rgdppc_2000"])
train = torch.tensor(df.values, dtype=torch.float)
3.2 SVI推理
和之前一样,我们将使用 SVI 来执行推理。
[6]:
from pyro.infer import SVI, Trace_ELBO
svi = SVI(model,
guide,
optim.Adam({"lr": .05}),
loss=Trace_ELBO())
is_cont_africa, ruggedness, log_gdp = train[:, 0], train[:, 1], train[:, 2]
pyro.clear_param_store()
num_iters = 5000 if not smoke_test else 2
for i in range(num_iters):
elbo = svi.step(is_cont_africa, ruggedness, log_gdp)
if i % 500 == 0:
logging.info("Elbo loss: {}".format(elbo))
埃尔博损失:5795.467590510845
埃尔博损失:415.8169444799423
埃尔博损失:250.71916329860687
埃尔博损失:247.19457268714905
埃尔博损失:249.2004036307335
埃尔博损失:250.96484470367432
埃尔博损失:249.35092514753342
埃尔博损失:248.7831552028656
埃尔博损失:248.62140649557114
埃尔博损失:250.4274433851242
[7]:
from pyro.infer import Predictive
num_samples = 1000
predictive = Predictive(model, guide=guide, num_samples=num_samples)
svi_samples = {k: v.reshape(num_samples).detach().cpu().numpy()
for k, v in predictive(log_gdp, is_cont_africa, ruggedness).items()
if k != "obs"}
让我们观察模型中不同潜在变量的后验分布。
[8]:
for site, values in summary(svi_samples).items():
print("Site: {}".format(site))
print(values, "\n")
站点:a
平均标准差 5% 25% 50% 75% 95%
0 9.177024 0.059607 9.07811 9.140463 9.178211 9.217098 9.27152
地点:bA
平均标准差 5% 25% 50% 75% 95%
0 -1.890622 0.122805 -2.08849 -1.979107 -1.887476 -1.803683 -1.700853
站点:bR
平均标准差 5% 25% 50% 75% 95%
0 -0.157847 0.039538 -0.22324 -0.183673 -0.157873 -0.133102 -0.091713
站点:bAR
平均标准差 5% 25% 50% 75% 95%
0 0.304515 0.067683 0.194583 0.259464 0.304907 0.348932 0.415128
网站:西格玛
平均标准差 5% 25% 50% 75% 95%
0 0.902898 0.047971 0.824166 0.870317 0.901981 0.935171 0.981577
3.3 HMC
与使用变分推理(为我们提供潜在变量的近似后验)相反,我们还可以使用马尔可夫链蒙特卡罗(MCMC)进行精确推理,这是一类算法,在极限情况下,允许我们从真实的样本中提取无偏样本。后部。我们将使用的算法称为 No-U Turn Sampler (NUTS) [1],它提供了一种运行哈密顿蒙特卡罗的高效且自动化的方法。它比变分推理稍慢,但提供了精确的估计。
[9]:
from pyro.infer import MCMC, NUTS
nuts_kernel = NUTS(model)
mcmc = MCMC(nuts_kernel, num_samples=1000, warmup_steps=200)
mcmc.run(is_cont_africa, ruggedness, log_gdp)
hmc_samples = {k: v.detach().cpu().numpy() for k, v in mcmc.get_samples().items()}
样本:100%|██████████| 1200/1200 [00:30,38.99it/s,步长=2.76e-01,根据。概率=0.934]
[10]:
for site, values in summary(hmc_samples).items():
print("Site: {}".format(site))
print(values, "\n")
站点:a
平均标准差 5% 25% 50% 75% 95%
0 9.182098 0.13545 8.958712 9.095588 9.181347 9.277673 9.402615
地点:bA
平均标准差 5% 25% 50% 75% 95%
0 -1.847651 0.217768 -2.19934 -1.988024 -1.846978 -1.70495 -1.481822
站点:bR
平均标准差 5% 25% 50% 75% 95%
0 -0.183031 0.078067 -0.311403 -0.237077 -0.185945 -0.131043 -0.051233
站点:bAR
平均标准差 5% 25% 50% 75% 95%
0 0.348332 0.127478 0.131907 0.266548 0.34641 0.427984 0.560221
网站:西格玛
平均标准差 5% 25% 50% 75% 95%
0 0.952041 0.052024 0.869388 0.914335 0.949961 0.986266 1.038723
3.4 比较后验分布
让我们将通过变分推理获得的潜在变量的后验分布与哈密顿蒙特卡罗获得的潜在变量的后验分布进行比较。如下所示,对于变分推理,不同回归系数的边缘分布相对于真实后验(来自 HMC)而言是分散不足的。这是通过变分推理最小化的KL(q||p)损失(真实后验与近似后验的 KL 散度)的伪影。
当我们绘制来自联合后验分布的不同横截面并覆盖来自变分推理的近似后验时,可以更好地看到这一点。请注意,由于我们的变分族具有对角协方差,因此我们无法对潜在变量之间的任何相关性进行建模,并且所得的近似值过于自信(分散不足)
[11]:
sites = ["a", "bA", "bR", "bAR", "sigma"]
fig, axs = plt.subplots(nrows=2, ncols=2, figsize=(12, 10))
fig.suptitle("Marginal Posterior density - Regression Coefficients", fontsize=16)
for i, ax in enumerate(axs.reshape(-1)):
site = sites[i]
sns.distplot(svi_samples[site], ax=ax, label="SVI (DiagNormal)")
sns.distplot(hmc_samples[site], ax=ax, label="HMC")
ax.set_title(site)
handles, labels = ax.get_legend_handles_labels()
fig.legend(handles, labels, loc='upper right');

[12]:
fig, axs = plt.subplots(nrows=1, ncols=2, figsize=(12, 6))
fig.suptitle("Cross-section of the Posterior Distribution", fontsize=16)
sns.kdeplot(x=hmc_samples["bA"], y=hmc_samples["bR"], ax=axs[0], shade=True, label="HMC")
sns.kdeplot(x=svi_samples["bA"], y=svi_samples["bR"], ax=axs[0], label="SVI (DiagNormal)")
axs[0].set(xlabel="bA", ylabel="bR", xlim=(-2.5, -1.2), ylim=(-0.5, 0.1))
sns.kdeplot(x=hmc_samples["bR"], y=hmc_samples["bAR"], ax=axs[1], shade=True, label="HMC")
sns.kdeplot(x=svi_samples["bR"], y=svi_samples["bAR"], ax=axs[1], label="SVI (DiagNormal)")
axs[1].set(xlabel="bR", ylabel="bAR", xlim=(-0.45, 0.05), ylim=(-0.15, 0.8))
handles, labels = axs[1].get_legend_handles_labels()
fig.legend(handles, labels, loc='upper right');

3.5 多元正态指南
与之前从对角正态指南获得的结果相比,我们现在将使用从多元正态分布的 Cholesky 分解生成样本的指南。这使我们能够通过协方差矩阵捕获潜在变量之间的相关性。如果我们手动编写此代码,我们将需要组合所有潜在变量,以便我们可以联合采样多元正态分布。
[13]:
from pyro.infer.autoguide import AutoMultivariateNormal, init_to_mean
guide = AutoMultivariateNormal(model, init_loc_fn=init_to_mean)
svi = SVI(model,
guide,
optim.Adam({"lr": .01}),
loss=Trace_ELBO())
is_cont_africa, ruggedness, log_gdp = train[:, 0], train[:, 1], train[:, 2]
pyro.clear_param_store()
for i in range(num_iters):
elbo = svi.step(is_cont_africa, ruggedness, log_gdp)
if i % 500 == 0:
logging.info("Elbo loss: {}".format(elbo))
埃尔博损失:703.0100790262222
埃尔博损失:444.6930855512619
埃尔博损失:258.20718491077423
埃尔博损失:249.05364602804184
埃尔博损失:247.2170884013176
埃尔博损失:247.28261297941208
埃尔博损失:246.61236548423767
埃尔博损失:249.86004841327667
埃尔博损失:249.1157277226448
埃尔博损失:249.86634194850922
我们再看一下后背的形状。您可以看到多变量指南能够捕获更多真实的后验信息。
[14]:
predictive = Predictive(model, guide=guide, num_samples=num_samples)
svi_mvn_samples = {k: v.reshape(num_samples).detach().cpu().numpy()
for k, v in predictive(log_gdp, is_cont_africa, ruggedness).items()
if k != "obs"}
fig, axs = plt.subplots(nrows=2, ncols=2, figsize=(12, 10))
fig.suptitle("Marginal Posterior density - Regression Coefficients", fontsize=16)
for i, ax in enumerate(axs.reshape(-1)):
site = sites[i]
sns.distplot(svi_mvn_samples[site], ax=ax, label="SVI (Multivariate Normal)")
sns.distplot(hmc_samples[site], ax=ax, label="HMC")
ax.set_title(site)
handles, labels = ax.get_legend_handles_labels()
fig.legend(handles, labels, loc='upper right');

现在让我们比较对角法线引导和多元法线引导计算的后验。请注意,多元分布比对角正态分布更不均匀。
[15]:
fig, axs = plt.subplots(nrows=1, ncols=2, figsize=(12, 6))
fig.suptitle("Cross-sections of the Posterior Distribution", fontsize=16)
sns.kdeplot(x=svi_samples["bA"], y=svi_samples["bR"], ax=axs[0], label="SVI (Diagonal Normal)")
sns.kdeplot(x=svi_mvn_samples["bA"], y=svi_mvn_samples["bR"], ax=axs[0], shade=True, label="SVI (Multivariate Normal)")
axs[0].set(xlabel="bA", ylabel="bR", xlim=(-2.5, -1.2), ylim=(-0.5, 0.1))
sns.kdeplot(x=svi_samples["bR"], y=svi_samples["bAR"], ax=axs[1], label="SVI (Diagonal Normal)")
sns.kdeplot(x=svi_mvn_samples["bR"], y=svi_mvn_samples["bAR"], ax=axs[1], shade=True, label="SVI (Multivariate Normal)")
axs[1].set(xlabel="bR", ylabel="bAR", xlim=(-0.45, 0.05), ylim=(-0.15, 0.8))
handles, labels = axs[1].get_legend_handles_labels()
fig.legend(handles, labels, loc='upper right');

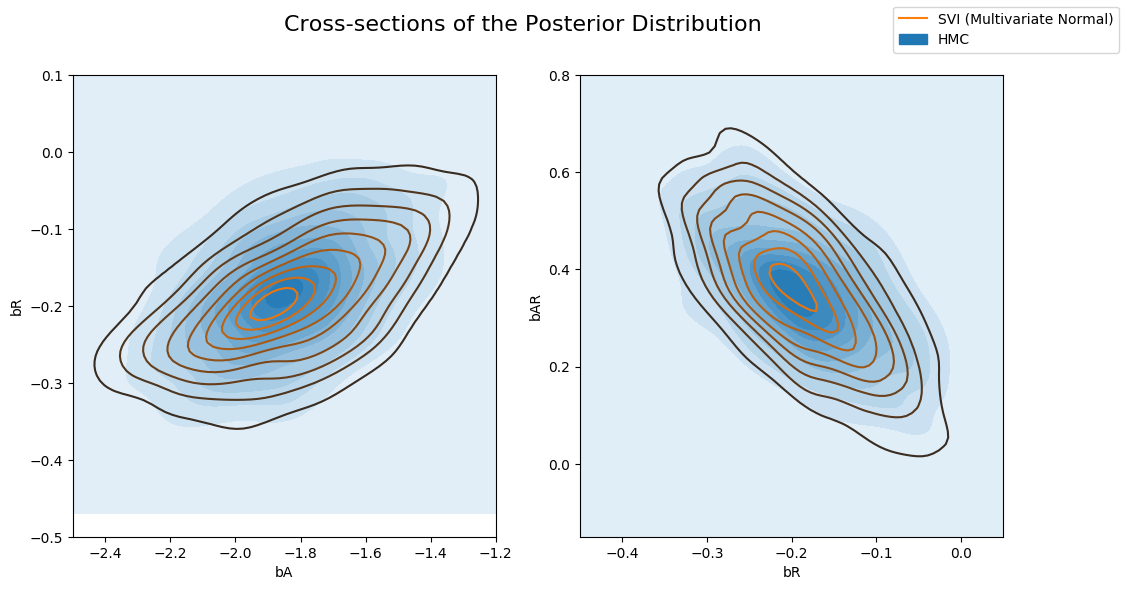
以及由 HMC 计算后验的多变量指南。请注意,多变量指南可以更好地捕获真实的后验。
[16]:
fig, axs = plt.subplots(nrows=1, ncols=2, figsize=(12, 6))
fig.suptitle("Cross-sections of the Posterior Distribution", fontsize=16)
sns.kdeplot(x=hmc_samples["bA"], y=hmc_samples["bR"], ax=axs[0], shade=True, label="HMC")
sns.kdeplot(x=svi_mvn_samples["bA"], y=svi_mvn_samples["bR"], ax=axs[0], label="SVI (Multivariate Normal)")
axs[0].set(xlabel="bA", ylabel="bR", xlim=(-2.5, -1.2), ylim=(-0.5, 0.1))
sns.kdeplot(x=hmc_samples["bR"], y=hmc_samples["bAR"], ax=axs[1], shade=True, label="HMC")
sns.kdeplot(x=svi_mvn_samples["bR"], y=svi_mvn_samples["bAR"], ax=axs[1], label="SVI (Multivariate Normal)")
axs[1].set(xlabel="bR", ylabel="bAR", xlim=(-0.45, 0.05), ylim=(-0.15, 0.8))
handles, labels = axs[1].get_legend_handles_labels()
fig.legend(handles, labels, loc='upper right');
参考
[1] 霍夫曼、马修·D.和安德鲁·格尔曼。“禁止掉头采样器:在哈密顿蒙特卡罗中自适应设置路径长度。” 机器学习研究杂志 15.1(2014):1593-1623。https://arxiv.org/abs/1111.4246。