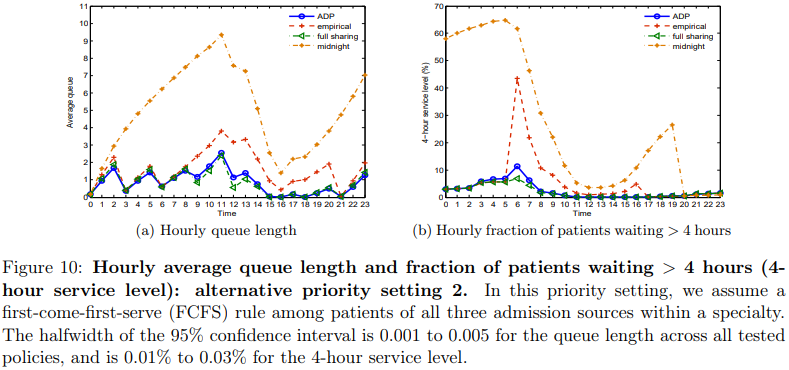
由于之前哔站作者整理的LUNA16数据处理方式过于的繁琐,于是,本文就对LUNA16数据做一个新的整理,最终得到的数据和形式是差不多的。但是,主要不同的是代码逻辑比较的简单,便于理解。
对于数据集的学习,可以去参考这里:【LIDC-IDRI】 CT 肺结节 XML 标记特征良恶性标签PKL转储(一)
步骤和中心内容,包括一下几个部分:
masks生成:从xml文件中,抽取出对应序列series的结节标记位置坐标(可能一个结节多人多次标注),生成对应的mask数组文件,大小与图像数组大小一致;- 肺实质提取操作:从肺区分割的数据中,与原始图像和
mask图做乘积操作,非肺区部分进行填充,或者去除操作均可; resample操作:根据spacing,进行resample操作,可以在zyx三个维度进行resample,也可以仅仅在z方向进行resample操作位1mm(这个我在论文中看到有类似这样做的);- 根据
mask,获取结节的zyx中心点坐标,和半径。
至此,我们将收获以下几个文件:
- 包含
ct的图像数据; - 对应的
mask数据; - 记录
zyx中心点坐标,和半径的文件。
相比于luna16给出的数据形式,目前的数据就比较好理解,和方便查看了。无论是可视化,还是后续的数据处理和训练,都更加的直观、明了。后面就会针对这部分,一一进行展开。
由于代码量还是比较大,处理的东西,和涉及的文件比较多,可能会几个篇章展开。本篇就先对xml文件进行处理,转出来,以便于查看。这里涉及到xml文件的格式,和处理,就单独开一篇,链接去参考:【医学影像数据处理】 XML 文件格式处理汇总
一、xml文件转储
1.1、认识标注文件xml
对于LIDC-IDRI数据集中,xml文件内各个字段表示什么意思的介绍,可以参考我的另一篇文章,点击这里:【LIDC-IDRI】 CT 肺结节 XML 标记特征良恶性标签PKL转储(一)
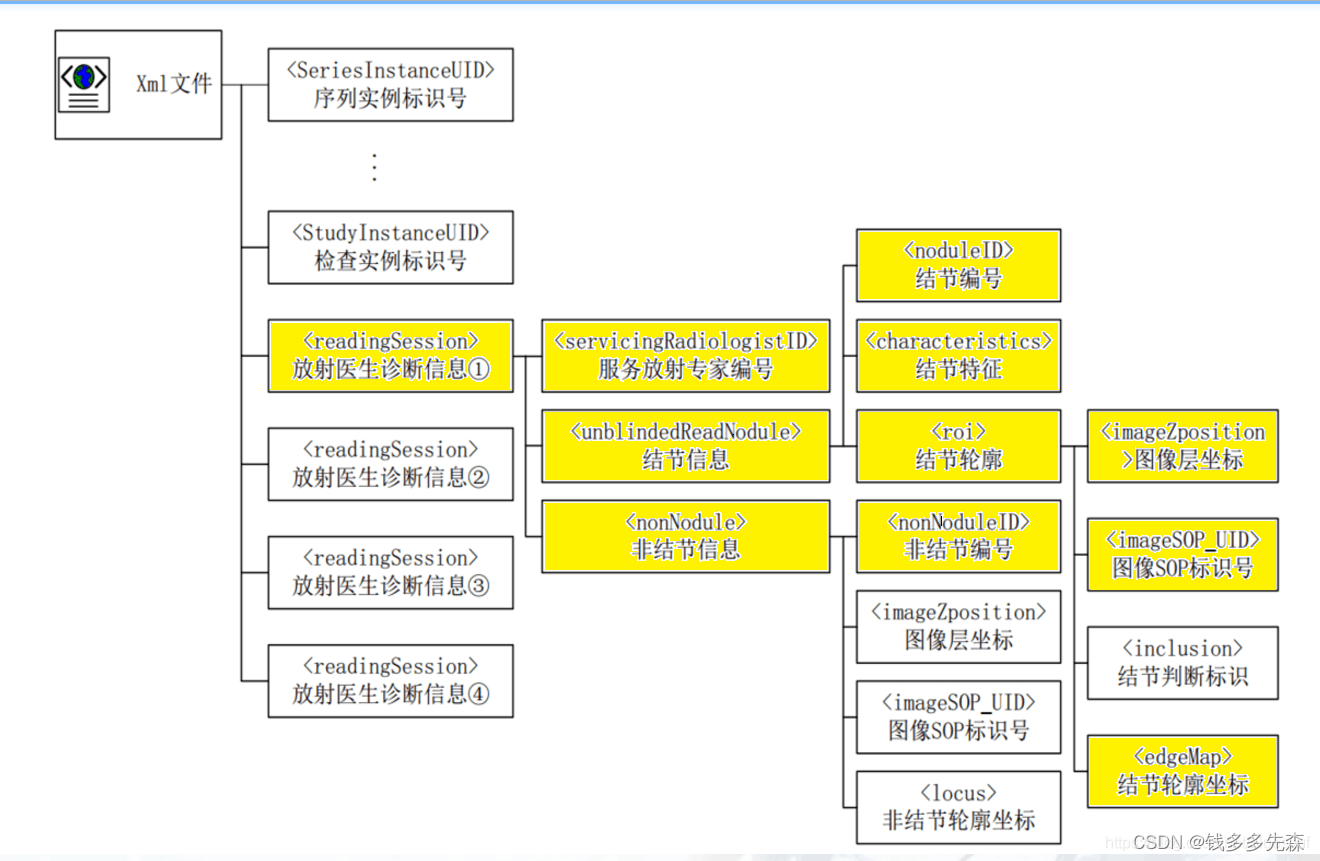
在这篇文章里面,着重介绍了这个数据的结构,以及xml各个记录的tag是什么意思。相信你看完,对这个数据集的处理,有更深的理解。
其中大部分代码都是跟上面这个链接介绍和获取的内容是一样的,可以参考这个GitHub:NoduleNet - utils -LIDC
有些内容没有介绍到,简单做个补充
ResponseHeader:这个是头部分,记录了这个病例(也就是单个病人的CT图像)的信息。
为了方便查看,和学习xml文件,可以参考这篇文章:【医学影像数据处理】 XML 文件格式处理汇总。我们就采用其中xml转字典的形式,方便我们查看。下面就展示了转字典后的前后部分内容对比,如下:
原始xml的数据形式,节选了其中的一小段,展示如下:
<?xml version="1.0" encoding="UTF-8"?>
<LidcReadMessage uid="1.3.6.1.4.1.14519.5.2.1.6279.6001.1308168927505.0" xmlns="http://www.nih.gov" xmlns:xsi="http://www.w3.org/2001/XMLSchema-instance" xsi:schemaLocation="http://www.nih.gov http://troll.rad.med.umich.edu/lidc/LidcReadMessage.xsd">
<ResponseHeader>
<Version>1.7</Version>
<MessageId>1148851</MessageId>
<DateRequest>2005-11-03</DateRequest>
<TimeRequest>12:25:10</TimeRequest>
<RequestingSite>removed</RequestingSite>
<ServicingSite>removed</ServicingSite>
<TaskDescription>Second unblinded read</TaskDescription>
<CtImageFile>removed</CtImageFile>
<SeriesInstanceUid>1.3.6.1.4.1.14519.5.2.1.6279.6001.131939324905446238286154504249</SeriesInstanceUid>
<StudyInstanceUID>1.3.6.1.4.1.14519.5.2.1.6279.6001.303241414168367763244410429787</StudyInstanceUID>
<DateService>2005-11-03</DateService>
<TimeService>12:25:40</TimeService>
<ResponseDescription>1 - Reading complete</ResponseDescription>
<ResponseComments></ResponseComments>
</ResponseHeader>
转换成dictionary字典后的形式。(更便于查看了)
{
"LidcReadMessage": {
"@uid": "1.3.6.1.4.1.14519.5.2.1.6279.6001.1308168927505.0",
"@xmlns": "http://www.nih.gov",
"@xmlns:xsi": "http://www.w3.org/2001/XMLSchema-instance",
"@xsi:schemaLocation": "http://www.nih.gov http://troll.rad.med.umich.edu/lidc/LidcReadMessage.xsd",
"ResponseHeader": {
"Version": "1.7",
"MessageId": "1148851",
"DateRequest": "2005-11-03",
"TimeRequest": "12:25:10",
"RequestingSite": "removed",
"ServicingSite": "removed",
"TaskDescription": "Second unblinded read",
"CtImageFile": "removed",
"SeriesInstanceUid": "1.3.6.1.4.1.14519.5.2.1.6279.6001.131939324905446238286154504249",
"StudyInstanceUID": "1.3.6.1.4.1.14519.5.2.1.6279.6001.303241414168367763244410429787",
"DateService": "2005-11-03",
"TimeService": "12:25:40",
"ResponseDescription": "1 - Reading complete",
"ResponseComments": null
},
}
1.2、xml综合记录转为按series的npy文件
LIDC-IDRI有1018个检查,在标记文件夹tcia-lidc-xml中6 个文件夹中,有1318 个xml文件。并且,这些xml文件的名称,和图像的序列名称不是一一对应的。
所以,就需要现将xml文件内标注的信息,给重新整理出来,转为人能轻易看懂和理解的内容。并且,标注文件如果能与图像文件是一一对应的,那么后续的处理也会方便了许多。
这一小节做的事情,就是将xml文件,给抽取出来,留下关心的内容,其他不重要的,不关心的内容暂时不管。
下面是处理的代码,主要的步骤如下概述:
- 遍历所有的
xml文件,一一处理; - 对单个
xml文件,解析出seriesuid和标注的结节坐标; - 存储到以
seriesuid命名的npy文件,存储的内容就是一个个结节坐标。
完整代码如下:
from tqdm import tqdm
import sys
import os
import numpy as np
from pylung.utils import find_all_files
from pylung.annotation import parse
def xml2mask(xml_file):
header, annos = parse(xml_file) # get one xml info
ctr_arrs = []
for i, reader in enumerate(annos):
for j, nodule in enumerate(reader.nodules):
ctr_arr = []
for k, roi in enumerate(nodule.rois):
z = roi.z
for roi_xy in roi.roi_xy:
ctr_arr.append([z, roi_xy[1], roi_xy[0]]) # [[[z, y, x], [z, y, x]]]
ctr_arrs.append(ctr_arr)
seriesuid = header.series_instance_uid
return seriesuid, ctr_arrs
def annotation2masks(annos_dir, save_dir):
# get all xml file path
files = find_all_files(annos_dir, '.xml')
for f in tqdm(files, total=len(files)):
print(f)
try:
seriesuid, masks = xml2mask(f)
np.save(os.path.join(save_dir, '%s' % (seriesuid)), masks) # save xml 3D coor [[z, y, x], [z, y, x]]
except:
print("Unexpected error:", sys.exc_info()[0])
if __name__ == '__main__':
annos_dir = './LUNA16/annotation/LIDC-XML-only/tcia-lidc-xml' # .xml
ctr_arr_save_dir = './LUNA16/annotation/noduleCoor' # 保存每个注释器解析的中间结节mask的地方
os.makedirs(ctr_arr_save_dir, exist_ok=True)
# xml信息,转储npy(临时文件)
annotation2masks(annos_dir, ctr_arr_save_dir)
下面打开一个·npy·文件进行查看,记录的内容如下,是所有医生对这个序列标注的所有结节的polygon坐标点:
[list([[-299.8, 206, 42], [-299.8, 207, 41], [-299.8, 208, 41], [-299.8, 209, 40], [-299.8, 210, 40], [-299.8, 211, 41], [-299.8, 212, 41], [-299.8, 213, 42], [-299.8, 214, 42], [-299.8, 215, 43], [-299.8, 216, 44], [-299.8, 216, 45], [-299.8, 215, 46], [-299.8, 215, 47], [-299.8, 215, 48], [-299.8, 214, 49], [-299.8, 213, 49], [-299.8, 212, 49], [-299.8, 211, 49], [-299.8, 210, 49], [-299.8, 209, 49], [-299.8, 208, 48], [-299.8, 207, 47], [-299.8, 207, 46], [-299.8, 206, 45], [-299.8, 206, 44], [-299.8, 206, 43], [-299.8, 206, 42], [-298.0, 206, 46], [-298.0, 207, 45], [-298.0, 207, 44], [-298.0, 208, 43], [-298.0, 209, 42], [-298.0, 209, 41], [-298.0, 210, 40], [-298.0, 211, 40], [-298.0, 212, 39], [-298.0, 213, 40], [-298.0, 214, 41], [-298.0, 215, 42], [-298.0, 215, 43], [-298.0, 216, 44], [-298.0, 216, 45], [-298.0, 216, 46], [-298.0, 216, 47], [-298.0, 215, 48], [-298.0, 214, 48], [-298.0, 213, 48], [-298.0, 212, 48], [-298.0, 211, 48], [-298.0, 210, 48], [-298.0, 209, 48], [-298.0, 208, 48], [-298.0, 207, 47], [-298.0, 206, 46], [-296.2, 209, 42], [-296.2, 210, 41], [-296.2, 211, 40], [-296.2, 212, 40], [-296.2, 213, 41], [-296.2, 214, 42], [-296.2, 215, 43], [-296.2, 216, 44], [-296.2, 216, 45], [-296.2, 216, 46], [-296.2, 216, 47], [-296.2, 216, 48], [-296.2, 215, 49], [-296.2, 214, 49], [-296.2, 213, 49], [-296.2, 212, 49], [-296.2, 211, 48], [-296.2, 210, 47], [-296.2, 209, 46], [-296.2, 209, 45], [-296.2, 209, 44], [-296.2, 209, 43], [-296.2, 209, 42]])
list([[-227.8, 151, 405], [-227.8, 152, 404], [-227.8, 153, 403], [-227.8, 154, 402], [-227.8, 155, 402], [-227.8, 156, 402], [-227.8, 157, 403], [-227.8, 157, 404], [-227.8, 157, 405], [-227.8, 158, 406], [-227.8, 158, 407], [-227.8, 158, 408], [-227.8, 157, 409], [-227.8, 156, 409], [-227.8, 155, 409], [-227.8, 154, 408], [-227.8, 153, 408], [-227.8, 152, 407], [-227.8, 151, 406], [-227.8, 151, 405], [-226.0, 152, 405], [-226.0, 153, 404], [-226.0, 154, 404], [-226.0, 155, 403], [-226.0, 156, 404], [-226.0, 157, 405], [-226.0, 157, 406], [-226.0, 157, 407], [-226.0, 156, 408], [-226.0, 155, 408], [-226.0, 154, 408], [-226.0, 153, 408], [-226.0, 152, 407], [-226.0, 152, 406], [-226.0, 152, 405]])
list([[-226.0, 158, 407], [-226.0, 157, 408], [-226.0, 156, 409], [-226.0, 155, 409], [-226.0, 154, 409], [-226.0, 153, 409], [-226.0, 152, 408], [-226.0, 151, 407], [-226.0, 152, 406], [-226.0, 153, 405], [-226.0, 153, 404], [-226.0, 154, 403], [-226.0, 155, 402], [-226.0, 156, 402], [-226.0, 157, 403], [-226.0, 158, 404], [-226.0, 158, 405], [-226.0, 158, 406], [-226.0, 158, 407], [-227.8, 159, 407], [-227.8, 158, 408], [-227.8, 157, 409], [-227.8, 156, 410], [-227.8, 155, 410], [-227.8, 154, 410], [-227.8, 153, 409], [-227.8, 152, 408], [-227.8, 151, 407], [-227.8, 151, 406], [-227.8, 151, 405], [-227.8, 152, 404], [-227.8, 153, 403], [-227.8, 154, 402], [-227.8, 155, 402], [-227.8, 156, 402], [-227.8, 157, 403], [-227.8, 158, 404], [-227.8, 158, 405], [-227.8, 158, 406], [-227.8, 159, 407]])
list([[-296.2, 214, 46], [-296.2, 213, 47], [-296.2, 212, 47], [-296.2, 211, 47], [-296.2, 210, 46], [-296.2, 209, 45], [-296.2, 208, 44], [-296.2, 208, 43], [-296.2, 208, 42], [-296.2, 209, 41], [-296.2, 210, 42], [-296.2, 211, 42], [-296.2, 212, 43], [-296.2, 213, 44], [-296.2, 214, 45], [-296.2, 214, 46], [-298.0, 216, 47], [-298.0, 215, 48], [-298.0, 214, 49], [-298.0, 213, 49], [-298.0, 212, 49], [-298.0, 211, 49], [-298.0, 210, 49], [-298.0, 209, 48], [-298.0, 208, 47], [-298.0, 207, 46], [-298.0, 207, 45], [-298.0, 207, 44], [-298.0, 208, 43], [-298.0, 208, 42], [-298.0, 209, 41], [-298.0, 210, 41], [-298.0, 211, 41], [-298.0, 212, 41], [-298.0, 213, 41], [-298.0, 214, 42], [-298.0, 215, 43], [-298.0, 216, 44], [-298.0, 216, 45], [-298.0, 216, 46], [-298.0, 216, 47], [-299.8, 216, 50], [-299.8, 215, 51], [-299.8, 214, 51], [-299.8, 213, 50], [-299.8, 212, 50], [-299.8, 211, 50], [-299.8, 210, 49], [-299.8, 209, 48], [-299.8, 208, 47], [-299.8, 207, 46], [-299.8, 207, 45], [-299.8, 207, 44], [-299.8, 208, 43], [-299.8, 209, 42], [-299.8, 210, 42], [-299.8, 211, 41], [-299.8, 212, 41], [-299.8, 213, 42], [-299.8, 214, 42], [-299.8, 215, 43], [-299.8, 216, 44], [-299.8, 216, 45], [-299.8, 216, 46], [-299.8, 216, 47], [-299.8, 216, 48], [-299.8, 216, 49], [-299.8, 216, 50]])
list([[-226.0, 158, 407], [-226.0, 157, 408], [-226.0, 156, 409], [-226.0, 155, 409], [-226.0, 154, 409], [-226.0, 153, 409], [-226.0, 152, 409], [-226.0, 151, 409], [-226.0, 151, 408], [-226.0, 151, 407], [-226.0, 151, 406], [-226.0, 151, 405], [-226.0, 152, 404], [-226.0, 152, 403], [-226.0, 153, 403], [-226.0, 154, 402], [-226.0, 154, 401], [-226.0, 155, 401], [-226.0, 156, 401], [-226.0, 157, 401], [-226.0, 157, 402], [-226.0, 158, 403], [-226.0, 158, 404], [-226.0, 158, 405], [-226.0, 158, 406], [-226.0, 158, 407], [-227.8, 159, 407], [-227.8, 158, 408], [-227.8, 158, 409], [-227.8, 157, 409], [-227.8, 156, 410], [-227.8, 155, 410], [-227.8, 154, 409], [-227.8, 153, 409], [-227.8, 152, 409], [-227.8, 151, 408], [-227.8, 151, 407], [-227.8, 151, 406], [-227.8, 151, 405], [-227.8, 151, 404], [-227.8, 152, 403], [-227.8, 152, 402], [-227.8, 153, 401], [-227.8, 154, 401], [-227.8, 155, 401], [-227.8, 156, 401], [-227.8, 157, 401], [-227.8, 158, 402], [-227.8, 158, 403], [-227.8, 159, 404], [-227.8, 159, 405], [-227.8, 159, 406], [-227.8, 159, 407]])
list([[-296.2, 215, 47], [-296.2, 214, 48], [-296.2, 213, 48], [-296.2, 212, 48], [-296.2, 211, 48], [-296.2, 210, 47], [-296.2, 209, 47], [-296.2, 208, 46], [-296.2, 208, 45], [-296.2, 207, 44], [-296.2, 207, 43], [-296.2, 208, 42], [-296.2, 209, 42], [-296.2, 210, 42], [-296.2, 211, 42], [-296.2, 212, 43], [-296.2, 213, 43], [-296.2, 214, 44], [-296.2, 215, 45], [-296.2, 215, 46], [-296.2, 215, 47], [-298.0, 216, 47], [-298.0, 215, 48], [-298.0, 214, 49], [-298.0, 214, 50], [-298.0, 213, 50], [-298.0, 212, 50], [-298.0, 211, 49], [-298.0, 210, 49], [-298.0, 209, 48], [-298.0, 208, 48], [-298.0, 207, 47], [-298.0, 207, 46], [-298.0, 207, 45], [-298.0, 207, 44], [-298.0, 207, 43], [-298.0, 207, 42], [-298.0, 207, 41], [-298.0, 208, 41], [-298.0, 209, 41], [-298.0, 210, 41], [-298.0, 211, 41], [-298.0, 212, 41], [-298.0, 213, 41], [-298.0, 214, 41], [-298.0, 215, 42], [-298.0, 215, 43], [-298.0, 216, 44], [-298.0, 216, 45], [-298.0, 216, 46], [-298.0, 216, 47], [-299.8, 217, 46], [-299.8, 216, 47], [-299.8, 216, 48], [-299.8, 215, 49], [-299.8, 214, 50], [-299.8, 213, 50], [-299.8, 212, 50], [-299.8, 211, 50], [-299.8, 210, 50], [-299.8, 209, 49], [-299.8, 208, 48], [-299.8, 208, 47], [-299.8, 207, 46], [-299.8, 207, 45], [-299.8, 207, 44], [-299.8, 208, 43], [-299.8, 209, 42], [-299.8, 209, 41], [-299.8, 210, 41], [-299.8, 211, 41], [-299.8, 212, 41], [-299.8, 213, 41], [-299.8, 214, 42], [-299.8, 215, 42], [-299.8, 215, 43], [-299.8, 216, 44], [-299.8, 217, 45], [-299.8, 217, 46], [-301.6, 214, 45], [-301.6, 213, 46], [-301.6, 212, 47], [-301.6, 211, 47], [-301.6, 210, 46], [-301.6, 209, 45], [-301.6, 210, 44], [-301.6, 211, 43], [-301.6, 212, 43], [-301.6, 213, 44], [-301.6, 214, 45]])
list([[-296.2, 209, 43], [-296.2, 209, 44], [-296.2, 210, 45], [-296.2, 211, 46], [-296.2, 212, 47], [-296.2, 212, 48], [-296.2, 213, 48], [-296.2, 214, 48], [-296.2, 215, 47], [-296.2, 215, 46], [-296.2, 215, 45], [-296.2, 214, 44], [-296.2, 213, 43], [-296.2, 212, 43], [-296.2, 211, 43], [-296.2, 210, 43], [-296.2, 209, 43], [-298.0, 208, 42], [-298.0, 208, 43], [-298.0, 208, 44], [-298.0, 208, 45], [-298.0, 208, 46], [-298.0, 208, 47], [-298.0, 209, 47], [-298.0, 210, 48], [-298.0, 211, 48], [-298.0, 211, 49], [-298.0, 212, 49], [-298.0, 213, 48], [-298.0, 214, 48], [-298.0, 215, 47], [-298.0, 216, 46], [-298.0, 216, 45], [-298.0, 216, 44], [-298.0, 215, 43], [-298.0, 214, 43], [-298.0, 213, 42], [-298.0, 212, 42], [-298.0, 212, 41], [-298.0, 211, 41], [-298.0, 210, 41], [-298.0, 209, 42], [-298.0, 208, 42], [-299.8, 210, 43], [-299.8, 209, 43], [-299.8, 208, 44], [-299.8, 207, 44], [-299.8, 207, 45], [-299.8, 207, 46], [-299.8, 208, 47], [-299.8, 209, 48], [-299.8, 210, 49], [-299.8, 211, 49], [-299.8, 212, 49], [-299.8, 213, 50], [-299.8, 214, 49], [-299.8, 215, 48], [-299.8, 215, 47], [-299.8, 216, 46], [-299.8, 216, 45], [-299.8, 215, 44], [-299.8, 215, 43], [-299.8, 214, 43], [-299.8, 214, 42], [-299.8, 213, 42], [-299.8, 212, 41], [-299.8, 211, 41], [-299.8, 210, 42], [-299.8, 210, 43]])] <class 'numpy.ndarray'>
二、标记次数和mask数组生成
生成npy文件并不是此次标注信息的最终结果,有以下几个原因:
xml文件内标注的结节坐标是多个医生分别标注的,所以会存在标注上的重叠(也就是一个结节被多个医生重复标注,很多是背靠背标注,也不知道其他医生标注了什么)。所以需要对多人标注的内容进行处理,留下最终的结节坐标;- 只是坐标点,还需要生成和
image一样shape,相互对应的mask文件。
根据上面几个原因,生成最终mask文件,就需要经历以下几个步骤:
- 标记的结节坐标点,需要将
hu z到instanceNum处理,对应的图像上; - 对多个医生标注的结节,进行处理,根据
iou重叠规则,留下最终的结节; - 留下的结节坐标,绘制到
mask上,存储下来。
实现代码如下:
import nrrd
import SimpleITK as sitk
import cv2
import os
import numpy as np
def load_itk_image(filename):
"""
Return img array and [z,y,x]-ordered origin and spacing
"""
# sitk.ReadImage返回的image的shape是x、y、z
itkimage = sitk.ReadImage(filename)
numpyImage = sitk.GetArrayFromImage(itkimage)
numpyOrigin = np.array(list(reversed(itkimage.GetOrigin())))
numpySpacing = np.array(list(reversed(itkimage.GetSpacing())))
return numpyImage, numpyOrigin, numpySpacing
def arrs2mask(img_dir, ctr_arr_dir, save_dir):
cnt = 0
consensus = {1: 0, 2: 0, 3: 0, 4: 0} # 一致意见
# generate save document
for k in consensus.keys():
if not os.path.exists(os.path.join(save_dir, str(k))):
os.makedirs(os.path.join(save_dir, str(k)))
for f in os.listdir(img_dir):
if f.endswith('.mhd'):
pid = f[:-4]
print('pid:', pid)
# ct
img, origin, spacing = load_itk_image(os.path.join(img_dir, '%s.mhd' % (pid)))
# mask coor npy
ctr_arrs = np.load(os.path.join(ctr_arr_dir, '%s.npy' % (pid)), allow_pickle=True)
cnt += len(ctr_arrs)
nodule_masks = []
# 依次标注结节处理
for ctr_arr in ctr_arrs:
z_origin = origin[0]
z_spacing = spacing[0]
ctr_arr = np.array(ctr_arr)
# ctr_arr[:, 0] z轴方向值,由hu z到instanceNum [-50, -40, -30]-->[2, 3, 4]
ctr_arr[:, 0] = np.absolute(ctr_arr[:, 0] - z_origin) / z_spacing # 对数组中的每一个元素求其绝对值。np.abs是这个函数的简写
ctr_arr = ctr_arr.astype(np.int32)
print(ctr_arr)
# 每一个标注的结节,都会新临时生成一个与img一样大小的mask文件
mask = np.zeros(img.shape)
# 遍历标注层的 z 轴序列
for z in np.unique(ctr_arr[:, 0]): # 去除其中重复的元素 ,并按元素 由小到大排序
ctr = ctr_arr[ctr_arr[:, 0] == z][:, [2, 1]]
ctr = np.array([ctr], dtype=np.int32)
mask[z] = cv2.fillPoly(mask[z], ctr, color=(1,))
nodule_masks.append(mask)
i = 0
visited = []
d = {}
masks = []
while i < len(nodule_masks):
# If mached before, then no need to create new mask
if i in visited:
i += 1
continue
same_nodules = []
mask1 = nodule_masks[i]
same_nodules.append(mask1)
d[i] = {}
d[i]['count'] = 1
d[i]['iou'] = []
# Find annotations pointing to the same nodule
# 当前结节mask[i],与其后面的所有结节,依次求iou
for j in range(i + 1, len(nodule_masks)):
# if not overlapped with previous added nodules
if j in visited:
continue
mask2 = nodule_masks[j]
iou = float(np.logical_and(mask1, mask2).sum()) / np.logical_or(mask1, mask2).sum()
# 如果iou超过阈值,则当前第i个mask记为被重复标记一次
if iou > 0.4:
visited.append(j)
same_nodules.append(mask2)
d[i]['count'] += 1
d[i]['iou'].append(iou)
masks.append(same_nodules)
i += 1
print(visited)
exit()
# only 4 people, check up 4 data
for k, v in d.items():
if v['count'] > 4:
print('WARNING: %s: %dth nodule, iou: %s' % (pid, k, str(v['iou'])))
v['count'] = 4
consensus[v['count']] += 1
# number of consensus
num = np.array([len(m) for m in masks])
num[num > 4] = 4 # 最多4次,超过4次重复标记的,记为4次
if len(num) == 0:
continue
# Iterate from the nodules with most consensus
for n in range(num.max(), 0, -1):
mask = np.zeros(img.shape, dtype=np.uint8)
for i, index in enumerate(np.where(num >= n)[0]):
same_nodules = masks[index]
m = np.logical_or.reduce(same_nodules)
mask[m] = i + 1 # 区分不同的结节,不同的结节给与不同的数值,依次增加(如果是分割,可以直接都给1,或者最后统一处理为1也可以)
nrrd.write(os.path.join(save_dir, str(n), pid+'.nrrd'), mask) # mask
print(consensus)
print(cnt)
if __name__ == '__main__':
img_dir = r'./LUNA16/image_combined' # data
ctr_arr_save_dir = r'./LUNA16/annotation/noduleCoor' # 保存每个注释器解析的中间结节mask的地方
noduleMask_save_dir = r'./LUNA16/nodule_masks' # 保存合并结节掩码的文件夹
# 对转储的临时文件,生成mask
arrs2mask(img_dir, ctr_arr_save_dir, noduleMask_save_dir)
至此,和image一样shape的mask是生成了。下面用itk-snap打开查看处理后的结果,如下所示:

属于分别打开image和mask的nrrd图像,mhd格式的image,转nrrd,可以参考下面的代码:
nii_path = os.path.join(r'./LUNA16/image_combined', '1.3.6.1.4.1.14519.5.2.1.6279.6001.184412674007117333405073397832.mhd')
image = itk.array_from_image(itk.imread(nii_path))
nrrd.write(r'./image.nrrd', image)
三、总结
lidc-idri的数据集内的数据格式,都是我们不常遇到的数据形式,尤其是mhd文件的raw文件,同时表示一个数据的两个不同部分,也是很少遇到的。
但是对于初学者来说,理解这种数据形式,还是有些陌生,这部分相信通过本系列可以有较深的理解。与此同时,本篇还存储为nrrd文件,这是我比较喜欢的数组存储格式,理解的好理解和简单。
到这里,你就收获了一个新的一一对应关系。这样比你看xml文件,理解起来会简单很多。下一节,我们就对初步得到的image和mask,与肺区分割结合,进一步进行精细化处理。resample操作,调整数据到统一的尺度。