支持向量机(实战)
目录
- 一、准备工作(设置 jupyter notebook 中的字体大小样式等)
- 二、线性支持向量机(核函数为线性核)
- 三、数据标准化的影响
- 四、软间隔
- 五、非线性支持向量机
- 5.1 手动升维
- 5.2 对比试验:手动升维与核函数的差异
- 方法1:利用 PolynomialFeatures 来对原始数据进行升维,然后再利用线性 SVM 对数据进行训练
- 方法 2:利用核技巧(选用 poly 核)
- 方法 3:利用核技巧(选用高斯核)
- 六、对比试验: 𝛾 和 C 值对 SVM 的影响。
实战部分将结合着 理论部分 进行,旨在帮助理解和强化实操(以下代码将基于 jupyter notebook 进行)。
一、准备工作(设置 jupyter notebook 中的字体大小样式等)
import numpy as np
import os
%matplotlib inline
import matplotlib
import matplotlib.pyplot as plt
plt.rcParams['axes.labelsize'] = 14
plt.rcParams['xtick.labelsize'] = 12
plt.rcParams['ytick.labelsize'] = 12
import warnings
warnings.filterwarnings('ignore')
二、线性支持向量机(核函数为线性核)
下面将通过鸢尾花数据集来展示线性 SVM 的分类效果。
步骤一:从 sklearn 中导入鸢尾花数据集并构建模型
# 导入相关库函数
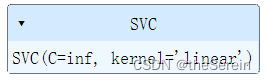
from sklearn.svm import SVC
from sklearn import datasets
# 这里选用鸢尾花数据集作为实验数据集
# 为便于查看分类效果,选用数据集中的两个特征(这样画图时就是一个二维平面,直观上看起来更易于理解)
iris = datasets.load_iris()
X = iris['data'][:,(2,3)]
y = iris['target']
# 设置数据的特征和标签
setosa_or_versicolor = (y==0) | (y==1)
X = X[setosa_or_versicolor]
y = y[setosa_or_versicolor]
# 先做一个线性支持向量机模型(不对原数据集做任何特征变换)
# C 值是用于控制过拟合的,由于这里的数据集比较简单,故此不考虑这个参数,因此将其设置为无穷大
svm_clf = SVC(kernel='linear',C=float('inf'))
# 训练模型
svm_clf.fit(X,y)
步骤二:可视化展示分类结果
# 下面进行画图展示
# 这里设置三条线,作为传统模型的分类结果(以作对比)
x0 = np.linspace(0,5.5,200)
pred_1 = 5*x0 - 20
pred_2 = x0 - 1.8
pred_3 = 0.1*x0 + 0.5
# 定义一个绘制决策边界的函数(注:该函数仅适用于特征数为 2 的数据集)
def plot_svc_decision_boundary(svm_clf,xmin,xmax,sv = True):
# 通过分类器获取权重参数
w = svm_clf.coef_[0]
# 通过分类器获取偏置参数
b = svm_clf.intercept_[0]
# 获取待绘制边界(在 x0 上)的两个端点值
x0 = np.linspace(xmin,xmax,200)
# 计算划分超平面的方程: w0x0+x1x1+b=0
# 现在已知 x0 的数据,画图还需要 x1 的值
x1 = - w[0]/w[1]*x0 - b/w[1]
# 计算“间隔”
margin = 1/w[1]
# 基于前面的值可得到两条支持向量分别对应的“间隔”边界线
gutter_up = x1 + margin
gutter_down = x1 - margin
# 判断是否展示图中的支持向量
if sv:
# 获取数据集中的支持向量
svs = svm_clf.support_vectors_
# 强调支持向量
plt.scatter(svs[:,0],svs[:,1],s=180,facecolors='#FFAAAA')
# 绘制三条线:支持向量所在决策边界×2、划分超平面
plt.plot(x0, x1,'k-' , linewidth=2)
plt.plot(x0, gutter_up, 'k--', linewidth=2)
plt.plot(x0, gutter_down,'k--', linewidth=2)
# 绘图展示
plt.figure(figsize=(14,4))
# 绘制传统分类算法的决策边界
plt.subplot(121)
plt.plot(X[:,0][y==1],X[:,1][y==1], 'bs')
plt.plot(X[:,0][y==0],X[:,1][y==0], 'ys')
plt.plot(x0, pred_1, 'g--', linewidth=2)
plt.plot(x0, pred_2, 'm-', linewidth=2)
plt.plot(x0, pred_3, 'r-', linewidth=2)
plt.axis([0,5.5,0,2])
# 绘制 SVM 的决策边界
plt.subplot (122)
plot_svc_decision_boundary(svm_clf,0,5.5)
plt.plot(X[:,0][y==1],X[:,1][y==1], 'bs')
plt.plot(X[:,0][y==0],X[:,1][y==0], 'ys')
plt.axis([0,5.5,0,2])
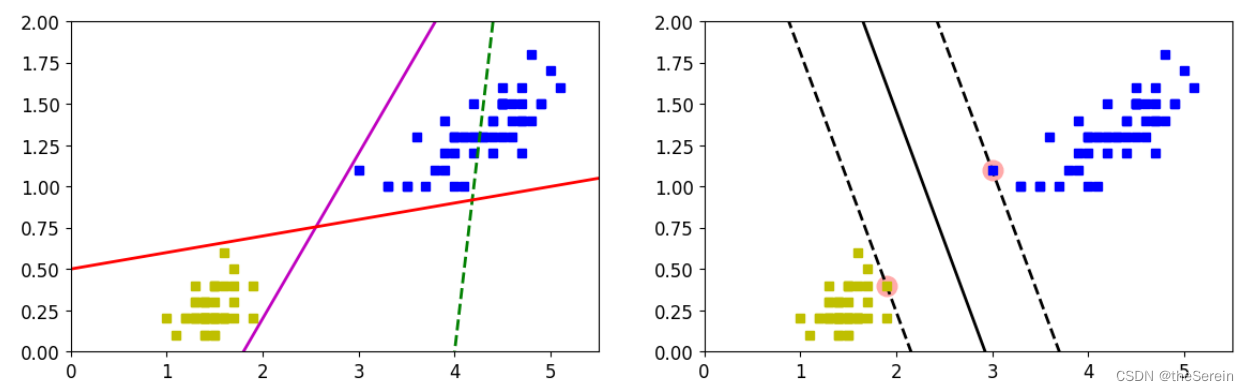
上图展示了用绘制传统分类算法和 SVM 算法的决策边界,很明显,SVM 的效果更好。
三、数据标准化的影响
如果不对数据标准化,可能会导致支持向量间隔非常小(如一些极端的离群点),从而对最终的分类效果产生影响。
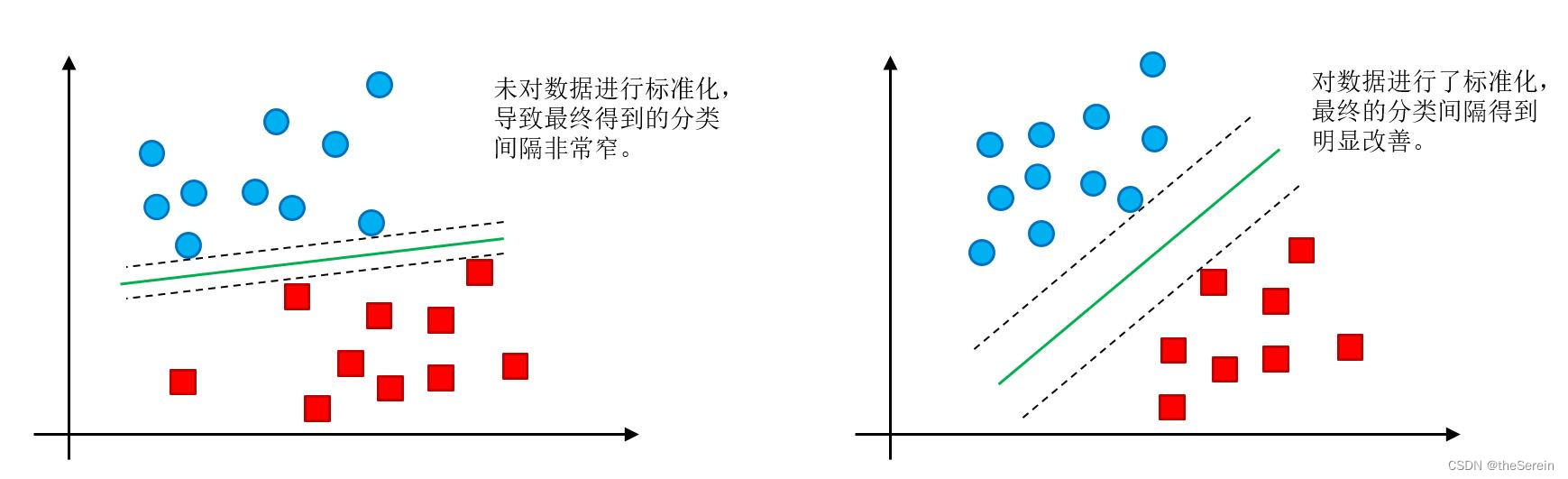
因此,在实验过程中,一定要注意对原数据集进行标准化。
四、软间隔
软间隔的存在,使得可对数据中的一些离群点进行适当放宽,因而改善最终的分类效果。
采取软间隔的两个好处:
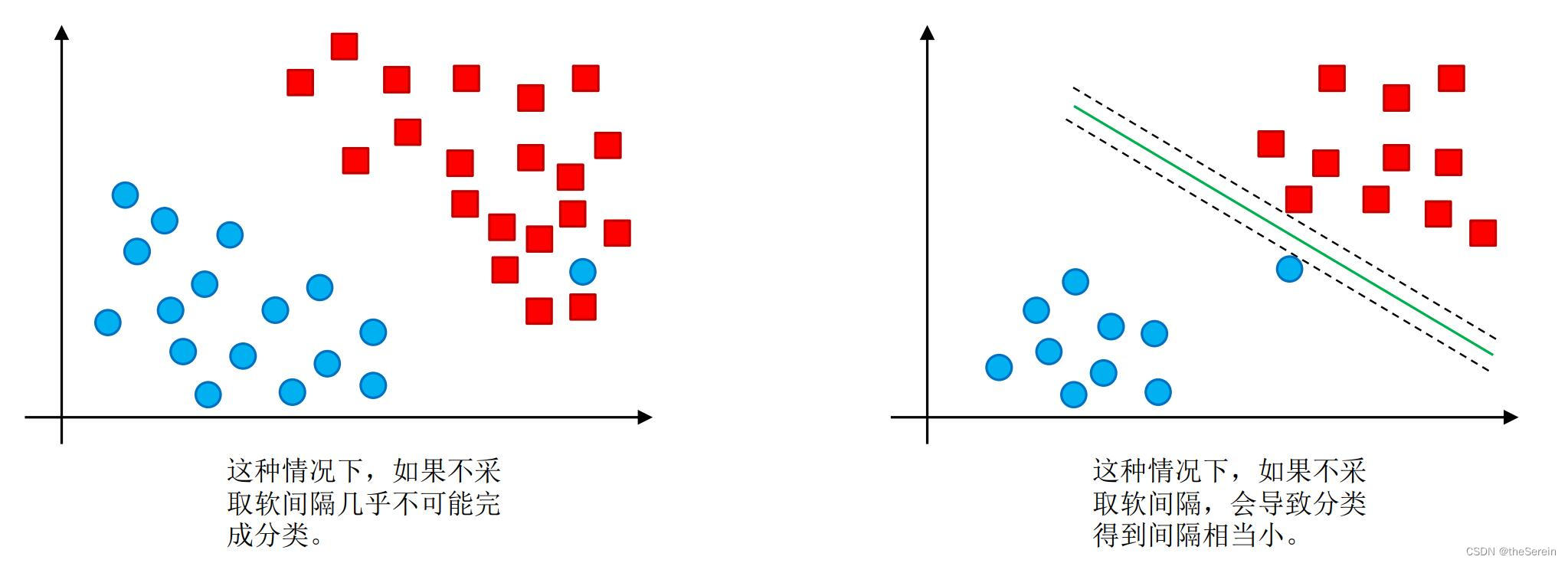
下面通过一个实验,直观地感受软间隔对 SVM 的影响。
# 导入如工具包
import numpy as np
from sklearn import datasets
from sklearn.pipeline import Pipeline
from sklearn.preprocessing import StandardScaler
from sklearn.svm import LinearSVC
# 依然选用鸢尾花数据集(并使用其 2 个特征)
iris=datasets.load_iris()
X = iris["data"][:,(2,3)]
y = (iris["target"] == 2).astype(np.float64)
# 用流水线封装 SVM 训练模型的过程
svm_clf = Pipeline((
('std',StandardScaler()),
('linear_svc',LinearSVC(C = 1))
))
# 训练模型
svm_clf.fit(X,y)

# 执行预测
svm_clf.predict([[4.3,1.4]])
[Out]
array([0.])
对比不同 C 值带来的差异(即控制软间隔的程度)
# 定义一个标准化过程
scaler = StandardScaler()
# 定义两个 C 值不同的分类器
svm_clf1 = LinearSVC(C=1,random_state = 42)
svm_clf2 = LinearSVC(C=1000,random_state = 42)
# 分类进行流水线封装
scaled_svm_clf1 = Pipeline((
('std',StandardScaler()),
('linear_svc',svm_clf1)
))
scaled_svm_clf2 = Pipeline((
('std',StandardScaler()),
('linear_svc',svm_clf2)
))
# 训练数据
scaler.fit(X,y)
scaled_svm_clf1.fit(X,y)
scaled_svm_clf2.fit(X,y)
# 按照前面的方式,分别计算出支持向量所在的决策边界和划分超平面的方程
b1 = svm_clf1.decision_function([-scaler.mean_ / scaler.scale_])
b2 = svm_clf2.decision_function([-scaler.mean_ / scaler.scale_])
w1 = svm_clf1.coef_[0] / scaler.scale_
w2 = svm_clf2.coef_[0] / scaler.scale_
svm_clf1.intercept_ = np.array([b1])
svm_clf2.intercept_ = np.array([b2])
svm_clf1.coef_ = np.array([w1])
svm_clf2.coef_ = np.array([w2])
# 画图展示两个分类器的分类效果
plt.figure(figsize=(14,4.2))
plt.subplot(121)
plt.plot(X[:,0][y==1],X[:,1][y==1], "g^", label="Iris-Virginica")
plt.plot(X[:,0][y==0],X[:,1][y==0],"bs", label="Iris-Versicolor")
plot_svc_decision_boundary(svm_clf1,4,6,sv=False)
plt.xlabel("Petal length", fontsize=14)
plt.ylabel("Petal width", fontsize=14)
plt.legend(loc="upper left", fontsize=14)
plt.title("$C = {}$".format(svm_clf1.C), fontsize=16)
plt.axis([4,6,0.8,2.8])
plt.subplot(122)
plt.plot(X[:,0][y==1],X[:,1][y==1],"g^")
plt.plot(X[:,0][y==0],X[:,1][y==0],"bs")
plot_svc_decision_boundary(svm_clf2,4,6,sv=False)
plt.xlabel("Petal length", fontsize=14)
plt.title("$C ={}$".format(svm_clf2.C),fontsize=16)
plt.axis([4,6,0.8,2.8])
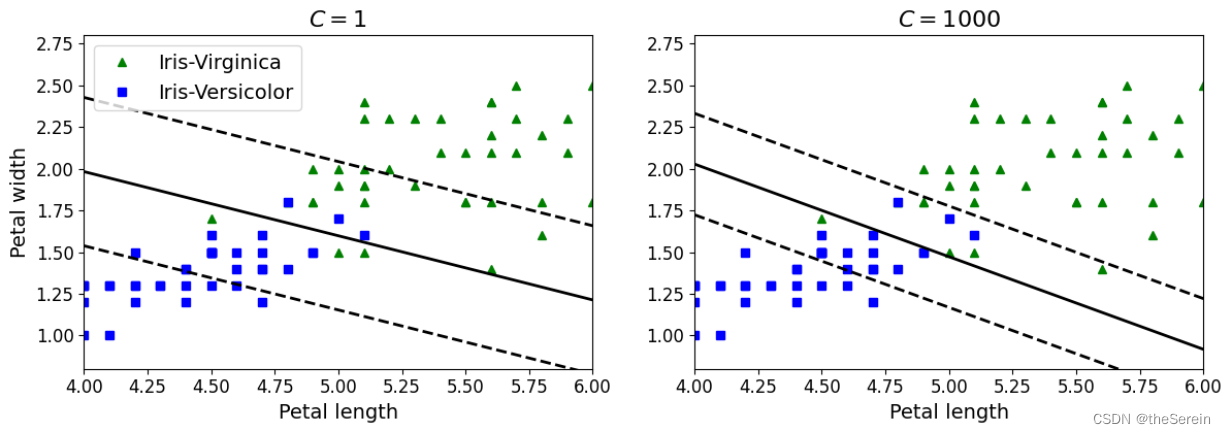
实验结果说明:
- 左图:使用较低的 C 值(软间隔程度更大),因此其分类结果中会有较多数据落在间隔内;
- 左图:使用较高的 C 值(软间隔程度更小),因此其分类结果中会有较少数据落在间隔内(但也容易出现过拟合现象)。
五、非线性支持向量机
主要可以通过两种方式来完成对任务的非线性变换:
- 手动升维(如将原始的 1 维数据变换至 2 维)。
- 采用核函数。
5.1 手动升维
# 构建一个一维数据集
X1D = np.linspace(-4,4,9).reshape(-1,1)
# 指定该数据的标签
y = np. array ([0,0,1,1,1,1, 1,0,0])
# 手动地将其维度提升
# 此时,采用“将原数据对应元素进行平方操作”的方式,来得到原始数据集在第 2 个维度上的特征值
X2D = np.c_[X1D,X1D**2]
# 下面画图展示这些数据在不同维度下的体现
plt.figure(figsize=(11,4))
# 原始数据( 1 维)
plt.subplot(121)
plt.grid(True, which='both')
plt.axhline(y=0,color='k')
plt. plot(X1D[:,0][y==0], np.zeros(4),"bs")
plt. plot(X1D[:,0][y==1], np.zeros(5),"g^")
plt.gca().get_yaxis().set_ticks([])
plt.xlabel(r"$x_1$",fontsize=20)
plt.axis ([-4.5,4.5,-0.2,0.2])
# 进行升维操作后的数据( 2 维)
plt.subplot(122)
plt.grid(True,which='both')
plt.axhline(y=0,color='k')
plt.axvline(x=0, color='k')
plt.plot(X2D[:,0][y==0],X2D[:,1][y==0],"bs")
plt.plot(X2D[:,0][y==1],X2D[:,1][y==1],"g^")
plt.xlabel(r"$x_1$",fontsize=20)
plt.ylabel(r"$x_2$",fontsize=20,rotation=0)
plt.gca().get_yaxis().set_ticks([0,4,8,12,16])
plt.plot([-4.5,4.5],[6.5,6.5],"r--",linewidth=3)
plt.axis([-4.5,4.5,-1,17])
# 指定图像的标题
plt.subplots_adjust(right=1)
# 展示
plt.show()
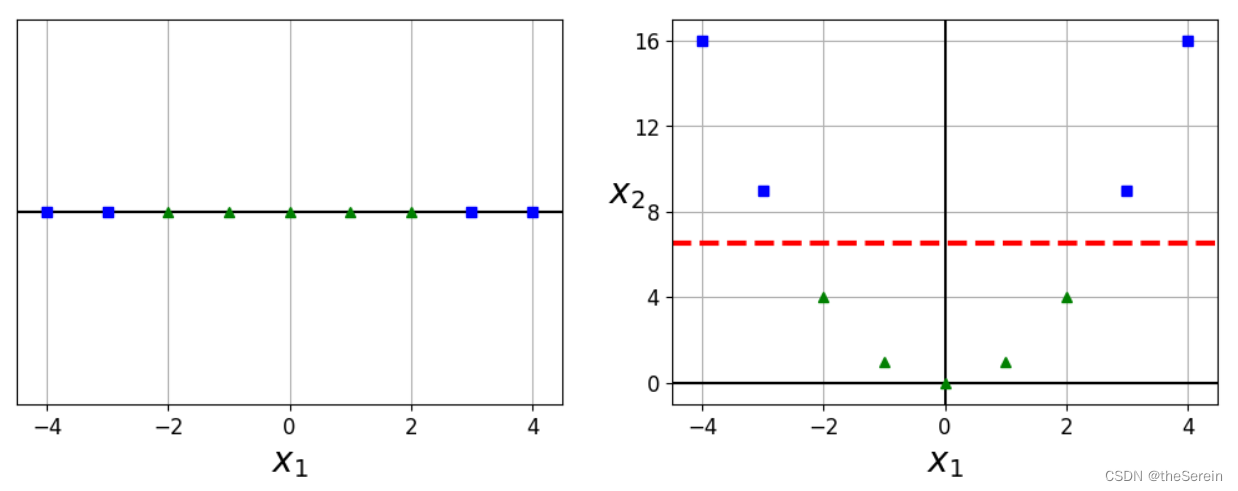
可以很明显地看出,在进行升维操作后,原始数据集变得更容易被分类。
5.2 对比试验:手动升维与核函数的差异
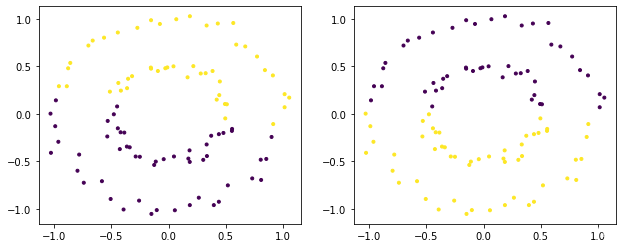
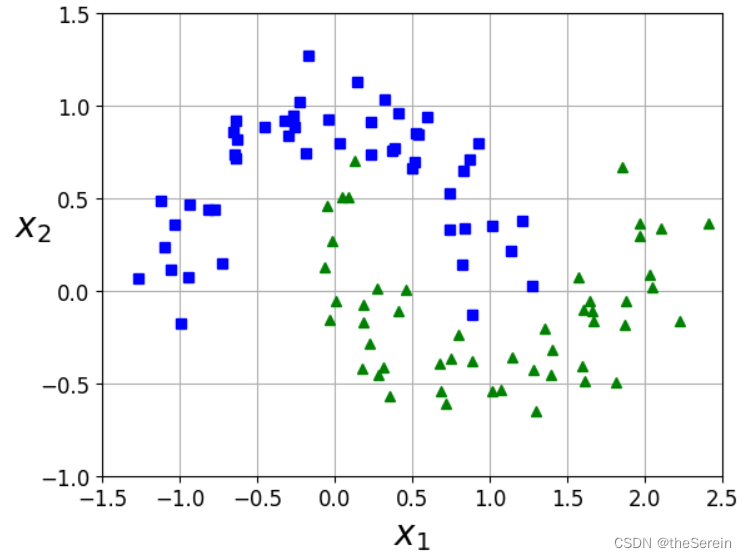
# 接下来创建一份稍有难度的数据(两个相交的月牙)
from sklearn.datasets import make_moons
X, y = make_moons(n_samples=100, noise=0.15, random_state=42)
# 定义绘图函数
def plot_dataset(X, y, axes):
plt.plot(X[:,0][y==0], X[:,1][y==0],"bs")
plt.plot(X[:,0][y==1], X[:,1][y==1],"g^")
plt.axis(axes)
plt.grid(True, which='both')
plt.xlabel(r"$x_1$", fontsize=20)
plt.ylabel(r"$x_2$", fontsize=20,rotation=0)
# 对构建的数据进行可视化展示
plot_dataset(X, y,[-1.5,2.5,-1,1.5])
plt.show()
方法1:利用 PolynomialFeatures 来对原始数据进行升维,然后再利用线性 SVM 对数据进行训练
# 导入相关库
from sklearn.datasets import make_moons
from sklearn. pipeline import Pipeline
from sklearn. preprocessing import PolynomialFeatures
# 进行流水线封装
polynomial_svm_clf = Pipeline((("poly_features", PolynomialFeatures(degree=3)),
("scaler" , StandardScaler()),
("svm_clf", LinearSVC(C=10,loss="hinge"))
))
# 训练模型
polynomial_svm_clf.fit(X, y)
# 定义用于绘制决策边界的函数
def plot_predictions(clf,axes):
# 构建坐标棋盘
x0s = np.linspace(axes[0], axes[1], 100)
x1s = np.linspace(axes[2], axes[3], 100)
x0,x1 = np.meshgrid(x0s, x1s)
# 组合得到一份数据集
X = np.c_[x0.ravel(),x1.ravel()]
# 利用训练好的模型进行预测
y_pred = clf.predict(X).reshape (x0.shape)
# 绘制决策边界
plt.contourf(x0,x1,y_pred, cmap = plt.cm.brg, alpha=0.2)
# 将数据传入并绘制决策边界
plot_predictions(polynomial_svm_clf,[-1.5,2.5,-1,1.5])
# 同时绘制出训练数据集
plot_dataset(X,y,[-1.5,2.5,-1,1.5])

方法 2:利用核技巧(选用 poly 核)
实际上,poly 核所做的工作就是方法 1 中所执行的。不同的是,当你选用了 poly 核后,就不需要再手动对数据集进行升维处理(此时,对原数据集的升维处理被整合到了整个模型的构建中)。
from sklearn.svm import SVC
# 定义一个 SVM 模型(并进行流水线封装)
# 注:coef0 参数表示偏置项
poly_kernel_svm_clf = Pipeline([
("scaler",StandardScaler()),
("svm_clf",SVC(kernel="poly", degree=3,coef0 = 10 ,C=5))
])
# 训练模型
poly_kernel_svm_clf.fit(X, y)
# 定义一个对比模型(将 degree 值设置更大一些)
poly100_kernel_svm_clf = Pipeline([
("scaler",StandardScaler()),
("svm_clf",SVC(kernel="poly", degree=10,coef0 = 10 ,C = 5))
])
# 训练模型
poly100_kernel_svm_clf.fit(X, y)
# 画图对比两个模型的效果
plt.figure(figsize=(11,4))
# 模型 1
plt.subplot(121)
plot_predictions(poly_kernel_svm_clf,[-1.5,2.5,-1,1.5])
plot_dataset(X, y,[-1.5,2.5,-1,1.5])
plt.title(r"$d=3, r=10,C=5$",fontsize=18)
# 模型 2
plt.subplot(122)
plot_predictions(poly100_kernel_svm_clf,[-1.5,2.5,-1,1.5])
plot_dataset(X, y,[-1.5,2.5,-1,1.5])
plt.title(r"$d=10,r=10,C=5$", fontsize=18)
# 展示
plt.show()
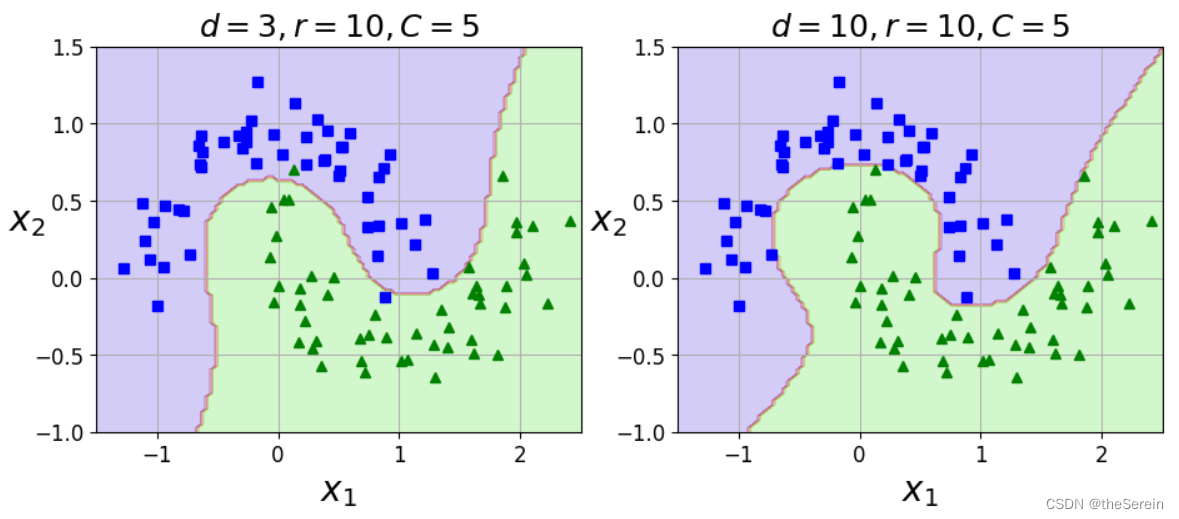
方法 3:利用核技巧(选用高斯核)
高斯核的本质是(假设原数据集的样本点个数为 n ):
计算样本点 i i i 与其他所有样本点的相似度,从而得到一个长度为 n − 1 n-1 n−1 的向量,并以该向量作为该样本点的新特征(此时的特征向量即为 [ x 1 , … , x i − 1 , x i + 1 … , x n ] [x_1,…,x_{i-1},x_{i+1}…,x_n] [x1,…,xi−1,xi+1…,xn])。
因此,在用高斯核函数对数据进行处理后,数据的特征将发生本质改变。而新特征的长度则仅与数据集中的样本点个数相关。
总结:高斯核是通过计算相似度来进行特征替换的。
下面介绍样本点的相似度计算规则(为便于理解和演示,采用一维数据作为原数据集。同时,下面的例子仅将数据的特征长度提升至二维,因此这里随机选择了两个点: x 1 ( j ) = − 2 x_1^{(j)}=-2 x1(j)=−2 和 x 1 ( k ) = 1 x_1^{(k)}=1 x1(k)=1 作为目标点):
- 为目标点其添加高斯函数;
- 取 γ = 0.3 \gamma=0.3 γ=0.3 的径向基函数(RBF)作为相似度函数,即:
φ ( x , l ) = e − γ ( x − l ) 2 \varphi(\text{x},l)=e^{-\gamma(\text{x}-l)^2} φ(x,l)=e−γ(x−l)2
这里演示下对于样本点 x 1 ( i ) x_1^{(i)} x1(i),其相似度是如何得到的:
取样本点 x 1 ( i ) = − 1 x_1^{(i)}=-1 x1(i)=−1时,它距离 x 1 ( j ) = − 2 x_1^{(j)}=-2 x1(j)=−2的长度为 1 ,距离 x 1 ( k ) = 1 x_1^{(k)}=1 x1(k)=1的长度为2。则得到其新特征 x 2 ( i ) = e x p − 0.3 × 1 2 ≈ 0.74 x_2^{(i)}=exp^{-0.3×1^2}≈0.74 x2(i)=exp−0.3×12≈0.74、 x 3 ( i ) = e x p − 0.3 × 2 2 ≈ 0.30 x_3^{(i)}=exp^{-0.3×2^2}≈0.30 x3(i)=exp−0.3×22≈0.30。此时,样本点由原来的特征 [ − 1 ] [-1] [−1] 变为 [ 0.74 , 0.30 ] [0.74,0.30] [0.74,0.30]。
下面画图展示这个升维过程:
# 绘图代码
# 定义径向基函数
def gaussian_rbf(x,landmark,gamma):
return np.exp(-gamma * np.linalg.norm(x - landmark,axis=1)**2)
# 定义 gamma 值
gamma = 0.3
# 设置原始数据集的坐标棋盘
x1s = np.linspace(-4.5,4.5,200).reshape(-1,1)
# 利用径向基函数对原始坐标棋盘进行相似度计算(这里构成的数据主要是用来绘制两个高斯分布的图像)
# 新的维度 1
x2s =gaussian_rbf(x1s,-2,gamma)
# 新的维度 2
x3s =gaussian_rbf(x1s,1,gamma)
# 利用径向基函数对原始数据进行相似度计算
XK = np.c_[gaussian_rbf(X1D,-2,gamma), gaussian_rbf(X1D, 1, gamma)]
# 设置指定原始数据的标签值
yk = np.array([0,0,1,1,1, 1, 1,0,0])
# 绘图展示
plt.figure(figsize=(11,4))
# 绘制经计算后得到的原始数据的相似度分布图像
plt.subplot(121)
plt.grid(True, which='both')
plt.axhline(y=0,color='k')
plt.scatter(x=[-2,1], y=[0,0],s=150,alpha=0.5, c="red")
plt.plot(X1D[:,0][yk==0], np.zeros(4),"bs")
plt.plot(X1D[:,0][yk==1], np.zeros(5),"g^")
plt.plot(x1s, x2s,"g--")
plt.plot(x1s, x3s,"b:")
plt.gca().get_yaxis().set_ticks([0,0.25,0.5,0.75,1])
plt.xlabel(r"$x_1$", fontsize=20)
plt.ylabel(r"Similarity",fontsize=14)
plt.annotate(r'$\mathbf{x}$',
xy=(X1D[3,0],0),
xytext=(-0.5,0.20),
ha="center",
arrowprops=dict(facecolor='black', shrink=0.1),
fontsize=18,
)
plt.text(-2,0.9,"$x_2$", ha="center", fontsize=20)
plt.text(1,0.9,"$x_3$", ha="center", fontsize=20)
plt.axis([-4.5,4.5,-0.1,1.1])
# 绘制将原始数据进行升维操作后的新数据分布情况
plt.subplot(122)
plt.grid(True,which='both')
plt.axhline(y=0,color='k')
plt.axvline(x=0,color='k')
plt.plot(XK[:,0][yk==0],XK[:,1][yk==0],"bs")
plt.plot(XK[:,0][yk==1],XK[:,1][yk==1],"g^")
plt.xlabel(r"$x_2$", fontsize=20)
plt.ylabel(r"$x_3$", fontsize=20,rotation=0)
plt.annotate(r'$\phi\left(\mathbf {x} \right)$',xy=(XK[3,0],XK[3,1]),
xytext=(0.65,0.50),
ha="center",
arrowprops=dict(facecolor='black', shrink=0.1),
fontsize=18,
)
# 根据图像可大致绘制一条分界线
plt.plot([-0.1,1.1],[0.57,-0.1],"r--",linewidth=3)
plt.axis([-0.1,1.1,-0.1,1.1])
plt.subplots_adjust(right=1)
plt.show()
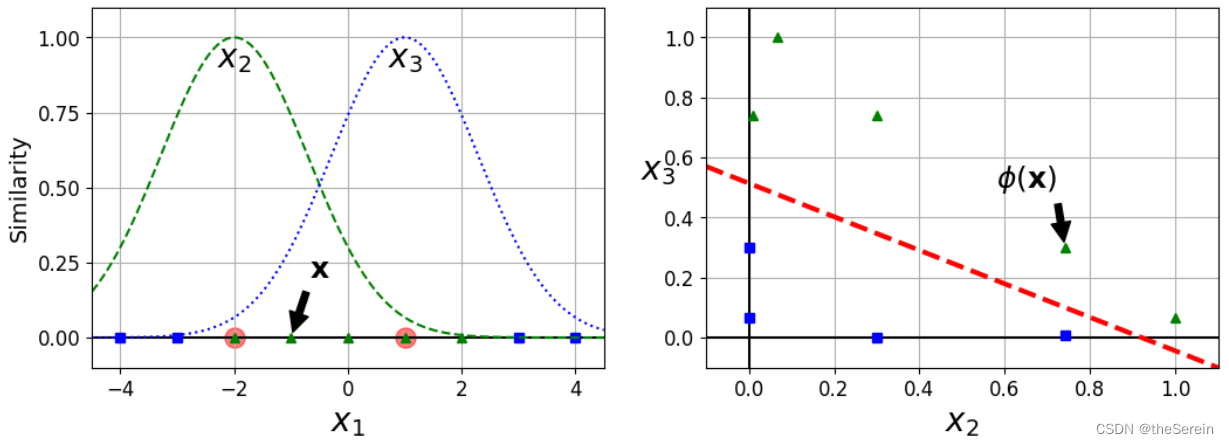
上示左图,展示了以数据点 x = − 2 x=-2 x=−2 和 x = 1 x=1 x=1 为高斯函数(中心点)的概率密度函数。根据选定的这两个样本点,可以计算其余所有数据点在这两个高斯函数上的取值。因此,对于原始数据集中的每个数据点,都会得到两个新的值。我们视这两个值分别为该数据点到指定数据点 x = − 2 x=-2 x=−2 和 x = 1 x=1 x=1 的相似度,并以这两个值构成原数据点的新特征绘至上示右图中。
此时,我们再来看原始分类任务:
- 左图是在一维数据中区分出“蓝色”与“绿色”,显然不太容易;
- 右图是在二维数据中区分出“蓝色”与“绿色”,可以很轻松地找出若干条简单而有效的分界线。
# 现在我们尝试将 gamma 的值减小,看会发生怎样的改变
gamma = 0.1
# 设置原始数据集的坐标棋盘
x1s = np.linspace(-4.5,4.5,200).reshape(-1,1)
# 利用径向基函数对原始坐标棋盘进行相似度计算(这里构成的数据主要是用来绘制两个高斯分布的图像)
# 新的维度 1
x2s =gaussian_rbf(x1s,-2,gamma)
# 新的维度 2
x3s =gaussian_rbf(x1s,1,gamma)
# 利用径向基函数对原始数据进行相似度计算
XK = np.c_[gaussian_rbf(X1D,-2,gamma), gaussian_rbf(X1D, 1, gamma)]
# 设置指定原始数据的标签值
yk = np.array([0,0,1,1,1, 1, 1,0,0])
# 绘图展示
plt.figure(figsize=(11,4))
# 绘制经计算后得到的原始数据的相似度分布图像
plt.subplot(121)
plt.grid(True, which='both')
plt.axhline(y=0,color='k')
plt.scatter(x=[-2,1], y=[0,0],s=150,alpha=0.5, c="red")
plt.plot(X1D[:,0][yk==0], np.zeros(4),"bs")
plt.plot(X1D[:,0][yk==1], np.zeros(5),"g^")
plt.plot(x1s, x2s,"g--")
plt.plot(x1s, x3s,"b:")
plt.gca().get_yaxis().set_ticks([0,0.25,0.5,0.75,1])
plt.xlabel(r"$x_1$", fontsize=20)
plt.ylabel(r"Similarity",fontsize=14)
plt.annotate(r'$\mathbf{x}$',
xy=(X1D[3,0],0),
xytext=(-0.5,0.20),
ha="center",
arrowprops=dict(facecolor='black', shrink=0.1),
fontsize=18,
)
plt.text(-2,0.9,"$x_2$", ha="center", fontsize=20)
plt.text(1,0.9,"$x_3$", ha="center", fontsize=20)
plt.axis([-4.5,4.5,-0.1,1.1])
# 绘制将原始数据进行升维操作后的新数据分布情况
plt.subplot(122)
plt.grid(True,which='both')
plt.axhline(y=0,color='k')
plt.axvline(x=0,color='k')
plt.plot(XK[:,0][yk==0],XK[:,1][yk==0],"bs")
plt.plot(XK[:,0][yk==1],XK[:,1][yk==1],"g^")
plt.xlabel(r"$x_2$", fontsize=20)
plt.ylabel(r"$x_3$", fontsize=20,rotation=0)
plt.annotate(r'$\phi\left(\mathbf {x} \right)$',xy=(XK[3,0],XK[3,1]),
xytext=(0.65,0.50),
ha="center",
arrowprops=dict(facecolor='black', shrink=0.1),
fontsize=18,
)
# 根据图像可大致绘制一条分界线
plt.plot([-0.1,1.1],[0.9,0.2],"r--",linewidth=3)
plt.axis([-0.1,1.1,-0.1,1.1])
plt.subplots_adjust(right=1)
plt.show()
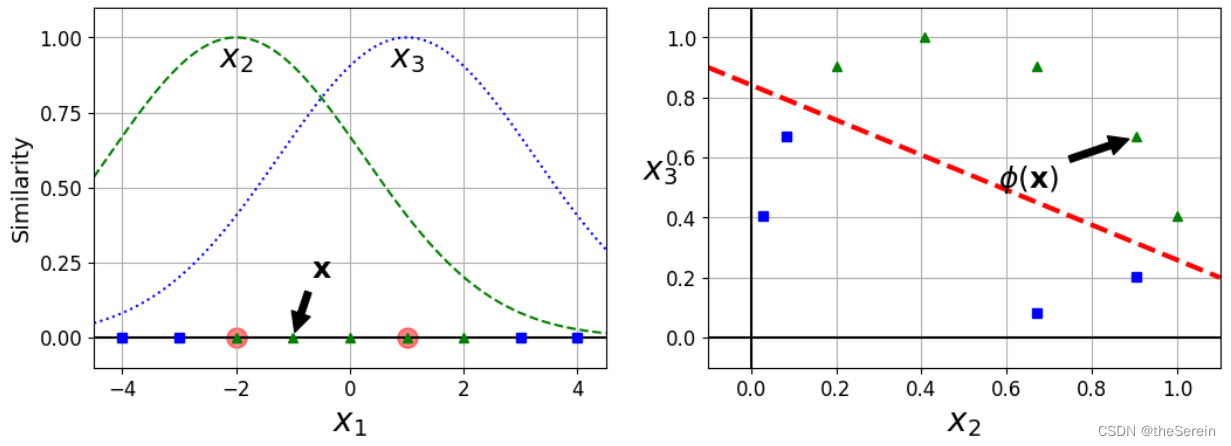
实际上,当 γ \gamma γ 值减小时,高斯分布会变得“矮胖”,其涵盖的数据范围将大大增加(也就是说会更“公平地”对待每一条数据)。此时,过拟合风险会大大降低。换言之,当 γ \gamma γ 值取得较大时,模型的过拟合风险会增加。
六、对比试验: 𝛾 和 C 值对 SVM 的影响。
from sklearn.svm import SVC
gamma1,gamma2 = 0.1,5
C1,C2 = 0.001,1000
hyperparams = (gamma1,C1),(gamma1,C2),(gamma2,C1),(gamma2,C2)
svm_clfs =[]
for gamma,C in hyperparams:
rbf_kernel_svm_clf = Pipeline([
("scaler", StandardScaler()),
("svm_clf", SVC(kernel="rbf", gamma=gamma,C=C))
])
rbf_kernel_svm_clf.fit(X, y)
svm_clfs.append(rbf_kernel_svm_clf)
plt.figure(figsize=(11,7))
for i,svm_clf in enumerate(svm_clfs):
plt.subplot(221 + i)
plot_predictions(svm_clf,[-1.5,2.5,-1,1.5])
plot_dataset(X, y,[-1.5,2.5,-1,1.5])
gamma,C = hyperparams[i]
plt.title(r"$\gamma = {}, C = {}$".format(gamma,C),fontsize=16)
plt.show()
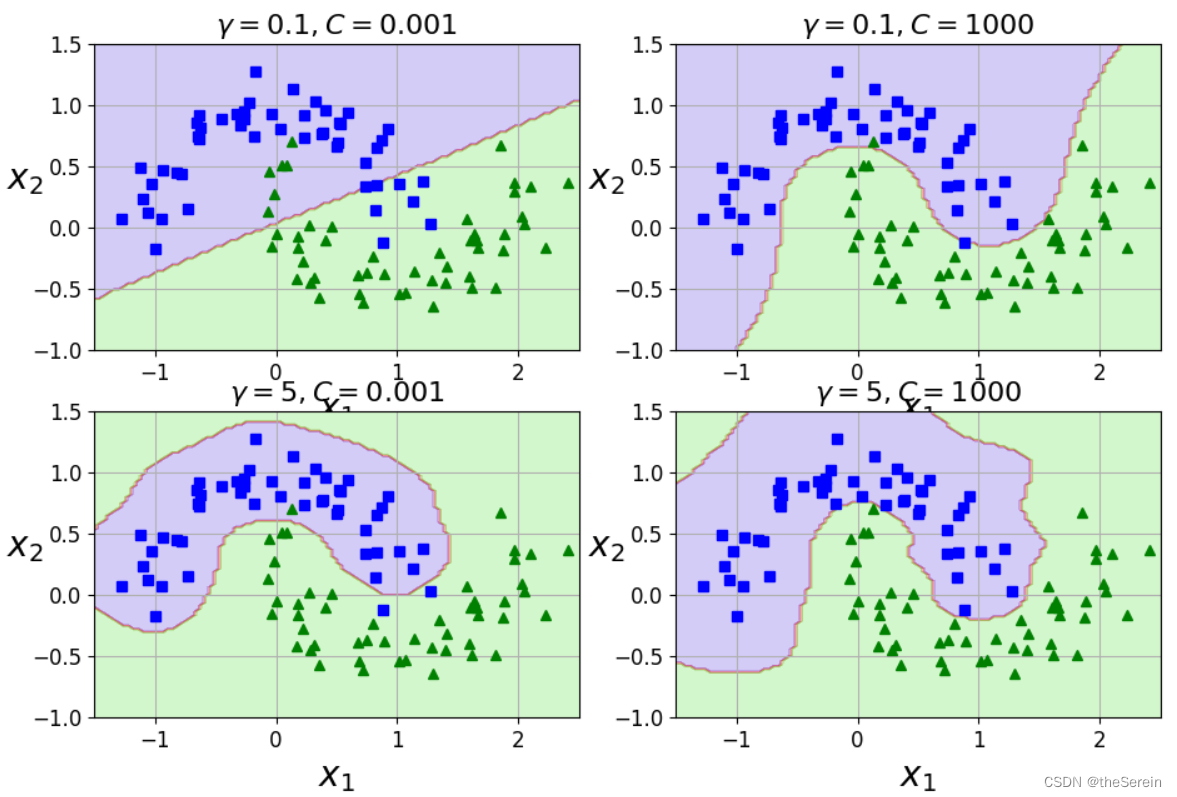
从上图可以做出如下总结:
- 由于增加 γ \gamma γ 会使地高斯函数变窄,因此这将降低其对数据集中各实例的影响范围,从而使得最终的决策边界变得更不规则(仅在个别实例周围摆动)。
- 增大 C 值,模型的拟合效果会提升,但过拟合风险也会增高。