-
发表单位:中国科学院江西省庐山植物园
-
发表日期:2023年9月13日
-
研究期刊:International Journal of Molecular Sciences(IF: 5.6)
-
研究材料:拟南芥
2023年9月13日,中国科学院江西省庐山植物园胡伟明课题组在International Journal of Molecular Sciences杂志在线发表了题为“The Change in Whole-Genome Methylation and Transcriptome Profile under Autophagy Defect and Nitrogen Starvation”的研究论文。本研究揭示了细胞自噬在基因组甲基化和转录组谱调控中的关键作用,为作者深入理解自噬与细胞生理过程之间的联系提供了重要的科学依据。同时,这也为未来深入研究自噬与植物生长发育之间的关系提供了坚实的基础。这一研究在细胞生物学领域具有重要的意义,也为相关领域的进一步研究提供了有力的支持。爱基百客为该研究提供WGBS和RNA-seq的技术支持。


1 研究背景
随着科技的不断进步,研究者对于细胞内各种生物学过程的认识也在不断深化。自噬作为一种细胞内的关键生物学过程,一直备受科学家们的关注。自噬是真核生物中一种促进细胞长寿和营养循环的普遍机制,有助于植物应对各种生物和非生物胁迫。
近年来,各种研究表明自噬在植物的多种生物过程中起着关键作用,包括生长、发育、淀粉降解、衰老和脂质代谢的控制[1]。在拟南芥中,自噬还影响了花粉管的生长[2]。此外,在动物中也验证了自噬与表观遗传学存在着密切的关系。在营养缺乏的条件下,成纤维细胞生长因子(FGF21)通过组蛋白(H3K27-ME3)的去甲基化酶激活自噬相关基因的表达,从而促进了脂质降解[3]。
自噬调控网络也会受到DNA甲基化的调控。有研究表明MAP1LC3是自噬核心机制的组成部分。由DNA甲基转移酶DNMT3A介导的该位点的DNA甲基化会导致MAP1LC的转录水平持续下调[4]。在动物中已经有大量的研究表明自噬与表观遗传学调控存在密切的关系。然而,目前尚不清楚植物自噬是否会影响全基因组DNA甲基化。因次,本研究旨在探讨自噬缺陷和氮饥饿条件下整个基因组的甲基化水平以及转录组谱的变化,从而深入揭示了这两种条件对于细胞生物学特性的影响。
2 研究思路
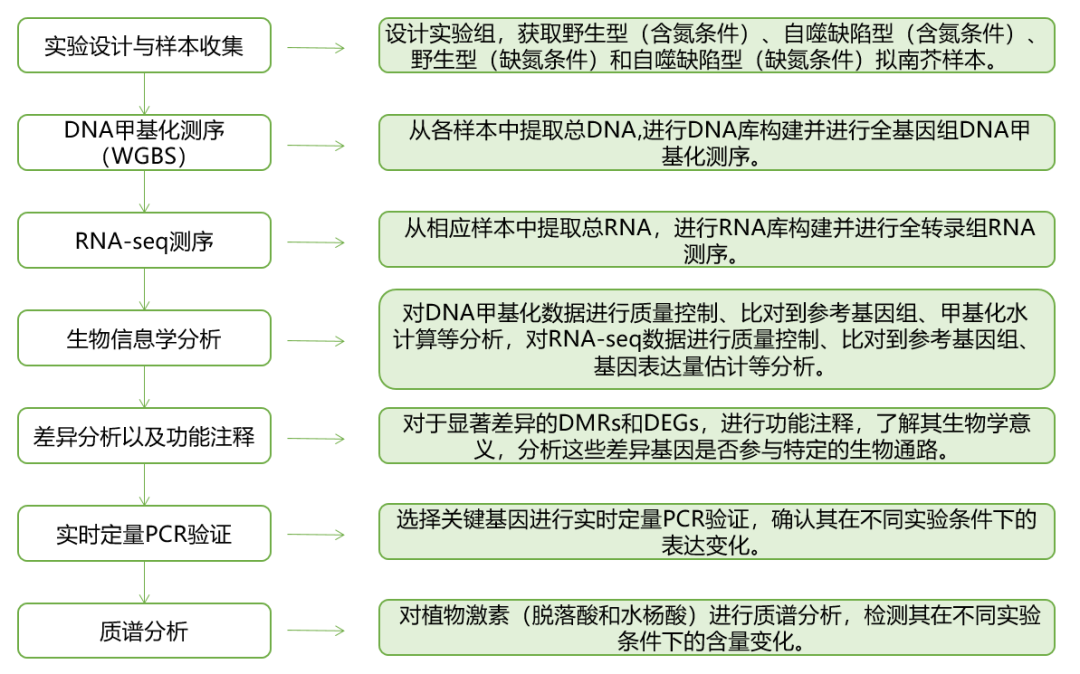
3 研究结果
01 氮饥饿条件下自噬突变体的全基因组甲基化水平受到显著影响
该研究探讨了自噬是否通过DNA甲基化影响植物在氮饥饿胁迫下的生长。研究使用拟南芥野生型(Col-0)和自噬突变体(atg5-1)在不同培养条件下进行了全基因组亚硫酸盐测序(WGBS)和转录组测序(RNA-seq)(图1A),并分析了DNA甲基化类型(CG、CHG和CHH)的变化。结果表明,在野生型拟南芥中,氮饥饿对甲基化水平的影响不显著,但在自噬缺陷的情况下,氮饥饿对甲基化水平的影响显著增加(图1B)。在atg5-1基因型样本和野生型样本中,生长在MS和氮饥饿条件下的DMR数量存在显著差异,这似乎表明在野生型样本中,一些补偿效应可能抵制了氮饥饿对甲基化水平的影响;然而,这种补偿效应可能会因为自噬缺陷而受到抑制(图1C)。作者检查了每个处理组的共同甲基化位点,结果显示,atg5-1样本的总体甲基化水平明显低于野生型样本,这进一步表明了DNA甲基化水平的下降很大程度上可以归因于自噬缺陷(图1D)。
综合来看,作者发现氮饥饿对拟南芥的甲基化水平影响不显著,但当自噬缺陷存在时,氮饥饿的影响会显著增强。这表明自噬在植物中通过影响DNA甲基化,可能在应对氮饥饿等胁迫条件下扮演着重要的调节角色。
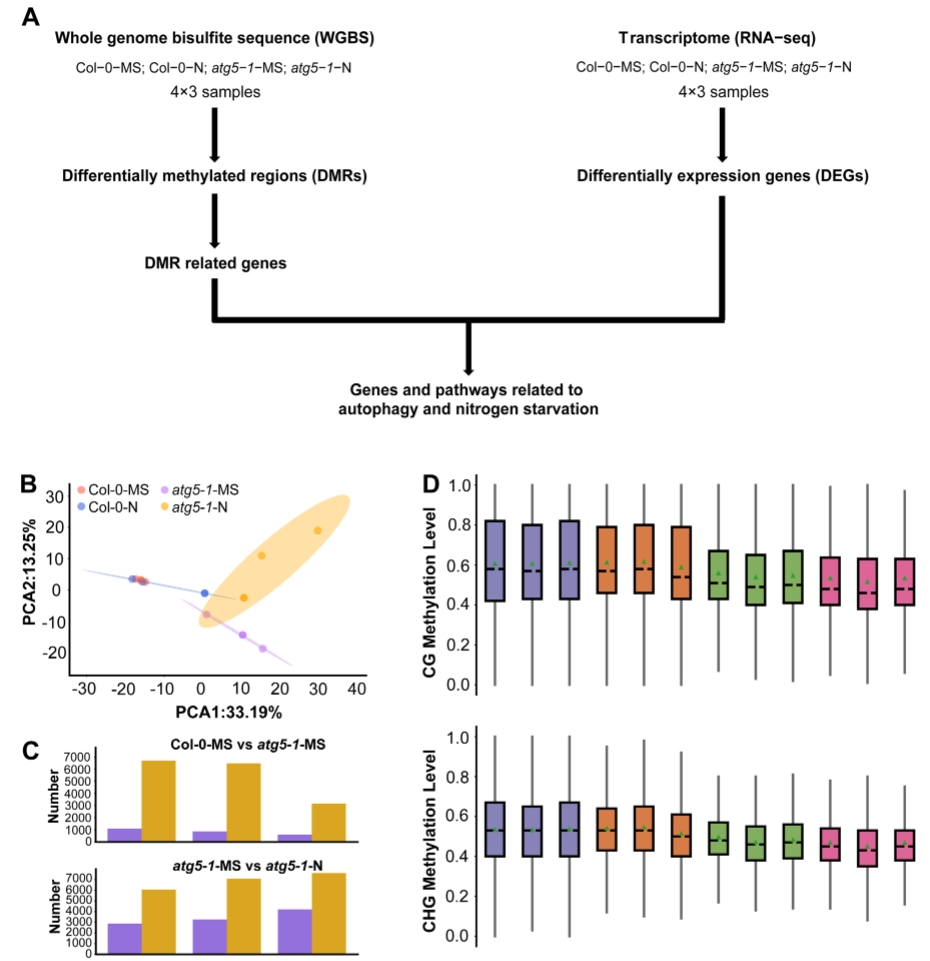
图1 自噬功能缺陷导致全基因组甲基化水平的变化
02 拟南芥基因组在氮饥饿条件下的全局表达
为了研究自噬对拟南芥在氮饥饿和正常条件下的基因表达变化。作者通过RNA-seq生成了不同条件下的转录组谱系。结果显示,在氮饥饿条件下,拟南芥野生型(Col-0)和自噬缺陷型(atg5-1)中上调基因数量明显多于下调基因,这表明氮饥饿激活了全局基因表达以维持拟南芥的生长。在自噬缺陷的情况下,氮饥饿仍然可能激活许多基因的表达作出响应。然而,自噬缺陷对正常条件下的植物生长影响似乎不大(图2A,B)。特别是在与细胞膜、防御反应相关的基因上调中,自噬缺陷和正常情况下的基因表达变化相对较小。进一步的功能富集分析显示,在氮饥饿条件下,与ABC转运蛋白、叶绿体类囊体膜等相关的基因上调明显富集,暗示了这些基因在氮饥饿应答中的重要性。与类囊体相关的基因在下调中富集,暗示了自噬在类囊体调控方面的作用(图2C)。在自噬过程中,蛋白质自我结合和蛋白质降解起着主要作用,反映在差异表达基因中与蛋白质结构、未折叠蛋白的处理等功能富集。此外,内质网中的蛋白质处理和色氨酸代谢通路也受到影响,与热休克蛋白等相关(图2D、E、F)。
综合来看,自噬缺陷会放大氮饥饿对基因表达的影响。自噬在植物应对氮饥饿等胁迫条件下扮演着重要的调节角色,特别是在蛋白质降解和细胞膜调控等方面发挥着关键作用。
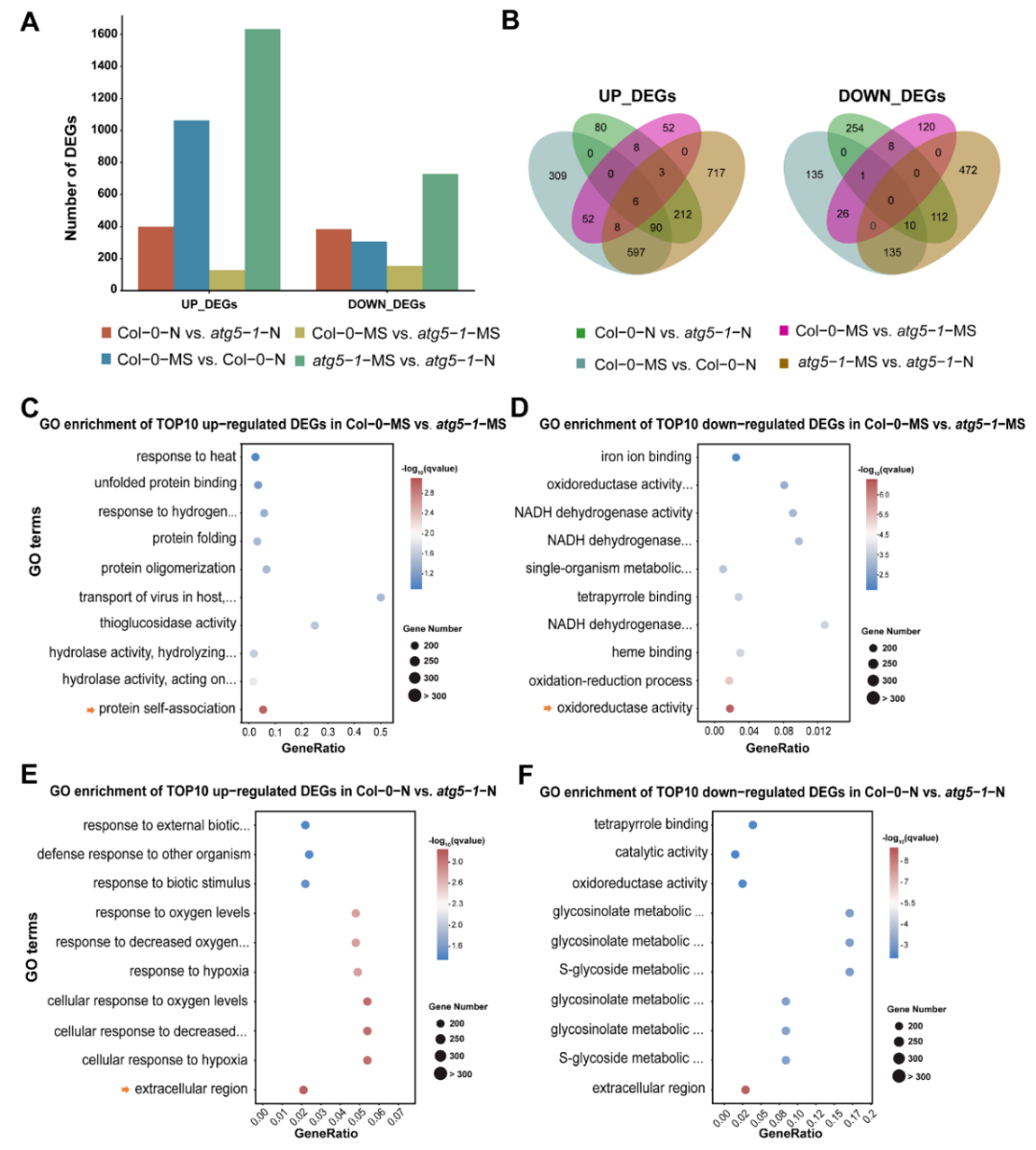
图2 氮饥饿激活了Col-0和自噬突变体整个基因组的差异表达
03 自噬缺陷可能导致DNA甲基化水平下调,从而激活与细胞死亡和分解代谢途径相关的基因表达
为了探究自噬缺陷引发的DNA低甲基化可能对基因表达产生的潜在影响,作者首先统计了与上调基因表达相关的低甲基化区域(hypo-DMRs)的数量,发现在各比较组中,大多数差异表达基因(DEGs)位于这些区域,但只有少数受到DNA甲基化调控(图3A)。进一步的分析显示,在自噬缺陷的情况下,与低甲基化区域相关的上调DEGs主要富集在与程序性细胞死亡、分解代谢过程和细胞对刺激的反应相关的功能中。同时,与细胞膜、防御反应相关的基因也显著富集,暗示了自噬缺陷可能通过DNA低甲基化影响拟南芥对氮缺乏的抵抗机制(图3B)。此外,通过GO富集和KEGG分析发现,ABC转运蛋白可能在自噬缺陷型植物对氮缺乏的应对中起到重要作用。这进一步证明了DNA低甲基化可能通过激活与细胞死亡和膜转运相关的基因表达来影响自噬缺陷型植物应对氮缺乏的机制(图3C)。作者还研究了DNA低甲基化对水解酶活性和代谢酶活性的调控作用。发现在自噬缺陷条件下,DNA低甲基化显著调节了水解酶活性。一些与hypo-DMR相关的DEGs富集在多种物质代谢和生物合成过程中,暗示了自噬缺陷可能会诱导代谢和生物合成活性(图3B)。
最后,研究表明DNA低甲基化可能通过调控与细胞死亡和膜转运相关的基因表达来影响自噬缺陷型植物应对氮缺乏的机制。此外,DNA甲基化水平的变化也可能参与了自噬缺陷型拟南芥对氮的吸收。整体而言,自噬缺陷可能会影响DNA甲基化水平,从而降低拟南芥对氮饥饿的耐受性(图3C)。此外,在氮饥饿条件下,DNA甲基化可能受到自噬的调控,这可能进一步影响了植物的应对机制。
图3 DNA甲基化影响了自噬突变体对氮缺乏的响应
04 氮缺乏可能诱导ERF和C2H2型锌指蛋白家族的转录因子在atg5-1自噬缺陷体中调节ABA诱导的生长抑制和SA生物合成
作者绘制了不同比较组的韦恩图,发现大多数上调的差异表达基因(DEGs)仅在Col-0-N 和 atg5-1-N 中出现了甲基化(图4A)。研究指出低甲基化区域(LMRs)通常涉及招募转录因子(TFs)与基因的上调表达相关联。通过对TFs的进一步分析发现,其中大部分属于NAC和ERF家族,如ANAC004、ATERF48、ANAC046等的表达显著差异,可能在自噬缺陷条件下扮演关键角色(图4B)。此外,研究者还观察到一些TFs可能与特定基因的DMRs结合,例如MYB101、PBS3等。这些基因参与了生物刺激、细胞死亡和有机氮化合物的应答,表明这些功能可能受到低甲基化区域招募的TFs调控。
此外,研究结果还揭示了MYB101与ERF家族的转录因子相互作用,这可能在氮缺乏条件下引起植物生长抑制。另一方面,C2H2型锌指蛋白家族蛋白(AT3G46070)与PBS3的结合也被预测,这可能导致了拟南芥的过早衰老。总的来说,自噬缺陷可能通过调控DNA甲基化水平和TFs活性,影响拟南芥对氮缺乏的应对机制。
图4 氮缺乏条件下,与衰老相关的基因受到DNA甲基化的强烈影响
05 SA和ABA合成途径中关键基因的qRT–PCR实验证实了WGBS和RNA-seq分析结果
根据作者的假设,在Col-0-N 和 atg5-1-N 中,DNA甲基化参与了拟南芥ABA诱导的生长抑制过程。因此,作者对上述提及的DEGs进行了qRT–PCR实验证明了作者的假设(图5A)。作者推测,如果ABA参与了自噬缺陷体在氮缺乏条件下的过早衰老,那么ABA的水平将会上升,这也是ABA诱导的衰老受到DNA甲基化影响的证据。因此,作者还验证了与ABA生物合成相关的关键基因的表达水平,结果也与RNA-seq的结果相一致(图5B)。根据WGBS和RNA-seq分析结果,SA的生物合成可能也与DNA甲基化相关,DNA甲基化可能会影响参与SA生物合成的PBS3的表达。作者还对参与SA生物合成途径的基因进行了qRT–PCR实验,结果显示PBS3的表达显著增加,而与ABA生物合成途径的关键基因,如ICS1,也发生了显著变化(图5C)。
qPCR结果表明,植物激素SA和ABA的水平显著上调,与作者的假设一致。这也证实了RNA-seq结果的准确性,并表明差异性DNA甲基化很可能与ABA和SA调控植物生长抑制或过早衰老的机制密切相关。
图5 负责调控水杨酸(SA)和脱落酸(ABA)激素途径的关键基因及预测转录因子的实时定量PCR(qRT-PCR)结果
06 在氮缺乏条件下,atg5-1突变体中SA和ABA激素水平显著升高
作者假设ABA和SA途径可能受到DNA甲基化的调控,从而影响植物的过早衰老。因此,作者检验了SA和ABA的水平。ABA测定的结果显示,在atg5-1-N组中,激素水平明显高于Col-0-N组(图6A)。相对于Col-0-N ,SA的水平在atg5-1-N中显著增加(图6B)。激素测量结果表明,自噬功能的缺失可能降低了拟南芥对氮饥饿的耐受性,从而直接或间接改变了DNA甲基化水平,引起SA和ABA水平的增加,从而导致植株过早衰老。
图6 脱落酸(ABA)和水杨酸(SA)植物激素的定量分析结果
4 研究结论
细胞自噬是真核生物维持细胞稳态的关键生物学过程,广泛参与植物生长发育和生物非生物逆境响应等多种生理过程。作者通过WGBS和RNA-seq技术,发现了许多基因在缺氮条件下的表达会处于激活,特别是当自噬功能缺陷时,DNA甲基化水平发生了显著变化,这揭示了自噬在调控拟南芥DNA甲基化中的潜在影响。这项研究为进一步理解自噬的分子机制以及植物如何应对氮缺乏提供了坚实的基础,也为未来研究提供了更多关于细胞如何应对各种环境压力的机制的新见解。
· 爱基百客WGBS相关介绍 ·
WGBS被认为是DNA甲基化研究的“金标准”,亚硫酸氢盐处理将基因组中未发生甲基化的C碱基转换成U,进行PCR扩增后变成T,与原本具有甲基化修饰的C碱基区分开来,再结合高通量测序技术,可精确绘制全基因组甲基化图谱。爱基百客WGBS产品可提供:
一、WGBS测序分析
01. 全基因组甲基化分析
全基因组DNA甲基化测序(WGBS)可获得全基因组范围内单碱基分辨率的所有DNA甲基化情况,信息最为丰富和全面,是绘制单碱基DNA甲基化图谱的首选方法。
02. 甲基化差异分析
可以进行甲基化差异区域(DMR)的检测、分布,DMR关联基因的分析、DMR关联基因的功能富集分析(GO、KEGG),转录因子预测及motif分析。
二、后续验证
01. 甲基化位点验证——TBS
对后续目标基因进行甲基化验证,其具有高准确性、通量高、周期短、低成本的优势,广泛用于临床大样本多位点的甲基化位点筛选与验证。
02. CpG位点定量分析——MassARRAY
MassARRAY技术可以对多个CpG位点进行定量分析,从而揭示DNA甲基化在基因组范围内的分布和变化情况。
三、甲基化+转录组学关联分析
通过WGBS获得基因甲基化相关信息,与转录组测序(RNA-seq)获得基因的表达量信息相关联,获得甲基化修饰水平与基因表达共同上/下调的正向调控基因(或一方上调另一方下调的基因,即负向调控关系),进行对应基因的富集分析,挖掘差异甲基化修饰潜在的功能调控机制。
· 爱基百客技术优势 ·
①. 协助客户发表WGBS相关文章于New Phytologist、Cell Discovery和Postharvest Biology and Technology等高水平期刊。
②. 拥有人、家蚕、罗非鱼、黄鳝、拟南芥、水稻、大豆、油菜、番茄、葡萄、苹果、碎米荠、青稞、桂花、杨树和樟树等50+物种经验,全面覆盖医口、农口等各类样本。
③. 提供甲基化一站式服务:方案设计、DNA修饰整体水平检测(LC-MS和dot blot)、DNA甲基化测序和验证等服务。
参考文献
[1]. Cheng, S.; Wang, Q.; Manghwar, H.; Liu, F. Autophagy-Mediated Regulation of Different Meristems in Plants. Int. J. Mol. Sci. 2022, 23, 6236.
[2]. Yan, H.; Zhuang, M.; Xu, X.; Li, S.; Yang, M.; Li, N.; Du, X.; Hu, K.; Peng, X.; Huang, W.; et al. Autophagy and its mediated mitochondrial quality control maintain pollen tube growth and male fertility in Arabidopsis. Autophagy 2023, 19, 768–783.
[3]. Byun, S.; Seok, S.; Kim, Y.C.; Zhang, Y.; Yau, P.; Iwamori, N.; Xu, H.E.; Ma, J.; Kemper, B.; Kemper, J.K. Fasting-induced FGF21 signaling activates hepatic autophagy and lipid degradation via JMJD3 histone demethylase. Nat. Commun. 2020, 11, 807.
[4]. Gonzalez-Rodriguez, P.; Cheray, M.; Fullgrabe, J.; Salli, M.; Engskog-Vlachos, P.; Keane, L.; Cunha, V.; Lupa, A.; Li, W.; Ma, Q.; et al. The DNA methyltransferase DNMT3A contributes to autophagy long-term memory. Autophagy 2021,17, 1259–1277.
[5]. Henderson, I.R.; Jacobsen, S.E. Epigenetic inheritance in plants. Nature 2007, 447, 418–424.
[6]. Li, S.; Tollefsbol, T.O. DNA methylation methods: Global DNA methylation and methylomic analyses. Methods 2021,187, 28–43.
[7]. Wang, P.; Nolan, T.M.; Yin, Y.; Bassham, D.C. Identification of transcription factors that regulate ATG8 expression and autophagy in Arabidopsis. Autophagy 2020, 16, 123–139.
[8]. Yang, X.; Srivastava, R.; Howell, S.H.; Bassham, D.C. Activation of autophagy by unfolded proteins during endoplasmic reticulum stress. Plant J. 2016, 85, 83–95.
[9]. Stadler, M.B.; Murr, R.; Burger, L.; Ivanek, R.; Lienert, F.; Scholer, A.; van Nimwegen, E.; Wirbelauer, C.; Oakeley, E.J.; Gaidatzis, D.; et al. DNA-binding factors shape the mouse methylome at distal regulatory regions. Nature 2011, 480, 490–495.
[10]. Fernandez-Jimenez, N.; Sklias, A.; Ecsedi, S.; Cahais, V.; Degli-Esposti, D.; Jay, A.; Ancey, P.B.; Woo, H.D.; Hernandez-Vargas, H.; Herceg, Z. Lowly methylated region analysis identifies EBF1 as a potential epigenetic modifier in breast cancer. Epigenetics 2017 ,12, 964–972.