介绍dcmtk和dcm2bids
BIDS(Brain Imaging Data Structure)是一种用于规范化神经影像数据的标准,它定义了数据的目录结构、文件命名规则和元数据格式。BIDS使得数据的共享和分析更加容易,同时很多主流的图像处理自动化软件也需要BIDS存储格式来作为输入。
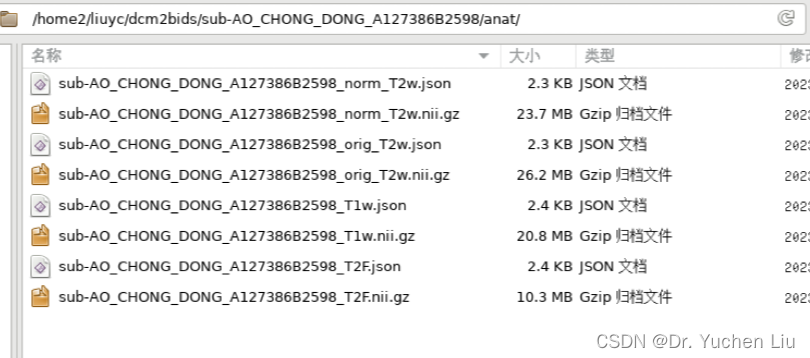
数据存储模式如下:

每个影像模态文件夹内包含了对应文件:
介绍dcm2bids
dcm2bids工具用于将DICOM格式的医学影像数据转换为BIDS格式。
安装Conda
在开始安装dcm2bids之前,您需要安装Conda,这是一个流行的包管理器,用于创建和管理Python环境。以下是在Linux上安装Conda的步骤:
- 下载Miniconda:访问Miniconda官网(https://docs.conda.io/en/latest/miniconda.html),下载适配系统的版本。
- 运行安装脚本:打开终端并导航到包含下载的脚本的目录。运行以下命令,其中Miniconda3-latest-Linux-x86_64.sh是您下载的脚本文件名:
bash Miniconda3-latest-Linux-x86_64.sh - 按照安装程序的提示:按照安装程序的提示进行安装,可以通过按回车键接受默认选项。安装完成后,您需要重新启动终端。
安装dcm2bids
一旦您成功安装了Conda,接下来,您可以使用Conda创建一个新的环境并在其中安装dcm2bids与相关工具包。
- 创建Conda环境:在终端中运行以下命令以创建一个新的Conda环境,命名为"dcm2bids"(您可以根据需要选择不同的环境名):
conda create -n dcm2bids python=3 - 激活环境:激活新创建的环境:
conda activate dicom_env - 安装dcm2bids:使用pip(Python包管理器)安装dcm2bids:
pip install dcm2bids - 安装dcm2niix:
conda install -c conda-forge dcm2niix - 安装pigz:
conda install -c conda-forge pigz
现在,您的环境已经准备好,可以使用dcm2bids工具将DICOM数据转换为BIDS格式。
使用dcm2bids
dcm2bids工具使用命令行完成:
dcm2bids -d DICOM_DIR -p PARTICIPANT_ID <-s SESSION_ID> -c CONFIG_FILE-d 对应原始所有dicom所在路径,-p对应您想给被试的命名,-s指纵向队列中多次采集的次序,CONFIG_FILE是一个说明文件,用来从dicom文件源信息中抓取关键字,将转格式后生成的文件放进BIDS对应的文件夹中,其生成方式参考https://unfmontreal.github.io/Dcm2Bids/3.1.1/how-to/create-config-file/。
*需要注意的是,不同版本的dcm2bids对-p和CONFIG_FILE有不同要求:
2.*.*版本执行时,新生成的被试名可以包含字符和数字之外的符号,所用的config文件是老版本,即*.conf:

较新的3.*.*版本,新被试名只能包含字符和数字,所用的config文件是新版本:

新老版本CONFIG_FILE的差别参考https://unfmontreal.github.io/Dcm2Bids/3.0.1/upgrade/#description-keys