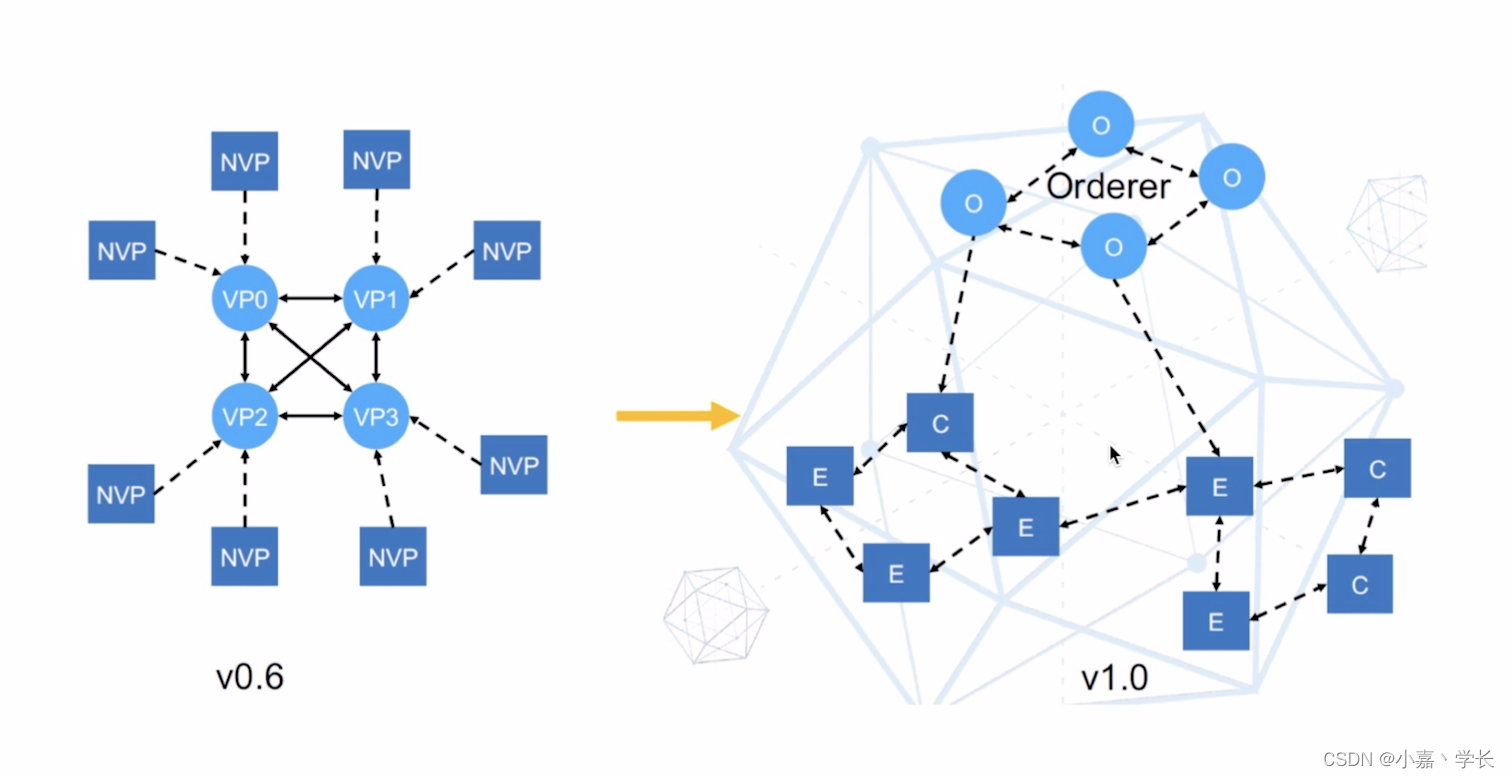
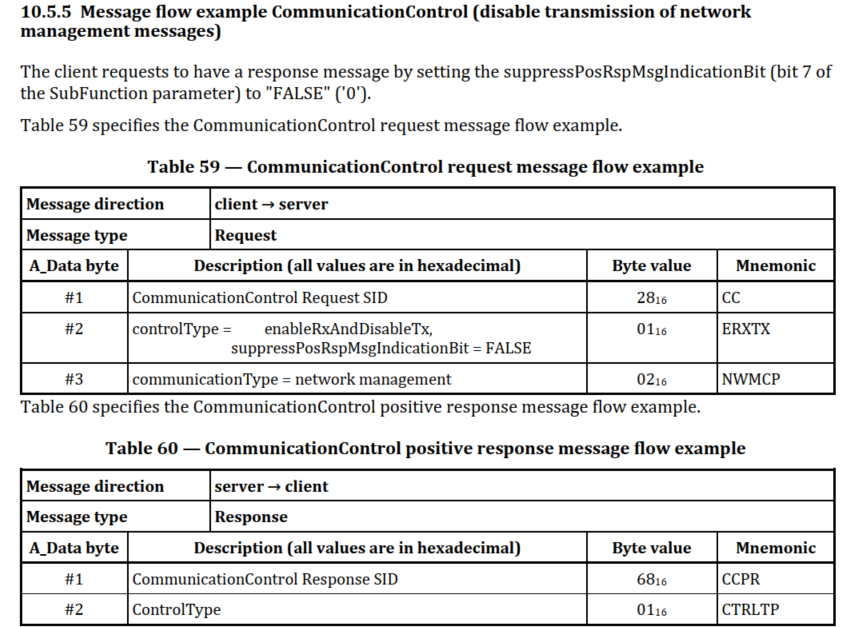
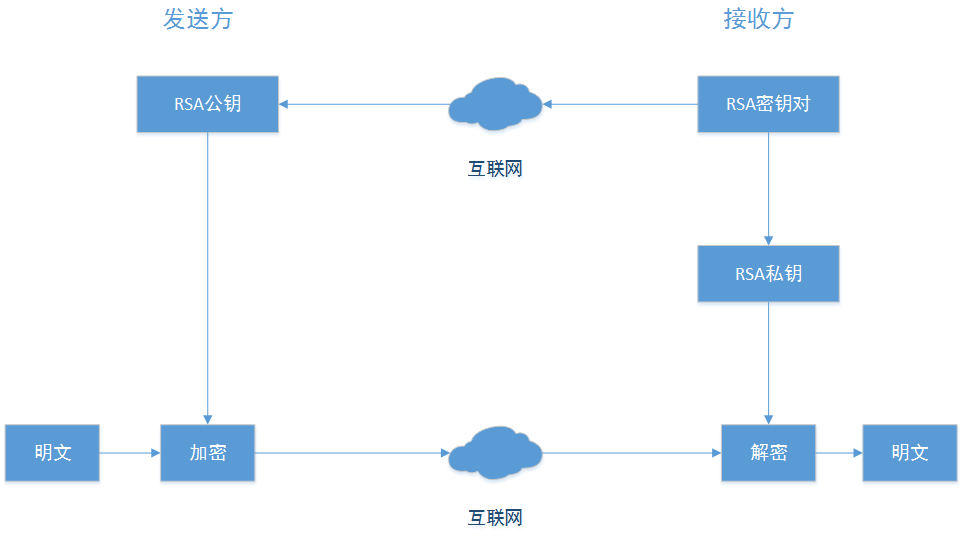
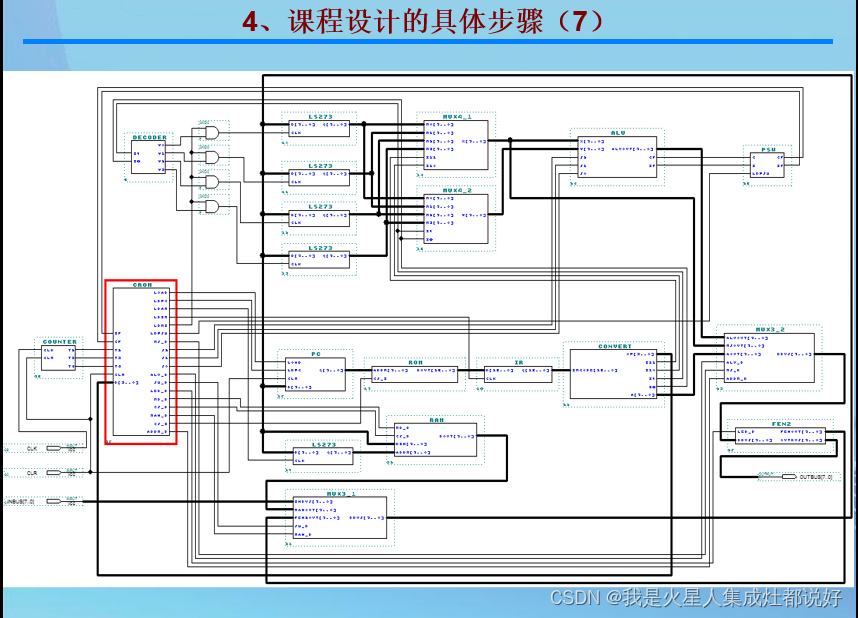
一、选择消除分析
所谓选择性清除:当一个有利突变发生后,这个突变基因的适合度越高,就越容易被选择固定。当这个基因被快速固定之后,与此基因座连锁的染色体区域,由于搭车效应也被固定下来,大片紧密连锁的染色体区域因此失去多态性,这种由于搭车效应引起多态性下降的现象,遗传上称为选择清除。
群体进化选择消除分析:简单的说就是基因组某区域由于受到了选择而消除多态性。选择消除分析是正向选择在物种基因组上留下的印迹。与野生祖先相比,栽培或驯化的物种发生选择消除的区域遗传多样性显著降低,这是驯化区域的典型特征。参考文章
下图可以形象的展示受选择后的结果。

选择消除分析可以做什么?
- 1.挖掘人工驯化过程中受选择的基因,如家猪与肉质相关的基因,大豆中与油含量相关的基因。
- 2.挖掘物种适应性进化中受选择的基因,如藏猪的高原适应性,毛果杨高低纬度气候适应性。
二、常见名词
A、Ka/Ks
Ks = 同义突变SNP数/同义位点数, 即同义突变率
Ka = 非同义突变SNP数/非同义位点数, 即非同义突变率
Ka/Ks >> 1,基因受正选择(positive selection)
Ka/Ks =1,基因中性进化(neutral evolution)
Ka/Ks << 1,基因受纯化选择(purify selection)
用于计算Ka/Ks的软件:KaKs_Calculator(常用)、PAML、Hyphy、MEGA等。
B、FST
FST:遗传分化指数,取值范围是[0,1],最大值为1表明两个群体完全分化,最小值为0表明群体间无分化。
在实际的研究中Fst值为0–0.05时说明群体间遗传分化很小,可以不做考虑;
为0.25以上的时候群体间就存在很大的遗传分化了.
C、π:表示核苷酸多态性,受选择程度越高(换而言之,受选择程度越高表示由于群体的选择清除的作用被固定的区域范围越大),导致群体的核苷酸多态性越低。
D、Tajima’D:在标准中性进化模型下,Tajima’s D的理论值为零。
如果Tajima’s D>0,表明存在大量的中等频率的等位基因,这可能是由于群体瓶颈效应,群体结构,或者平衡选择引起的。
如果Tajima’s D< 0,表明存在大量的低频等位基因位点。Tajima’s D分析表示中性进化,越偏离0,受选择程度越高。
E、XP-CLR :全称是 the cross-population composite likelihood ratio test(跨群体复合似然比检验),是一种是基于选择清除(selective sweep)的似然方法。XP-CLR 利用了两个群体之间的多基因座等位基因频率差异(multilocus allele frequency differentiation)建立模型,使用布朗运动来模拟中性下的遗传漂移,并使用确定性模型来近似地对附近的单核苷酸多态性(SNPs)进行选择性扫描。
F: θπ值(驯化/野生)
G: Hp (杂合率) - 通常选择杂合率Hp较小,群体分化系数Fst较大的点即为候选位点