💥💥💞💞欢迎来到本博客❤️❤️💥💥
🏆博主优势:🌞🌞🌞博客内容尽量做到思维缜密,逻辑清晰,为了方便读者。
⛳️座右铭:行百里者,半于九十。
📋📋📋本文目录如下:🎁🎁🎁
目录
💥1 概述
📚2 运行结果
🎉3 参考文献
🌈4 Matlab代码、数据、文献
💥1 概述

头影测量分析用于评估面部生长,研究面部内的解剖关系。头影测量评估基于矢状面或冠状面的二维放射影像,是一种本质上不准确的方法。2D成像技术(如计算机断层扫描和磁共振成像)的广泛可用性使得面部形态的常规3D分析成为可能。3D头颅测量法不仅可以更准确地量化颅面形态和纵向生长,还可以区分咬合的细微变化。然而,用于计算颅面对称性和量化颅面形态的可靠协议仍然是一个广泛研究的主题。在这里,提出并评估了一种用于识别自然头部位置(NHP)和准确量化面部生长和面部不对称的3D头影测量分析协议。进行了一项幻影研究,以评估该方案的性能,并量化重复可靠地将头骨与 NHP 对齐的能力,并量化可以测量面部生长和面部不对称的准确性。
头影测量分析用于评估面部生长,研究面部内的解剖关系,并作为正畸和颅颌面畸形手术治疗计划的常规工具。标准的头影测量评估基于在矢状面(侧头颅造影)或冠状平面(头后前位图)中拍摄的二维放射影像,其中识别多个标志、线条和角度以量化面部的垂直和水平关系。该分析主要取决于标准化头部位置的准确且可重复的定义,因此患者在定制的图像采集系统中成像[2]。无论筛查方案如何,头影测量图像都可能受到放大伪影、颅面不对称和解剖结构叠加的影响,所有这些都会导致面部形态的传统评估方法不精确和不准确。
二维头影测量是一种本质上不准确的方法,涉及面部生长小增量的量化和横向维度的分析,特别是面部不对称性的分析。由于图像捕获、图像质量和地标识别方面的变异、不一致和错误,以及观察者内部和观察者之间的可靠性和再现性有限,当代头影测量分析的准确性进一步复杂化[2]。无法检测颅面形态的微小变化限制了该技术的临床应用,并阻碍了其在纵向生长的量化或闭塞细微变化的区分等领域的研究有用性,例如尖间和回缩的接触位置。迄今为止,没有任何技术可以提供牙齿咬合的精确虚拟再现[2],研究模型和面弓记录是目前牙科中再现咬合间关系的黄金标准。
3D成像技术的广泛可用性,如计算机断层扫描(CT),锥形束CT(CBCT)和磁共振成像(MRI),使得面部形态的常规3D分析成为可能。与传统的头颅测量法相比,三维重建头颅测量法具有几个优点。它避免了对头部进行标准化固定的需要,并允许对解剖头部位置进行后验定义。它还可以防止成像过程中固有的结构放大和变形,同时可以全面评估3D颅面形态,这对于术前评估、手术计划和术后评估非常重要[3]。1D成像技术有可能提供显着的精度改进,但是将成熟的3D概念准确转换为2D框架需要进一步发展。3D头颅测量法的研究显示结果相互矛盾,一些研究得出结论,它提供了更准确的颅面解剖学评估[3,4,5],而另一些研究则认为这两种技术之间没有统计学差异[6,7],或者不如8D技术准确[2]。
3D头影测量评估的最关键方面是颅骨在空间中的准确定位,需要准确定义颅面对称平面和头部的适当解剖框架。在侧头颅图的二维分析中,通过识别真正的水平或真正的垂直方向来定位头骨,并识别S,作为图像叠加和比较的参考点。法兰克福水平(FH)平面用于在某些特定分析中定向侧头影图像,需要识别两个双边地标(porion,Po和orbitale,Or),由于人脸的自然不对称性,它们与使用2D成像评估的不在同一平面上。此外,与FH相关的地标是最主观的地标之一,因此容易被错误识别[3,5]。另一种方法是定义真正的垂直方向,例如Nasion-Pogonion线[10]。这种方法的优点是基于对鼻(N)和pogonion(Pog)的识别,它们也比Po和Or[11]主观性小,可以认为位于头部的对称平面上。这些地标提供有关面部中线结构的直接信息,并且不会因面部不对称而受到解剖学变化的影响。此外,在儿童和青少年期间,颅面生长的特征是颅底和大孔相对稳定,与颅穹窿的显着扩张以及上颌骨和下颌骨的远端和向前生长相比,颅底和大孔保持相对不变[5,1,12]。在13D环境中,碱性颅可以为进一步的颅面生长评估提供可靠的参考结构[3]。
开发一致且可靠的 3D 头影测量工作流程将为临床医生提供有价值的工具,用于面部生长的 3D 表征和量化、评估牙颌面关系和随后解剖学上准确的正畸和手术计划,以及评估治疗结果。我们提出了一种准确估计颅面对称性的协议,使用随机颅骨方向、面部生长载体和不同类型的面部不对称性来评估这一点。测试的原假设是分析面部生长和不对称性的3D方法无法可靠地量化。另一种假设是,所提出的3D算法可以准确量化颅面对称平面,面部生长和面部不对称。
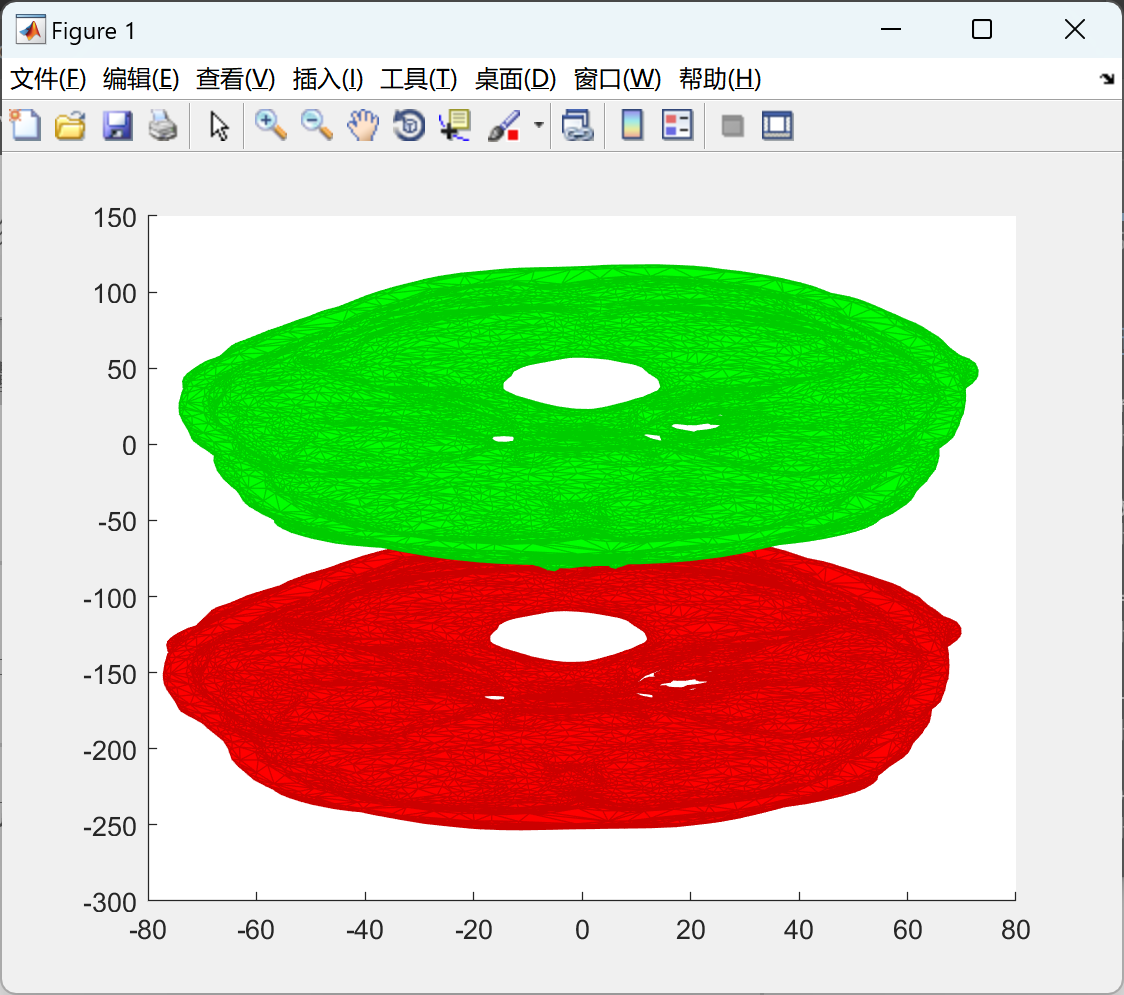
📚2 运行结果

部分代码:
function [v, f, n, name] = stlReadAscii(fileName)
%STLREADASCII reads a STL file written in ASCII format
%V are the vertices
%F are the faces
%N are the normals
%NAME is the name of the STL object (NOT the name of the STL file)
%======================
% STL ascii file format
%======================
% ASCII STL files have the following structure. Technically each facet
% could be any 2D shape, but in practice only triangular facets tend to be
% used. The present code ONLY works for meshes composed of triangular
% facets.
%
% solid object_name
% facet normal x y z
% outer loop
% vertex x y z
% vertex x y z
% vertex x y z
% endloop
% endfacet
%
% <Repeat for all facets...>
%
% endsolid object_name
fid = fopen(fileName);
cellcontent = textscan(fid,'%s','delimiter','\n'); % read all the file and put content in cells
content = cellcontent{:}(logical(~strcmp(cellcontent{:},''))); % remove all blank lines
fclose(fid);
% read the STL name
line1 = char(content(1));
if (size(line1,2) >= 7)
name = line1(7:end);
else
name = 'Unnamed Object';
end
% read the vector normals
normals = char(content(logical(strncmp(content,'facet normal',12))));
n = str2num(normals(:,13:end));
% read the vertex coordinates (vertices)
vertices = char(content(logical(strncmp(content,'vertex',6))));
v = str2num(vertices(:,7:end));
nvert = size(vertices,1); % number of vertices
nfaces = sum(strcmp(content,'endfacet')); % number of faces
if (nvert == 3*nfaces)
f = reshape(1:nvert,[3 nfaces])'; % create faces
end
% slim the file (delete duplicated vertices)
[v,f] = stlSlimVerts(v,f);function [v, f, n, name] = stlReadAscii(fileName)
%STLREADASCII reads a STL file written in ASCII format
%V are the vertices
%F are the faces
%N are the normals
%NAME is the name of the STL object (NOT the name of the STL file)
%======================
% STL ascii file format
%======================
% ASCII STL files have the following structure. Technically each facet
% could be any 2D shape, but in practice only triangular facets tend to be
% used. The present code ONLY works for meshes composed of triangular
% facets.
%
% solid object_name
% facet normal x y z
% outer loop
% vertex x y z
% vertex x y z
% vertex x y z
% endloop
% endfacet
%
% <Repeat for all facets...>
%
% endsolid object_name
fid = fopen(fileName);
cellcontent = textscan(fid,'%s','delimiter','\n'); % read all the file and put content in cells
content = cellcontent{:}(logical(~strcmp(cellcontent{:},''))); % remove all blank lines
fclose(fid);
% read the STL name
line1 = char(content(1));
if (size(line1,2) >= 7)
name = line1(7:end);
else
name = 'Unnamed Object';
end
% read the vector normals
normals = char(content(logical(strncmp(content,'facet normal',12))));
n = str2num(normals(:,13:end));
% read the vertex coordinates (vertices)
vertices = char(content(logical(strncmp(content,'vertex',6))));
v = str2num(vertices(:,7:end));
nvert = size(vertices,1); % number of vertices
nfaces = sum(strcmp(content,'endfacet')); % number of faces
if (nvert == 3*nfaces)
f = reshape(1:nvert,[3 nfaces])'; % create faces
end
% slim the file (delete duplicated vertices)
[v,f] = stlSlimVerts(v,f);
🎉3 参考文献
文章中一些内容引自网络,会注明出处或引用为参考文献,难免有未尽之处,如有不妥,请随时联系删除。