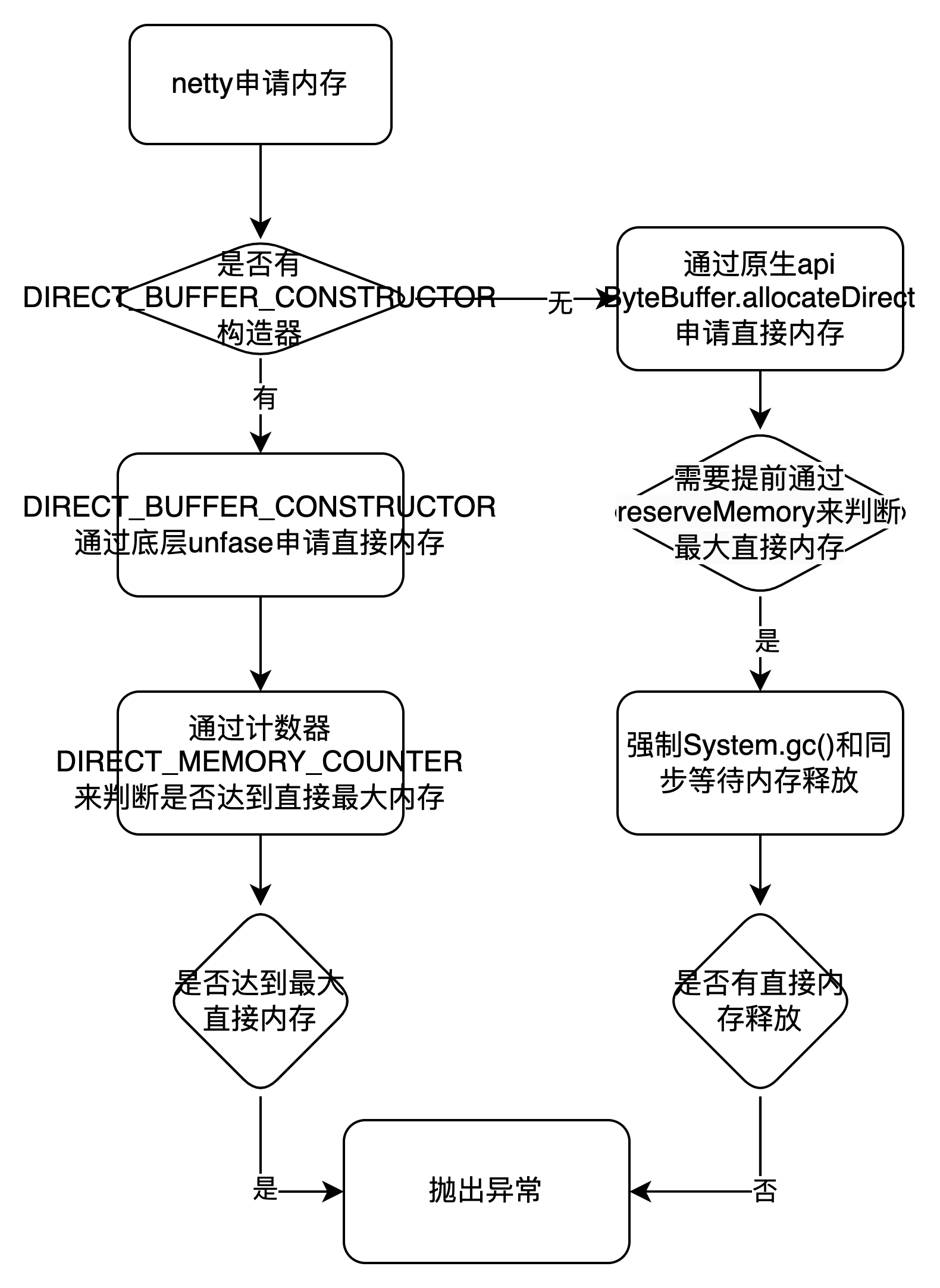
AutoDock Vina 1.2.0 对接计算(大批量)

AutoDockVina 1.2.0 的示例应用:A) 对接多个配体 (PDB 5x72);B) 使用 AutoDock4 (PDB 4ykq) 的水合对接方案与水分子对接;C)在锌存在的情况下使用 AutoDock4Zn 力场 (PDB 1s63); D) 柔性大环化合物。
1. AutoDock Vina 1.2.0 简介
AutoDock Vina 1.2.0 可以说是速度最快、使用最广泛的分子对接开源程序之一,是 AutoDock Vina 的升级版本。以下是 AutoDock Vina 1.2.0 的一些主要特点和功能:
-
多配体对接 (Simultaneous Multiple Ligand Docking)
Vina 现在能够同时对接多个配体。 这种功能可能会在基于片段的药物设计中得到应用,其中结合相同靶标的小分子可以生长或组合成具有潜在更好亲和力的更大化合物。 -
水合对接 (Hydrated Docking)
水合对接能够直接模拟配体-受体和水分子的相互作用。该方法基于与球形水分子显式水合的配体对接,可用于预测单个水分子的位置和作用(即桥接或置换),并总体上改进配体构象预测。 -
AutoDock4_Zn
AutoDock4_Zn是基于AD4的一种模拟锌离子配体的专用力场。基于使用假原子来描述复合在蛋白质中的锌离子的最佳四面体配位几何形状,以及改进电势的定义来描述其与配体中的配位元素(即氮、氧和硫)。该方法能够重现 AD4的对接性能,结果表明配体的晶体构象和最佳锌配位几何形状具有良好重叠。 -
大环构象采样 (Macrocycle Conformational Sampling)
大环化合物的对接是一项具有挑战性的任务,因为通过对导致不同构象的相关扭转变化进行建模来对环柔性进行采样很困难。AD4 有一个专门的协议来对接大环化合物,同时对其灵活性进行动态建模。 环结构中的一个键被破坏,形成大环的开放形式,从而进行独立探索扭转自由度。 在对接过程中,施加线性电势以恢复键合,从而形成闭合环形式。因此,在适应结合口袋的同时对大环构象进行采样,但代价是由于添加了额外的可旋转键而增加了搜索复杂性。 -
Python API
利用 Python 语言的流行度和实用性, AutoDock Vina1.2.0 版本中添加了Python API。为了生成尽可能符合 pythonic的 Python 接口,Vina 代码被重构为Python库。其大多数功能是通过直接绑定到现有的 C++ 代码来实现,或者通过附加函数来简化对 Python 环境的访问。提供的Python API具有以下功能:- 创建 AutoDock Vina 引擎的实例(评分函数选择、CPU 内核和随机种子)
- 读/写一个或多个PDBQT文件
- 计算Vina 亲和力
- 读/写 Vina 亲和力并读取 AutoDock 亲和力
- 随机化输入配体的方向和位置(randomize_only)
- 评估当前一个或多个姿势的能量(score_only)
- 执行局部优化(local_only)
- 设置蒙特卡罗全局搜索参数(详尽性、输出位姿数量、最大评估等…)
因此,基本的 Vina 计算可以配置并执行如下:
#!/usr/bin/env python
# Simple example with Vina Python bindings
from vina import Vina
v=Vina()
v.set_receptor("protein.pdbgt")ii
v.set_ligand_from_file("ligand.pdbqt")
v.compute_vina_maps([0., 0.,0.],[30,30,30])
v.dock(exhaustiveness=32)
v.write_poses("docking_results.pdbqt")
2. AutoDock Vina 1.2.0 安装
2.1 下载
根据自己的硬件环境,按需下载,官方下载网址。

2.2 Linux 安装
鉴于虚拟筛选任务需要进行大规模或超大规模的分子对接计算任务,最合适的选择是在Linux服务器上安装Vina。以下是安装、测试以及设置环境变量的步骤。
1. 安装
tar xzvf autodock_vina_1_1_2_linux_x86.tgz
2. 测试
./autodock_vina_1_1_2_linux_x86/bin/vina --help
输出help信息如下⬇
Input:
--receptor arg rigid part of the receptor (PDBQT)
--flex arg flexible side chains, if any (PDBQT)
--ligand arg ligand (PDBQT)
Search space (required):
--center_x arg X coordinate of the center
--center_y arg Y coordinate of the center
--center_z arg Z coordinate of the center
--size_x arg size in the X dimension (Angstroms)
--size_y arg size in the Y dimension (Angstroms)
--size_z arg size in the Z dimension (Angstroms)
Output (optional):
--out arg output models (PDBQT), the default is chosen based on the ligand file name
--log arg optionally, write log file
Misc (optional):
--cpu arg the number of CPUs to use (the default is to try to detect the number of CPUs or, failing that, use 1)
--seed arg explicit random seed
--exhaustiveness arg (=8) exhaustiveness of the global search (roughly proportional to time): 1+
--num_modes arg (=9) maximum number of binding modes to generate
--energy_range arg (=3) maximum energy difference between the best binding mode and the worst one displayed (kcal/mol)
Configuration file (optional):
--config arg the above options can be put here
Information (optional):
--help display usage summary
--help_advanced display usage summary with advanced options
--version display program version
3. 添加至环境变量
为了简化和方便使用vina,可以将其添加至环境变量。编辑~/.bashrc或~/.bash_profile等配置文件,然后添加vina的安装路径到环境变量。
#修改路径为vina的安装路径,最后将其写入.basrch文件中
export PATH=/home/.../autodock_vina_1_1_2_linux_x86/bin/:$PATH
注:前期的文件处理和后续的可视化分析可能会用到其他程序,建议安装ADFR,并将其也加入环境变量中。
3. 对接前受体配体处理
此部分需要用到ADFR,请自行下载安装后并将其加入至环境变量中。以下示例假设你位于包含受体receptor.pdbqt 和一组名为ligand_01.mol2、ligand_02.mol2 等的配体的目录中。
3.1 受体处理(PDB转为PDBQT格式)
在将受体的PDB格式转为PDBQT格式前,请务必确认受体是否质子化(从RCSB数据库中直接下载的结构缺乏质子化步骤)、是否有杂原子(溶剂、离子、辅因子和vina不支持计算的分子)、侧链缺失、loop缺失、结合位点的突变(病理性突变or其他)、水分子的保留与否等情况是否被一一考虑。我自己常用Schrödinger的Maestro中的Protein Prepare Wizard做蛋白的常态化处理,可以一次性解决质子化、侧链缺失、loop缺失、能量最小化等问题。
该脚本假定vina和ADFR已经在PATH环境变量中(参考2.2.3)。否则,请进行相应修改。
#! /bin/bash
prepare_receptor -r receptor.pdb -o receptor.pdbqt
#-r是输入的受体PDB格式文件
#-o是输出的受体PDBQT格式文件
3.2 配体处理(MOL2转为PDBQT格式)
还是那句老话,在转格式之前务必检查分子结构是否正确且检查质子化状态,是否是3D结构,否则需要2D转3D(参考Schrödinger中Ligprep小分子三维结构生成详解)。强烈建议不要使用 PDB 格式的小分子,因为它不包含有关键连接的信息。
目前,ADFR中的prepare_ligand可以将MOL2格式转换为PDBQT格式,通过for循环将所有小分子的格式批量转换为PDBQT格式。
该脚本假定vina和ADFR已经在PATH环境变量中(参考2.2.3)。否则,请进行相应修改。
#! /bin/bash
for mol2 in `ls ./*.mol2`
do
prepare_ligand -l ${mol2}
done
3.3 对接口袋参数生成
创建一个配置文件conf.txt,如下所示⬇
receptor = receptor.pdbqt
center_x = 2
center_y = 6
center_z = -7
size_x = 25
size_y = 25
size_z = 25
num_modes = 9
我自己通常通过PyMol插件GetBox-PyMOL-Plugin来生成口袋参数。在PyMol中安装GetBox-PyMOL-Plugin后,可以通过选中配体分子或指定的氨基酸来生成结合位点坐标。该插件不仅能够获取上述的vina 对接口袋参数,同时还能生成LeDock, AutoDock所需的对接口袋参数,对于需要多款分子对接软件交叉验证而言十分方便。
4. 大规模虚拟筛选
以下示例假设你位于包含受体receptor.pdbqt 和一组名为ligand_01.pdbqt、ligand_02.pdbqt 等的配体的目录(/home/…/vina_dock/)中。该脚本run_vina.sh假定 vina 已经在PATH环境变量中(参考2.2.3)。否则,请进行相应修改。
#! /bin/bash
cd /home/.../vina_dock/
for pdbqt in `ls ./*.pdbqt`
do
vina --config conf.txt --ligand ${pdbqt}
done
最终将产生ligand_01_out.pdbqt、ligand_02_out.pdbqt的对接后结果文件。
5. 结果处理
5.1 提取对接打分
对接完成后我们将对ligand_01_out.pdbqt、ligand_02_out.pdbqt文件中的对接打分提取进行过滤筛选。以下脚本可以提取每个小分子排名第一的对接打分。
#! /bin/bash
for i in `ls *_out.pdbqt`
do
name=`basename $i _out.pdbqt`
grep "REMARK VINA RESULT" -m 1 $i|awk -v id="$name" '{print id " " $4}'>>vina_score.txt
done
脚本功能:将每个小分子排名第一的对接打分保存在vina_score.txt文件中。
vina_score.txt的结果如下⬇
ligand_01 -10.267
ligand_02 -10.146
5.2 对接打分排序
按照vina_score.txt第二列打分值的升序排列,使用shell中的sort命令即可完成,也可将排序后的结果保存至vina_score_sort.txt文件中。
#结果排序
sort -k2,2n vina_score.txt
#保存排序后的结果
sort -k2,2n vina_score.txt >vina_score_sort.txt
5.3 提取小于一定值的打分结果
在一些有阳性或者阴性分子的打分结果作为对照的情况下,以他们的打分为阈值,我们可以通过过滤vina_score_sort.txt来获得更优分子的结果。
awk '$2 < -8' vina_score_sort.txt
5.4 对接后的构象拆分
以ligand_01_out.pdbqt为例,ligand_01_out.pdbqt包含了多个对接后的构象,他们按照打分的大小依次分布,通过使用vina的自带程序vina_split完成对对接后构象的拆分,以便后续的结果分析。该脚本假定 vina 已经在PATH环境变量中(参考2.1.3)。否则,请进行相应修改。
vina_split --input ligand_01_out.pdbqt --ligand ligand_01_split
将会获得拆分的单个构象:ligand_01_split1.pdbqt ligand_01_split3.pdbqt ligand_01_split5.pdbqt ligand_01_split7.pdbqt ligand_01_split9.pdbqt
ligand_01_split2.pdbqt ligand_01_split4.pdbqt ligand_01_split6.pdbqt ligand_01_split8.pdbqt
5.5 可视化分析
vina对接后的构象可以直接在MGLTools中的PMV中导入展示可视化,但对于我们更热衷于使用PyMol进行可视化分析的人来说,pdbqt格式无法在PyMol中直接加载展示,需要将其转换格式为MOL2、SDF等格式。格式转换可以直接调用ADFR中的babel。
babel -ipdbqt ligand_01_split1.pdbqt -osdf ligand_01_split1.sdf