nnUNet,这个医学领域的分割巨无霸!在论文和比赛中随处可见他的身影。大家对于nnUNet v1版本的教程都赞不绝口,因为它简单易懂、详细全面,让很多朋友都轻松掌握了使用方法。
最近,我也抽出时间仔细研究了nnUNet v2,并全程走过一遍流程。我努力将使用教程变得更加通俗易懂,希望能提高大家的学习效率。如果你正在进行分割任务,或者还没有尝试过nnUNet v2,那么现在是时候跟着我一起学习啦!
我们将一步步解析nnUNet v2的改进之处和操作步骤,用简洁明快的语言让你轻松理解。无论你是医学专业背景还是计算机科学领域的新手,都能够轻松上手,掌握这个强大的工具。
内容稍长,我把它分为三个章节,拆解难点
- nnUNet v2的不同之处,安装以及环境变量设置(这次内容)
- nnUNet v2的数据准备(下一篇)
- nnUNet v2的训练及推理(下下篇)
nnUNet v2版本与V1版有什么不同?
-
支持层次标签 ‘hierarchical labels’(在nnUNet中称为 ‘regions’)。例如,在 BraTS 这个数据集中,给定的 label 是“水肿”、“坏死”和“增强肿瘤”。但是对于临床应用而言,他们关注的不是这三个 label,而是整个肿瘤、肿瘤核心和增强肿瘤。 那么,这里的整个肿瘤就是 水肿+坏死+增强肿瘤; 肿瘤核心是 坏死+增强肿瘤。如果想按临床的这个进行分割,他的 label 是有重叠的。如下图:
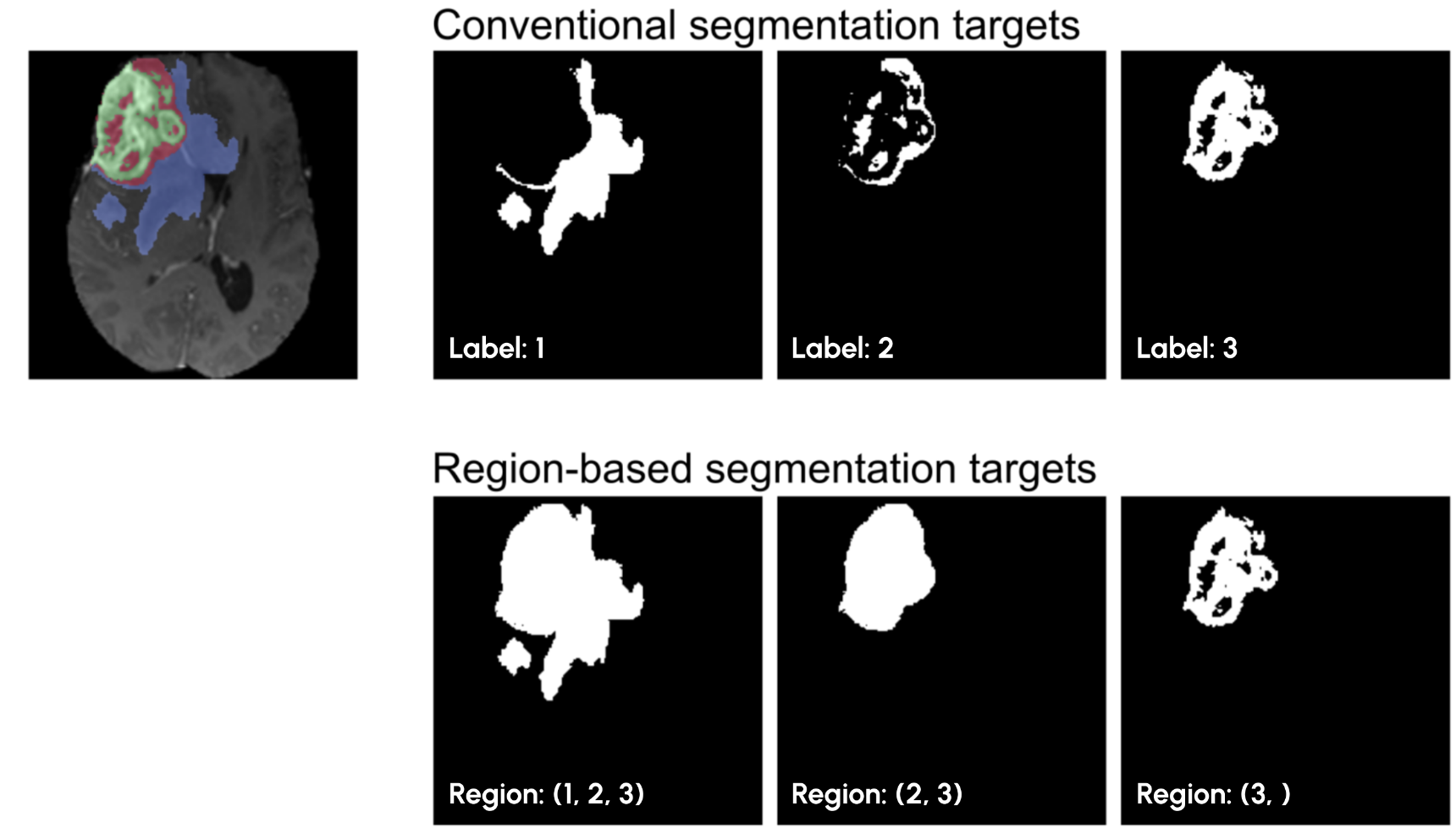
适用于某些有重叠任务的分割,具体详见 github -
支持跨平台, Cuda, mps (Apple M1/M2) and CPU
-
支持更多的输入/输出数据格式。
-
通过基于
BaseReaderWriter实现新的适配器,可以扩展I/O格式。 -
nnUNet_raw_cropped文件夹不再存在,这样可以节省磁盘空间,而且没有性能损失 -
在解压缩时,预处理数据和分割结果存储在不同的文件中。分割结果以int8的格式存储,因此每个像素占用的磁盘空间(和I/O吞吐量)只有v1版本的四分之一。
-
原生支持多GPU(DDP)训练。但是,多GPU推断仍然需要使用
CUDA_VISIBLE_DEVICES=X nnUNetv2_predict [...] -num_parts Y -part_id X来运行。在推断过程中,没有跨GPU的通信,所以增加DDP会增加额外的复杂性是没有意义的。 -
所有nnU-Net的功能现在也可以通过API进行访问。请查看
setup.py中对应的入口点,以了解需要调用哪些函数。 -
数据集指纹现在明确地创建并保存在一个json文件中(参见nnUNet_preprocessed文件夹)。
其实大改动不多,如果你只是一个初级用户,很多改进功能根本用不到。
如何安装
虽然 官方说 nnunet 的两个版本可以同时使用,但我还是建议将两个版本分开,放在不同的虚拟环境中。
安装需求
- 操作系统:nnU-Net 已在 Linux(Ubuntu 18.04、20.04、22.04;centOS、RHEL)、Windows 和 MacOS 上进行了测试!它应该开箱即用!
- 硬件要求:GPU(推荐)、CPU 和 Apple M1/M2 作为设备(目前 Apple mps 不实现 3D 卷积,因此您可能必须在这些设备上使用 CPU)。
- Python 3.9 或更高版本
在第一版本中,nnunet v1 安装教程介绍了如何在虚拟环境中安装 nnUNet, 但是略显复杂。这一次直接用 conda 安装
安装步骤
- 首先新建一个 conda 环境,命名为 nnunet2(名字由你决定)
conda create -n nnunet2 python==3.9
- 激活该环境,并安装 nnunetv2
conda activate nnunet2
pip install nnunetv2
设置环境变量/路径
nnU-Net 依靠环境变量来了解原始数据、预处理数据和训练模型权重的存储位置。要使用 nnU-Net 的完整功能,必须设置以下三个环境变量:
V1,v2 版本中路径有所不同。如果你已经装了 V1 版本,那么 V2 版本的路径最好和 V1不同,以防打架。
具体地,V2 应该包含以下三个路径:
-
nnUNet_raw:这是放置原始数据集的地方。该文件夹将为每个数据集名称创建一个子文件夹
DatasetXXX_YYY,其中 XXX 是 3 位标识符(例如 001、002、043、999…),YYY 是(唯一)
数据集名称。数据集必须采用 nnU-Net 格式树结构示例:
nnUNet_raw/Dataset001_NAME1 ├── dataset.json ├── imagesTr │ ├── ... ├── imagesTs │ ├── ... └── labelsTr ├── ... nnUNet_raw/Dataset002_NAME2 ├── dataset.json ├── imagesTr │ ├── ... ├── imagesTs │ ├── ... └── labelsTr ├── ... -
nnUNet_preprocessed:这是保存预处理数据的文件夹。数据也将从中读取
训练期间使用此文件夹。重要的是,该文件夹位于访问延迟低且高的驱动器上
吞吐量 -
nnUNet_results:指定 nnU-Net 保存模型权重的位置。如果下载了预训练模型,则此
就是保存他们的地方。
V1 版本的路径设置见之前的文章:给出了永久设置环境变量的方法
nnU-Net v1 如何安装–傻瓜式安装教程
注意:如果设置永久变量的话,V1和V2版本都安装了的话,其实有一个文件夹命名是相同的(nnUNet_preprocessed),那你调用的时候就分不清 V1和V2的文件夹了。
这里,再给一种更方便的临时路径设置办法
- step1: 创建一个 setPath.sh 文件,里面包含如下内容,
#!/bin/bash
export nnUNet_raw="/media/nunet2_folder/nnUNet_raw"
export nnUNet_preprocessed="/media/nnunet2_folder/nnUNet_preprocessed"
export nnUNet_results="/media/nnunet2_folder/nnUNet_results"
tips:怎么创建一个setPath.sh文件
要创建一个名为"setPath.sh"的文件,你可以按照以下步骤进行操作:
-
打开一个文本编辑器,例如记事本或者终端上的文本编辑器。
-
创建一个新的空白文件。
-
在文件中输入上述内容
-
保存并关闭文件。
当然,前提是,你要手动创建这个三个文件夹,再把地址告诉环境变量
- step2: 每次运行 nnunetv2 之前运行一下此文件
source setPath.sh
- step3: 检验是否起效
echo $nnUNet_results
如果返回的是我们设置的地址,则起效
V1和V2路径对比如下

好了,炼丹炉已就位。带上你的数据,下一篇进行数据预处理。

文章持续更新,可以关注微信公众号【医学图像人工智能实战营】获取最新动态,一个关注于医学图像处理领域前沿科技的公众号。坚持已实践为主,手把手带你做项目,打比赛,写论文。凡原创文章皆提供理论讲解,实验代码,实验数据。只有实践才能成长的更快,关注我们,一起学习进步~
我是Tina, 我们下篇博客见~
白天工作晚上写文,呕心沥血
觉得写的不错的话最后,求点赞,评论,收藏。或者一键三连