培训和测试样本
用于模型评估的数据集包括8652个F1杂交玉米样本,这些样本具有测量的抽穗天数(DTT)、株高(PH)和穗重(EW)表型,这些表型来自母体库和30名父系测试者小组的杂交,遵循北卡罗来纳-II设计(方法)。母体库是先前报道的立方(完全双列设计加上不平衡育种样杂交)群体,包含1428个近交系,由代表局部适应性等位基因的24个精英创始人系发展而来。父系库包含30个测试品系,涵盖6个主要杂种优势群,大部分是代表外来优势等位基因的改良海外种质。因此,该群体由30组父系半同胞亚群体(简称F1s)组成,表现出不同模式的杂种优势效应。说明了30个F1亚群的数据结构,用于设计不同的预测框架,以客观地评估模型的精度和稳定性。8652个样本包含两个完整的F1群体,由1428个母系和2个父系测试系郑58和京724之间的2856个杂种组成。207个母系品系和30个父系测试者的杂交产生了6210个F1,由于母系创始人和父系测试者的不同遗传起源,在基因型上表现出强群体分层(图1b,附加文件1:图S1)。为了消除亚群之间表型变异的系统偏差,表型值被标准化为每个F1群体内的z分数,相对排名用于表示绝对值。由于一组非冗余特征对于ML避免维数爆炸至关重要,因此均匀分布在基因组中的32,559个单倍型标签单核苷酸多态性(SNPs)被用作基因型特征(方法)。
基础ML模型的评价
为了确保对GS中的ML方法进行客观的评估,我们遵循了构建ML系统的过程,该系统包括特征选择、模型选择、模型评估、模型优化、特征重要性分析、模型解释,最后是软件基准测试。我们首先使用1428个母体系(方法)的数据集,在rrBLUP和其他15个先前发表的GS工具之间进行了一系列基准测试。rrBLUP表现出优越的预测精度和计算效率,然后被选为统计模型的代表,与ML方法进行比较(附加文件1:图S3)。由于不同的ML算法可能适用于不同的预测目标和数据特征,我们选了五个常用的基础ML模型进行初步评估,并将其与rrBLUP进行比较,即支持向量回归(SVR)、随机森林(RF)、人工神经网络(ANN)、k-最近邻(KNN)和梯度增强(GB)回归树。五个MLs模型的评估是在由网格搜索功能调整的最佳超参数下进行的(附加文件1:表S1)。具有现场测量表型的6210个F1主要用于评估模型精度。使用以下框架进行交叉验证(CV),重复30次,以生成每种方法的精度分布(r,Pearson相关系数):即29个F1亚群作为训练样本,使用32,559个SNP作为特征预测其余亚群。由于GB和rrBLUP的精确度相当,并且都优于其他四种ML方法(图1c),因此选择GB与rrBLUP进行进一步比较。
具有不同样本划分方式的预测框架可能会影响模型的稳定性,并且由于总体分层,不适当的划分可能会导致不正确的过度拟合。特别是对于主要依靠远亲杂种优势群体杂交的玉米杂交育种,预测框架是确保模型稳定性的关键因素。为了解决这个问题,使用四种不同的框架将训练与测试样本的比例设置为5:1:当训练样本覆盖测试样本中母亲和父亲(M和P)兄弟姐妹、仅母亲(M)兄弟姐妹、仅父亲(P)兄弟姐妹和父母(都不是)兄弟姐妹的基因型时(图2a)。如图2b所示,覆盖两种亲本基因型的训练样本表现出最高精度,其次是仅覆盖母体基因型的训练样本;当训练样本仅覆盖父系基因型或两种亲代基因型都不覆盖时,精确度会大大下降。特别是对于EW,当父母都不在训练样本中时,精确度下降了近两倍。然后,我们评估了两组完全表型化的郑58和京724 F1的模型稳定性,使用一个群体训练模型并预测另一个群体。rrBLUP的精确度略好于GB,可能是因为母系系是来自具有明确系谱的相同立方群体的密切相关的兄弟姐妹(图2c)。
模型精度评估
基于不同的预测目标和训练样本与测试样本的划分,我们使用不同的CV方法来评估模型的精度。相应图中使用的用于比较rrBLUP和LightGBM的统计检验的显著性水平(p值)在附加文件1:表S5中给出。
(1)为了使用6210个(207个母系× 30个父系)F1评估五个基础ML模型(图1c),29个F1群体被用作训练样本来预测F1群体的其余部分。重复该程序30次,以测试所有30个F1群体的精确度,从而可以为每个模型生成精确度分布(r,预测和测量表型之间的Pearson相关性)。
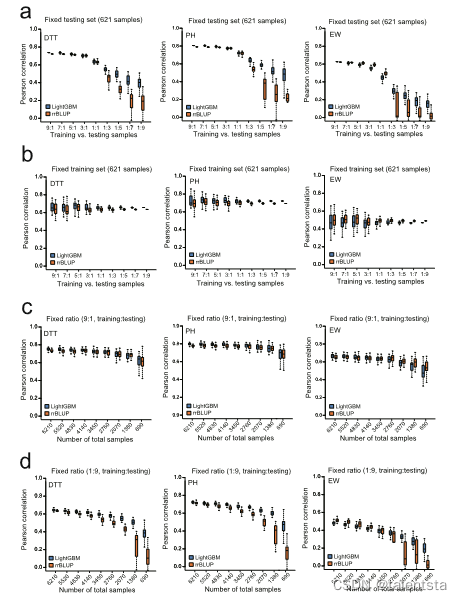
(2)为了评估训练数据的采样率对预测精度的影响,使用6210个f1的总体设计了四个场景,即测试集的固定大小(621个样本)、训练集的固定大小(621个样本)、9:1的固定比例(训练与测试)和1:9的固定比例(训练与测试)(图4)。对于前两个场景,设置了9个比率(9:1、7:1、5:1、3:1、1:1、1:3、1:5、1:7和1:9)的训练和测试,然后以每个比率随机构建训练和测试集30次,以生成LightGBM和rrBLUP的精度分布。对于最后两种情况,9个人口规模,固定比例为9:1和1:9,然后在每个尺寸下随机构建训练和测试集30次。
(3)为了评估亲本组成对预测的影响(图2b),将6210个F1分成30个亚组,其中25个用作训练样本来预测其余5个亚组中的F1。重复该程序30次,以生成精度分布。

(4)为了评估不同F1群体之间的精确度(图2c),首先使用1428个郑58 F1作为训练样本来预测1428个京724 F1;然后,首先使用1428个京724 F1作为训练样本来预测1428个正58 F1。

(5)为了评估不同GB变体的模型拟合能力(图3a),具有测量表型的8652 F1被用于首先训练模型,然后生成8652 F1的预测表型。随后,计算8652个f1的预测和测量表型之间的皮尔逊相关性,以代表每个模型的拟合能力。

(6)为了比较有和没有亲本表型作为附加特征的LightGBM和rrBLUP之间的精度(图3c),首先使用6210个F1s+1221个郑58 F1s作为训练样本来预测1221个京724 F1s然后,首先用6210个F1+京724个F1作为训练样本来预测1221个正58个F1。

(7)为了测试由LightGBM执行的分类任务(图5),用早期、中度和晚期DTT(EW的GCA)标记1428个母体系,并执行五倍CVs来训练模型,随后生成ROC曲线和AUC值来表示精度。为了测试rrBLUP的分类任务(附加文件1:图S6),根据观察到的表型,首先用早期、中度和晚期DTT(EW的GCA)标记1428个母体系。在rrBLUP预测后,根据预测表型的值对品系进行重新标记,随后生成ROC曲线和AUC值来表示精确度。

(8)为了评估通过LightGBM进行特征选择的有效性(图6a),将1428个母系品系中80%的取样重复30次,并在每次重复时计算IGs,以选择高效的SNPs来重新训练LightGBM,随后预测其余20%品系的表型。

熵是反映一组样本分散程度的数学指标。低熵值表示样品分散程度低,由于决策树以叶节点表型的平均值作为预测值,训练样本叶节点的熵越低,预测值的精度越高。
在LightGBM的特征重要性分析中,信息增益(IG)值用于表示分离样本前后熵的变化,这可以反映树节点(SNP)在区分表现出强表型差异的样本的两个分支方面的有效性。因此,具有高IGs的SNPs表明在分类样品中具有高功效。在模型训练期间完成树构建的所有迭代之后,IG值对于每个SNP,求和以代表其特征重要性,这可能反映了其在基因型和表型之间关联的能力。为了从测试样本中排除SNP效应的影响,首先由LightGBM在80%行的每次采样中计算IGs作为训练集,并根据IGs总和从高到低的排序,选择前12、24、48、96、192、384、1000、2000、3000和4000个SNP作为重新训练LightGBM的特征。然后用该模型预测其余20%品系的表型。重复取样30次,生成30个皮尔逊相关性,用于绘制每组SNP的精度分布图。同时,对从32,559个SNP中随机取样的相同数量的SNP应用相同的程序,以绘制精度分布图作为对比。最后,使用32,559个SNPs对1428个母系品系进行rrBLUP,以计算三个性状的基线精度。
基因型特征向数字特征的转换
对于rrBLUP和机器学习工具,应该首先根据训练和测试群体中每个SNP的等位基因频率将基因型特征转换为数字特征。当使用0,1,2编码方案时,两个主要等位基因的纯合基因型(AA)编码为0,一个主要等位基因和一个次要等位基因的杂合基因型(AB)编码为1,两个次要等位基因的纯合基因型(BB)编码为2。除了0,1,2编码方案之外,LightGBM可以采用替代的0-9编码方案来表示基因型的所有形式,例如在CropGBM中使用的转换规则,如下所示:AA(0),AT(1),TA(1),AC(2),CA(2),AG(3),GA(3),TT(4),TC(5),CT(5),TG(6),GT(6),CC(7),CG(8),GC(8),GG(9)。
一般配合力的计算
一般配合力(GCA)是基于其与其他品系杂交行为的自交系的平均值。计算如下:

其中GCAi代表亲本i的GCA,Xi是i亲本杂交后代的平均值,X是所有杂交后代的总体平均值。