没错,这是真的,沉积十年之后,多样性研究中最经典的16S数据库——Greengenes数据库,竟!然!更!新!了!惊不惊喜!意不意外!
遥想当年小编还是一个小白研究生的时候,被师兄带入门接触到多样性研究,Greengenes数据库还是当时最火最核心的数据库,结果后继无力,被后浪拍在了沙滩上……
时隔十年,本次更新的Greengenes2 2022.10版本数据库提供了Qiime2兼容插件以及分析流程,最重要的是它还将继续更新,快用起来呀!


图 微生物多样性16s rRNA物种注释常见的数据库有Silva数据库、RDP数据库以及Greengenes数据库。
微生物多样性16s rRNA物种注释常见的数据库有Silva数据库、RDP数据库以及Greengenes数据库。其中,Silva数据库因其拥有高质量的序列数据、广泛的物种覆盖范围、更新频率快等优势,近些年来被科研人员广泛使用。但是,由于缺乏标准化的分类系统,注释结果中经常会出现注释错误或注释信息不准确的现象,使得结果的比较和解释具有一定的困难性。
而在这点上,Greengenes数据库使用了一套标准化的分类系统,可以使得不同研究之间的微生物分类结果更加一致和可比较,使不同研究之间的微生物分类结果更加可靠和可重复。但是由于其更新频率太低,更新持续停留在2013年5月更新的gg_13_5版本,让无数科研人员爱而不得,望眼欲穿!
但是,今年它终于更新了!!!数据库全新升级,称之为Greengenes2,并且相关介绍已见刊于《nature biotechnology》。

全新升级之后的Greengenes2数据库,依然保留了GTDB的分类系统,包括其多系标签。宏基因组学和16S rRNA扩增子广泛用于微生物组研究,但两种方法之间缺乏标准化结果难以调和,限制了微生物组对可重复生物标志物发现的效用。本次更新后的Greengenes2数据库允许直接整合16S rRNA和宏基因组数据集,将其统一在一个参考树中,分析结果表明,由相同样本生成的16S rRNA和宏基因组数据在主坐标空间、分类学和表型效应大小方面一致。
Greengenes2的系统发育覆盖率远大于过去的资源(图1b),如SILVA、Greengenes和GTDB。过去在比对16s和宏基因组数据集上进行的努力仍然导致分布不重叠,只有像Procrustes分析这样的技术才能显示结果之间的关系,而Greengenes2树和UniFrac(一种系统发育的方法)一起使用提供了更好的一致性(图1f)。
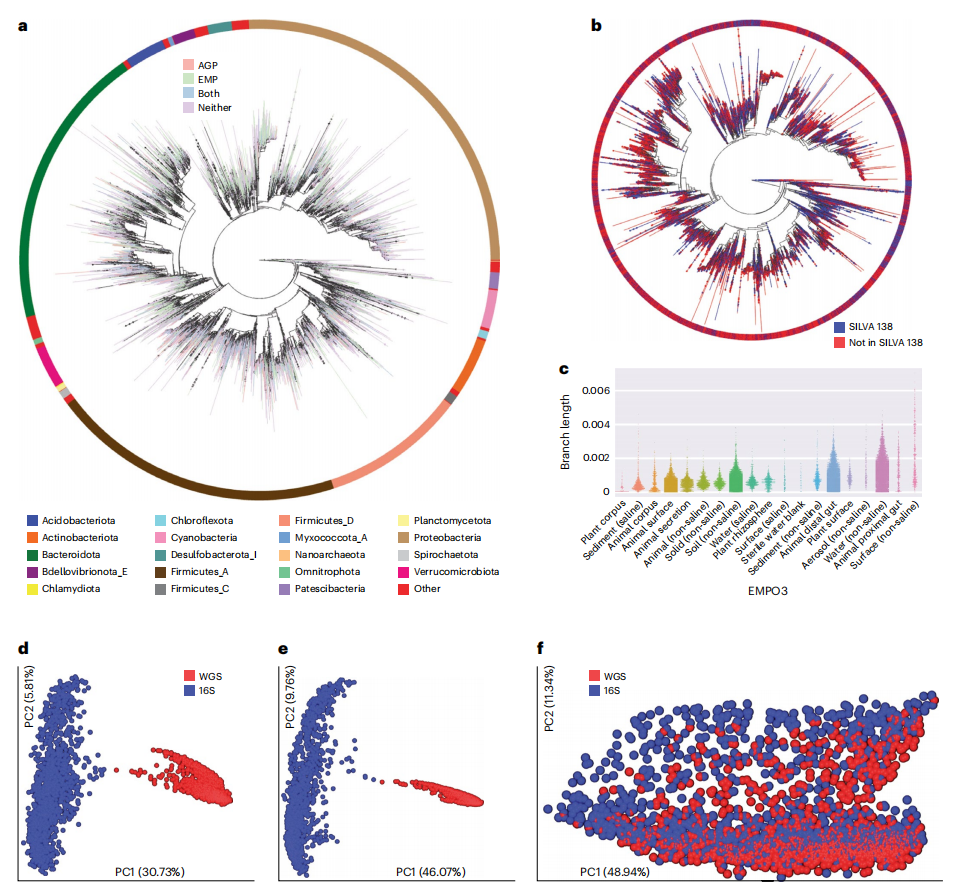
图1 Greengenes2总览
此外,对于16S rRNA基因V4区域的研究,可以直接从系统发育中获得分类结果,无需使用朴素贝叶斯方法,并且可能产生更高的分辨率结果。同时,相较于Silva数据库,Greengenes2在属水平(Pearson r = 0.85)和物种水平上(Pearson r = 0.65)提供了良好的一致性(图2)。
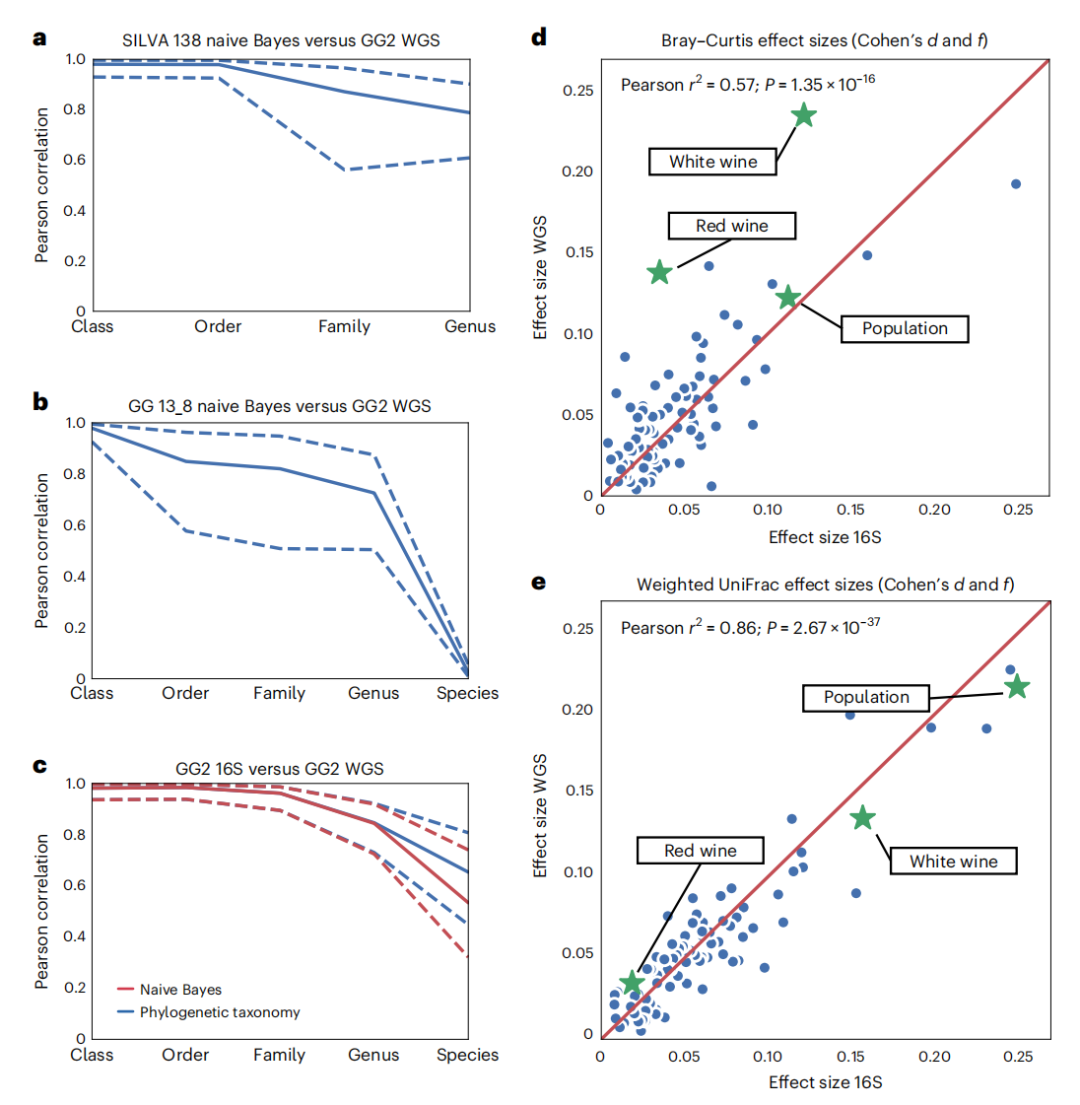
图2 16S rRNA ASV与宏基因组数据的分类和效应大小一致性
综上所述,时隔多年,重新回归的Greengenes2更加全面,使用一致的、综合的分类资源显著提高了使用不同数据类型的微生物组研究的可重复性,并允许在不同人群中可靠地确定影响微生物组变化的变量,这将会更加满足科研人员的需求。
参考文献
[1] Greengenes2 unifies microbial data in a single reference tree. nature biotechnology, 2023.