疾病建模和靶点识别是药物发现中最关键的初始步骤。传统的靶点识别是一个耗时的过程,需要数年至数十年的时间,并且通常从学术报告开始。鉴于其分析大型数据集和复杂生物网络的优势,人工智能在现代药物靶点识别中发挥着越来越重要的作用。该综述回顾了靶点发现的最新进展,重点关注人工智能驱动的靶点发现。目前,越来越多的人工智能识别靶点正在通过实验得到验证,多种人工智能衍生药物正在进入临床试验,标志着人工智能驱动药物发现新时代的到来。
来自:AI-powered therapeutic target discovery
目录
- 靶点识别概述
- 靶点识别策略:从实验到机器学习
- 基于实验
- 基于多组学
- 基于计算
- AI驱动的靶点识别
- 靶点发现的应用
- 使用AI合成数据进行靶点识别
- 靶点选择标准
靶点识别概述
药物发现被广泛认为是一个耗时、昂贵且充满风险的过程,通常需要大约10年时间和20亿美元才能将新药推向市场。到 2022 年,已确定的成功药物靶点不足500个,仅占人类估计可用药物靶向的一小部分。尽管众多候选药物在临床前阶段进行了广泛的优化,但2009年至2018年临床试验的平均失败率高达84.6%。 缺乏临床疗效仍然是导致2期和3期试验失败的关键因素,其导致巨大的财务损失和资源浪费。因此,确定正确的药物靶点对于增加开发临床有效疗法的可能性至关重要。
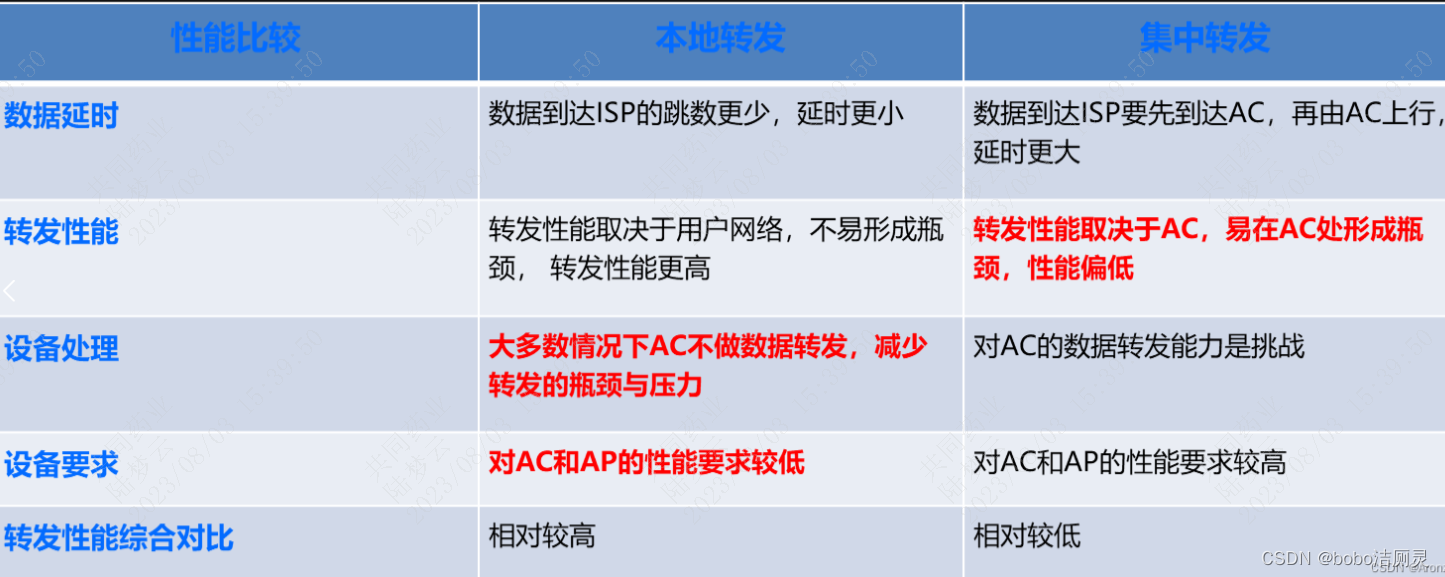
靶点识别是识别可以通过药物调节以实现治疗效果的正确生物分子或细胞途径的过程,在现代药物发现中变得越来越重要。 尽管过去几十年来实验和组学技术的创新不断增长,但确定可行的治疗靶点仍然具有挑战性。多组学数据与人工智能的集成最近已成为一种有前途的靶点识别方法(图1-up)。

- 图1-up:靶点识别历史上的关键技术进步分为三类:基于实验的(红色)、多组学(蓝色)和计算(绿色)方法。传统上,基于实验的方法一直是发现治疗靶点的首选方法。 然而,随着大数据的兴起,多组学数据的集成分析已成为更有效的靶点识别策略。此外,人工智能驱动的生物分析的最新进展已经确定了新的靶点,人工智能设计的药物现已进入临床试验。
- 缩写:AGC chemistry,亲和力引导催化剂化学(affinity-guided catalyst chemistry);ALS,肌萎缩侧索硬化症(amyotrophic lateral sclerosis);DL,深度学习;EGFR,表皮生长因子受体(epidermal growth factor receptor);GAN,生成对抗网络;GWAS,全基因组关联研究(genome-wide association study);LD chemistry,配体引导化学(ligand-directed chemistry);MTOR,雷帕霉素的哺乳动物靶标(mammalian target of rapamycin);NSCLC,非小细胞肺癌(non-small cell lung cancer);SILAC,细胞培养物中氨基酸的稳定同位素标记(stable isotope labeling with amino acids in cell culture);TID,靶点识别。

- 图1-low:人工智能在药物发现早期阶段的应用。
术语:
- Biomarker:任何类型体液或组织中作为生物状态标志的生物分子。
- Drug repurposing:利用已获得 FDA 批准或针对特定医学适应症进行临床研究的现有药物,再次确定新的治疗应用的过程。
- Drug–target interaction:药物发现的重要一步,识别化合物和蛋白质靶点如何在人体内相互作用。
- Genome-wide association study,GWAS:一种通过比较患有和不患有特定疾病或性状的人之间基因组变异的频率来识别与某种疾病或性状风险统计相关的基因组变异的方法。
- Indication prioritization,指示优先级:基于药物的预期相关性和使用人工智能的特定适应度对药物进行优先排序的过程。
- Induced pluripotent stem cells,iPSCs,诱导多能干细胞:通过特定多能性相关基因(即 c-Myc、Oct3/4、Sox2 和 Klf4)的共表达,从成体体细胞产生人工干细胞。
- Pharmacokinetics,药代动力学:研究化合物在生物体中的命运,即吸收、分布、代谢和排泄。
- Therapeutic modality,治疗模态:用于治疗疾病或医疗状况的疗法类型,包括小分子药物、基于蛋白质的疗法、先进疗法(例如细胞和基因疗法)和基于微生物的疗法。
靶点识别策略:从实验到机器学习
靶点识别可以分为三种不同的策略:实验方法、多组学方法和计算方法(图 2)。联合使用这些方法可以在探索靶点识别中产生新的治疗假设,从而显着增强我们对复杂疾病的理解。

- 图2:靶点识别的三种探索性策略:基于实验方法、多组学方法和计算方法。
- 实验方法包括进行湿实验,以根据亲和力、基因修饰筛选和比较分析来识别靶点。
- 多组学方法通过分析各种组学数据集(例如基因组学、转录组学、蛋白质组学、表观基因组学和代谢组学)来预测基因与疾病的关联。
- 计算发现方法通过使用机器学习或基于结构的方法(包括反向对接reverse docking、药效团筛选pharmacophore screening,结构相似性分析structure similarity analysis)有效地识别潜在靶点。
基于实验
自 20 世纪 60 年代以来,实验方法(包括基于亲和力的生化实验、比较分析和遗传筛选)已证明其对靶点识别的显着贡献。
使用小分子亲和探针(small-molecule affinity probes)可以在配体-蛋白质相互作用时进行无痕蛋白质标记,探针法是三种实验方法中最直接的方法。探针的选择高度依赖于起始分子的特性。
细胞培养中氨基酸稳定同位素标记 (SILAC) 是比较分析的一个例子,是一种流行的定量蛋白质组学工具,它使用稳定同位素标记的氨基酸来准确区分细胞蛋白质组。针对肝癌细胞HCC、多发性骨髓瘤、子宫内膜癌和结直肠癌等多种癌症类型进行的研究清楚地证明了SILAC在识别关键参与者方面的有效性。
数十年来,通过RNA干扰(RNAi)或CRISPR-Cas9基因编辑实现的基因筛选一直引起生物学家的极大兴趣。由于其高特异性和效率,CRISPR极大地扩展了我们对人类疾病的机制和药理学方面的了解。例如,通过靶向CRISPR干扰筛选,BRD2 被确定为宿主对 SARS-CoV-2 感染反应的重要调节因子。CRISPR 技术仍在不断发展,进一步增强了其灵活性、简单性和效率,从而为研究界带来了巨大的好处,不仅可以用于靶标识别,还可以作为基因治疗和诊断工具。
基于多组学
多组学数据为研究人员提供了不同角度的相互关联的分子信息,包括静态基因组数据和时空动态表达和代谢谱。作为最成熟的组学学科,基因组学(genomics)专注于DNA序列中的遗传变异。由下一代测序支持的大规模全基因组关联研究 (GWAS) 分析已产生遗传变异与复杂疾病或性状之间的数十万个关联,从而引领囊性纤维化调节剂等突破性疗法的开发,比如针对疾病相关基因IL23A的治疗炎症性肠病的新药。最近,对已发表的 GWAS 数据的分析揭示了可归因于不同疾病的新遗传位点,从而开辟了药物重新利用的机会。
尽管基因组一直是靶点识别中不可或缺的因素之一,但区分导致特定疾病的致病基因变异仍然具有挑战性。在这方面,整合多个组学信息是有用的。转录组和蛋白质组数据可用于识别调节基因和蛋白质水平的因果遗传位点,并促进发现疾病发病机制的基因和途径。同样,表观基因组和代谢组数据也可以作为 GWAS 识别的变异的功能证据,以支持其疾病关联和临床应用。与单组学方法相比,综合多组学分析可以提供更全面的疾病机制视图,因此越来越多地用于促进生物标志物和治疗靶标的发现、治疗反应和患者预后预测。
基因组和转录组的区别:
-
基因组(Genome):基因组是指生物体内所有遗传信息的总和,包括所有的DNA序列和基因。它代表了一个生物体的全部遗传信息,包括编码蛋白质的基因以及非编码区域等。基因组通常被描述为一个生物体的遗传蓝图,它决定了生物体的所有特征和功能。
-
转录组(Transcriptome):转录组是指在一个特定的细胞或组织中,所有转录产物(mRNA)的总和。它代表了基因组中正在被转录和表达的基因的信息,即基因表达的情况。转录组可以反映细胞或组织的基因表达水平,包括哪些基因在特定条件下被表达,以及它们的表达量。
在基因组中,每个生物体都包含一套完整的DNA序列,但在不同细胞或组织中,只有一部分基因会被表达成转录产物(mRNA),从而决定了细胞的特定功能和表型。因此,基因组和转录组是两个不同层次的遗传信息,前者代表了所有的遗传信息,后者则代表了当前生物体在特定条件下的基因表达情况。
基于计算
由于典型的基于实验的靶点识别是费力且资源密集型的,因此计算方法已成为实现有效靶点筛选的替代方案。根据蛋白质结构的可用性和感兴趣的化合物的化学结构,药效团筛选、反向对接和结构相似性评估已被用来预测小分子的新生物靶点。另一方面,人工智能是靶点发现计算科学中一门不断发展的学科。机器学习是人工智能不可或缺的组成部分,可以在有监督或无监督的情况下应用。监督学习利用标记数据集来训练模型以进行数据分类和可靠的结果预测。相比之下,无监督学习无需人工干预即可探索未标记数据的隐藏结构。机器学习的应用不限于预测现有药物或化合物的生物靶点,还可以识别任何感兴趣的疾病的新治疗靶点。
AI驱动的靶点识别
近年来,生物医学数据爆炸式增长,包括从疾病机制的基础研究到患者的临床研究。数据的增长也给数据分析带来了挑战。鉴于人工智能在处理复杂生物医学数据方面的优势,使用AI可以揭示数据中人类可能发现不了的模式和关系,并可能有助于更好地理解和治疗疾病。人工智能在促进生物标志物和靶点识别、适应度优先排序、类药分子设计、药代动力学预测、药物-靶点相互作用和临床试验设计方面发挥作用。尽管仍处于临床试验的早期阶段,人工智能衍生药物越来越多地出现在临床研究中,例如用于治疗非酒精性脂肪性肝炎的 GS-0976、用于治疗实体瘤的 EXS-21546 和用于治疗特发性肺纤维化的 INS018_055,这是第一个在1期临床试验中取得积极结果的 AI 衍生药物。
靶点识别的结果可以是基因、蛋白质,或者其他生物分子。具体取决于研究的目标和所使用的方法。
- 在基因组学和转录组学中,靶点识别通常涉及寻找与某个特定性状或生物过程相关的基因。这些基因可能参与特定的生物学过程,或者是影响特定性状的遗传因素。
- 在蛋白质组学中,靶点识别可能涉及寻找与特定生物过程或疾病相关的蛋白质。这些蛋白质可能具有重要的生物学功能,如酶活性、信号传导、细胞结构等。
- 除了基因和蛋白质,靶点识别的结果还可以是其他生物分子,例如非编码RNA、代谢产物等,这取决于研究的具体内容和分析的数据类型。
药物可以靶向基因或除了蛋白质的其他生物分子。传统上,药物的靶点主要集中在蛋白质上,因为蛋白质在生物体内扮演着各种重要的功能角色,如酶活性、信号传导、细胞结构等。因此,大多数药物是通过与蛋白质相互作用来发挥其生物学效应的。然而,近年来,随着生物技术和药物研究的不断发展,科学家们逐渐认识到药物还可以靶向其他生物分子,包括基因和非编码RNA等。例如,一些药物可以通过干扰或调控特定基因的表达来实现其治疗效果,这被称为基因靶向治疗。此外,一些药物也可以靶向非编码RNA,如微小RNA (miRNA) 或长链非编码RNA (lncRNA),以调节细胞的基因表达和生物过程。
药物可以直接靶向基因,也可以通过与转录因子等蛋白质相互作用间接影响基因表达:
- 直接靶向基因的药物通常是指能够直接与基因序列或其调控元件相互作用,并对基因的表达产生影响的药物。这些药物可能会改变基因的启动子活性、转录因子结合、DNA甲基化状态等,从而直接调节基因的表达水平。例如,一些药物可以作为DNA甲基转移酶抑制剂,影响DNA甲基化修饰,进而调节基因的转录活性。
- 另一方面,药物也可以通过与转录因子等蛋白质相互作用间接影响基因表达。转录因子是调节基因表达的关键蛋白质,它们能够结合到基因的启动子区域,促进或抑制基因的转录。一些药物可以与特定转录因子相互作用,影响其活性或稳定性,从而改变基因的表达。这种间接调控基因表达的方式在药物研发中也是很常见的。
靶点发现的应用
近年来,深度学习方法引起了人们的广泛关注,并在制药领域取得了优异的成果。与传统的机器学习方法相比,最近的基于深度学习的架构,例如生成对抗网络(GAN)、循环神经网络和迁移学习技术,已引起越来越多的关注,并已应用于医疗保健的各个方面,例如新的小分子设计、衰老研究以及基于药物扰动细胞系转录数据的药物药理学预测。
利用公开的多组学数据和文本挖掘方法(图3),深度学习最近已用于研究具有紧急且未满足的临床需求的致命疾病。为了确定肌萎缩侧索硬化症 (ALS) 的可行治疗靶点,Pun 等人结合了各种基于生物信息学和深度学习的模型,这些模型使用特定疾病的多组学和基于文本的数据进行训练,以优先考虑可用药物基因,揭示了 ALS 治疗的18个潜在靶点。此外,Fabris等人建立了一种基于深度学习的方法,具有新颖的模块化架构,通过学习从基因或蛋白质特征(例如基因本体术语Gene Ontology terms、蛋白质-蛋白质相互作用和生物途径)检索的模式来识别与多种年龄相关,疾病相关的人类基因。West 等人开发了一个深度学习集成模型,使用超过12000个胚胎和成体细胞的转录组图谱进行训练,揭示了控制胚胎-胎儿转变的新靶点COX7A1,这可以促进我们对正常发育、外胚组织再生和癌症的理解。

- 图3:AI通过使用利用各种公开可用的组学和文本数据来优先考虑特定适应度的靶点。组学数据包括基因组学、转录组学、蛋白质组学、表观基因组学和代谢组学。这些数据提供了有关变化的信号通路、分子相互作用和蛋白质-蛋白质相互作用的信息,可以作为目标优先级的额外输入。基于文本的数据是从资助报告、专利、出版物和临床试验中检索的。在目标优先排序过程中,可以应用多种目标选择标准,例如蛋白质家族类别、开发状态、成药性、毒性和新颖性,以细化人工智能驱动的靶点列表,以符合特定的研究目标。
此外,大型语言模型还可以通过生物医学文本挖掘来帮助发现治疗靶点。基于从数百万出版物中提取的大量文本数据进行预训练,基于大型语言模型的聊天功能,例如来自Microsoft的BioGPT和来自Insilico Medicine的ChatPandaGPT,可以连接疾病、基因和生物过程,从而快速识别涉及疾病发生和进展的生物学机制,以及潜在药物靶点和生物标志物的识别。
大型语言模型理解自然语言和解释复杂科学概念的能力可以使其成为加速疾病假设生成的宝贵工具。然而,通常根据人类生成的文本进行训练的大型语言模型可能无法确定输入数据的准确性和适当性。可能会无意中延续人类的偏见。此外,鉴于这些模型严重依赖已发表的数据,它们识别真正新颖靶点的潜力有限。 因此,重要的是要承认这些局限性,并将其与其他模型一起使用,以确保发现真正新颖且相关的靶点。
使用AI合成数据进行靶点识别
"合成数据"是指模仿现实世界模式和特征的人工生成的数据。通过利用人工智能算法,可以创建合成数据来模拟各种生物场景,从而使研究人员能够探索和分析更广泛的可能性。这种方法在实验数据稀缺或难以获得的领域特别有价值。例如,在罕见疾病或患者数据有限的情况下,人工智能可以根据现有知识和模式生成合成数据。然后,这些合成数据可用于训练人工智能模型并识别可能被忽视的潜在治疗靶点(The discovery of new drug–target interactions for breast cancer treatment)。 合成数据还可用于验证人工智能算法做出的预测,从而为靶点发现过程提供额外的信心。
此外,人工智能生成的合成数据可以帮助解决数据不平衡或偏差问题。在某些治疗领域,特定患者群体在可用数据集中的代表性可能不足,从而导致靶点识别面临挑战。人工智能可以生成代表这些代表性不足的人群的综合数据,从而实现更全面和更具包容性的分析。
尽管人工智能生成的合成数据可以在探索更广泛的可能性和解决数据稀缺方面提供优势,但必须认识到其局限性。模型无法模拟包含模型不知道的复杂性的数据,应该充分认识到这一限制。模拟代表性不足的人群虽然由于数据稀缺而很诱人,但也会引起道德问题,实际上应尽可能收集相关数据,而不是仅仅依赖合成数据。此外,确保合成数据准确捕获现实世界生物系统复杂而细致的方面也是一项重大挑战。因此,实施稳健的验证和质量控制措施对于建立生成数据的可靠性和相关性至关重要。
为了负责任地验证和控制合成组学数据的质量,可以考虑多种选择。首先,可以进行比较分析来评估合成数据和真实数据之间的相似性。这可能涉及统计测量,例如比较分布特征、相关模式。此外,如果可用的话,针对已知的真实数据进行基准测试可以帮助评估合成数据的准确性和性能。另一种方法涉及功能分析,例如在单细胞数据的情况下关注合成数据集中特定细胞类型的表示,以确定合成数据是否捕获生物知识并表现出连贯的功能关系。最后,让领域专家参与提供有价值的见解,并确保用于靶点识别的合成数据的适当性和相关性。
靶点选择标准
用于选择药物靶点的标准对药物开发的成功有很大影响(图3)。因果关系是选择药物靶点的重要标准。了解疾病背后的因果机制可以帮助研究人员识别最有潜力有效治疗疾病的驱动基因和关键pathway。除了实验方法之外,推断靶点与疾病之间因果关系的常见计算方法是基于网络的分析,其中涉及构建生物网络来捕获不同基因、蛋白质、药物和其他分子实体之间的关系(Network analysis reveals rare disease signatures across multiple levels of biological organization)。这些网络可用于根据网络内的中心性和连接性来识别可能与疾病有因果关系的潜在靶点。
另一个重要的考虑因素是靶点的成药性:靶点被药物分子调节的能力。影响成药性的因素包括治疗方式、蛋白质定位、类别和结构可用性。例如,小分子药物通常用于具有明确结合口袋的靶点,而基于蛋白质的疗法更适合小分子难以处理的靶点。 药物靶点的结构信息有助于基于人工智能的预测(例如 AlphaFold)进行药物设计和优化,从而扩大蛋白质结构覆盖范围。 还必须通过评估所涉及的细胞过程、基因必要性和组织特异性来考虑靶点毒性。
除了因果关系、成药性和毒性之外,新颖性是靶点选择的另一个关键因素。基于文本的证据可用于评估给定靶点的新颖性和可信度。通过仔细研究批准的药物、分子靶点和治疗适应症之间的关系,Santos 等人表明高置信度靶点占批准药物的大多数,而针对新型靶点的药物仅占一小部分,尽管这一比例正在增加,特别是在肿瘤学领域。在新颖性和置信度之间取得平衡对于靶点选择至关重要。基于人工智能的自然语言处理方法可以通过提取支持证据,将潜在靶点与基于涉及科学出版物、资助和临床试验的大量数据的适应症联系起来,来帮助靶点选择过程。此外,AI可以通过将高置信度靶点与已知药物与尚未研究药物的新疾病联系起来,促进药物的重利用,从而为常见疾病和罕见疾病实现经济有效且节省时间的药物发现。









![[FlareOn6]FlareBear](https://img-blog.csdnimg.cn/75e6f7a6644c42eb9ef7fe97617e0d5b.png)