本文介绍在计算机多核上通过parallel包进行并行计算。
并行计算运算步骤:
- 加载并行计算包,如library(parallel)。
- 创建几个“workers”,通常一个workers=一个核(core);
- 这些workers什么都不知道,它们的全局环境没有变量,也没有加载的R包,所以无论你想让这些workers做什么你都需要提供相应的对象、库;
- 使用一些可以并行运行循环的函数,如parApply,parLapply,parSapply。
- 当使用完并行后端,且不需要workers的时候,停止它们,否则,它们将继续挂在内存中。
说明:
1-关于并行版本的循环函数。在BaseR中提到循环,有for、while用于循环的函数,也有apply家族函数。相应的,在并行运算中,也有并行运算的apply家族函数。
library(parallel)
# run this code instead to use all available CPU cores
#variable c1 is workers (clusters)
#启动workers,包括确定使用的workers数量。workers=cores
cl <- makeCluster(detectCores())
#将当前R中的变量(这里命名为object1和object2,是任何R对象)导出到新创建的workers的全局变量中,以便workers使用它们。注意第一个参数是workers。
clusterExport (cl, varlist = c("object1", "object2"))
#对some.vector中的每个元素,分别使用FUN作用,返回结果是向量。
#parSapply函数的第一个参数是workers;
#操作类似sapply函数,可先查阅saplly函数的用法。
#将返回结果存储在result对象中
result <- parSapply (cl, some.vector, FUN = function (i) {some.function1; some.function2})
#关闭workers
stopCluster (cl)
示例:
从标准正态分布中生成1e6个随机数,计算这些随机数的均值,这个过程重复100次。
非并行版本代码:
lapply (1:100, FUN = function (x) mean (rnorm (1000000)))并行版本代码:
library (parallel)
cl <- makeCluster (4)
res <- parLapply (cl, X = 1:100, fun = function (x) mean (rnorm (1000000)))
stopCluster (cl)注意:这里使用的lapply和parLapply,sapply函数是lapply函数的简化版,sapply函数返回的是向量,lapply返回的是列表。lapply(list + apply),sapply (simplify+apply).
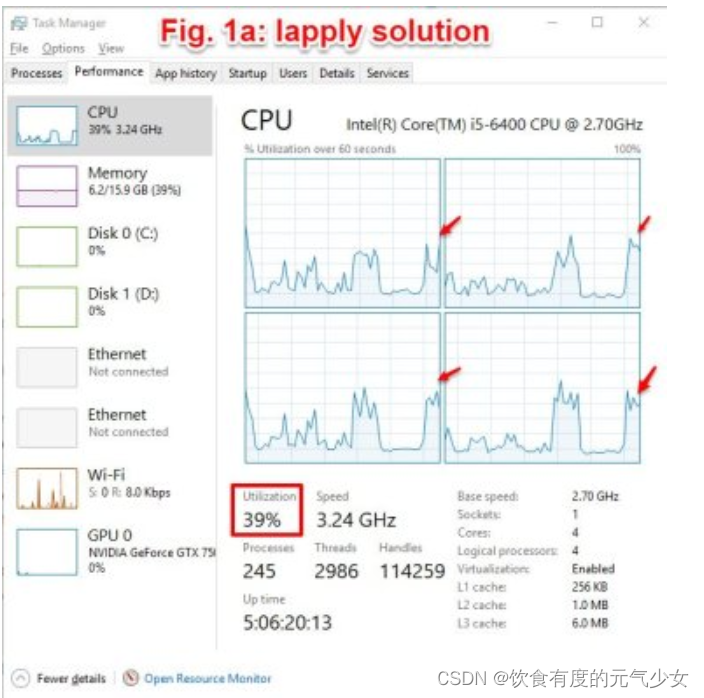
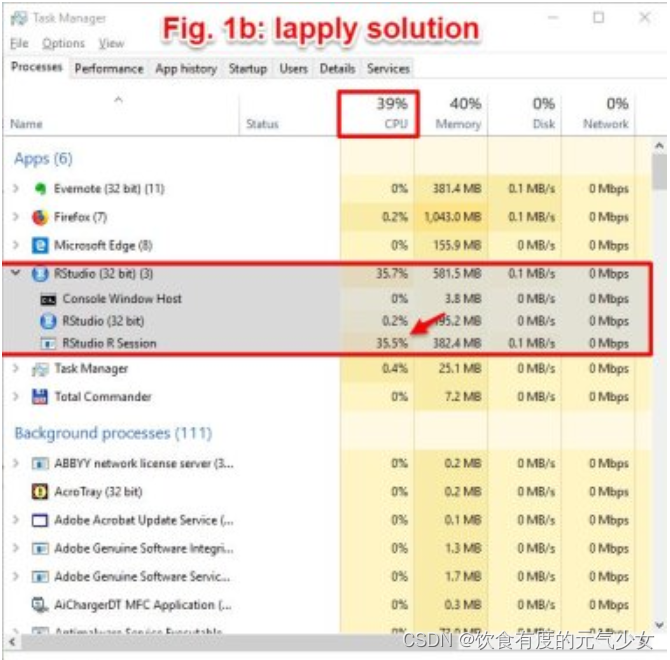
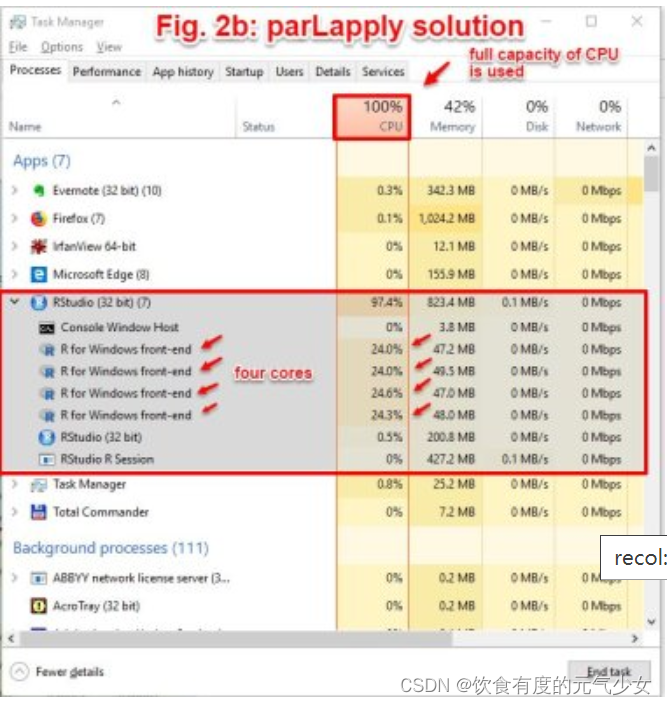
当计算机运行上述两个函数的时候,我们打开任务管理器(快捷键:ctrl+Alt+Del),非并行程序仅使用部分计算机容量,在这个例子中,非并行版本的程序仅使用了39%的CPU,而并行版本的CPU为100%。

R语言的microbenchmark包来进行性能测试,microbenchmark函数是microbenchmark包中的一个函数,用于测量代码块的执行时间。microbenchmark函数的结果将返回一个数据框,其中包含了每次执行的时间结果,以及一些统计信息,如平均时间、最小时间、最大时间等。这段代码的目的是通过microbenchmark函数来测试和比较不同代码块的执行时间,以评估它们的性能。
mb <- microbenchmark::microbenchmark (
{
lapply (1:100, FUN = function (x) mean (rnorm (1e6)))
},
{
library (parallel)
cl <- makeCluster (4L)
res <- parLapply (cl, X = 1:100, fun = function (x) mean (rnorm (1e6)))
stopCluster (cl)
},
times = 10)
mb运行结果:
Unit: seconds
...
min lq mean median uq max neval cld
7.389548 7.522466 7.566548 7.585431 7.605311 7.703006 10 b
2.853429 2.890022 2.954747 2.943975 2.968527 3.114184 10 a
通过两个版本程序运行时间的对比,可以看到,并行版本的程序的计算时间没有比非并行版本的程序快4倍,因为我们使用的是4个核,按照预期应该是并行版本的程序运行速度要快4倍,没有达到这个预期原因是:管理并行也需要花费一些时间:拆分数据、将它们发送给单个workers,收集结果,并将结果合并在一起。
因此,并行计算适应于计算所花费的时间远高于R与单个内核通信所花费的时间。
事实上,如果将计算1e6个随机数的均值,增加到计算1e7个随机数的均值,重复100次,此时,并行版本的速度将增加几乎4倍(非并行83.8 vs 并行21.5).
注意:除非你有一台相当强大的计算机,否则不要尝试运行下面的代码,因为计算机运行下面的代码需要一段时间。注意到,下面的代码中,将重复次数减少到了5,否则需要更长的时间。
mb <- microbenchmark::microbenchmark (
{
lapply (1:100, FUN = function (x) mean (rnorm (1e7)))
},
{
library (parallel)
cl <- makeCluster (4L)
res <- parLapply (cl, X = 1:100, fun = function (x) mean (rnorm (1e7)))
stopCluster (cl)
},
times = 5)
mb
Unit: seconds
...
min lq mean median uq max neval cld
83.08273 83.82933 83.95855 83.97395 84.39401 84.51273 5 b
21.42050 21.43552 21.58001 21.49912 21.58116 21.96373 5 a
参考:
Parallelization in R [David Zelený]