【Matlab】智能优化算法_遗传算法GA
- 1.背景介绍
- 2.数学模型
- 3.文件结构
- 4.详细代码及注释
- 4.1 crossover.m
- 4.2 elitism.m
- 4.3 GeneticAlgorithm.m
- 4.4 initialization.m
- 4.5 Main.m
- 4.6 mutation.m
- 4.7 selection.m
- 4.8 Sphere.m
- 5.运行结果
- 6.参考文献
1.背景介绍
遗传算法(Genetic Algorithm,简称GA)是一种基于生物进化理论的优化算法,由John Holland于20世纪70年代初提出。它通过模拟自然选择和遗传机制,利用群体中个体之间的遗传信息交流和变异来搜索问题的解空间。
遗传算法的设计灵感来源于达尔文的进化论。达尔文提出,自然界中的生物通过遗传信息的传递和变异,逐步适应环境并进化。类似地,遗传算法通过对问题解空间中的个体进行选择、交叉和变异操作,模拟了生物进化的过程,以寻找问题的最优解或次优解。
2.数学模型
遗传算法的核心思想是通过不断迭代的过程,从初始的随机个体群体出发,通过选择、交叉和变异操作产生新一代的个体群体,使得群体中的个体逐渐适应环境并优化问题的目标函数。具体而言,遗传算法的步骤如下:
- 初始化:随机生成初始的个体群体,代表解空间中的潜在解。
- 评估:根据问题的目标函数,对每个个体进行评估,计算其适应度值,表示解的优劣程度。
- 选择:根据适应度值,选择优秀的个体作为父代,用于产生下一代的个体。选择策略可以是轮盘赌选择、竞争选择等。
- 交叉:从父代个体中选取一对个体,通过交叉操作产生新的个体,将两个个体的染色体信息进行混合。
- 变异:对新生成的个体进行变异操作,引入随机性,增加搜索空间的多样性。
- 更新:用新生成的个体替换原有的个体群体,形成下一代个体群体。
- 终止条件:通过设定的终止条件,如达到最大迭代次数、目标函数值达到一定阈值等,判断算法是否停止。
- 返回最优解:遗传算法迭代完成后,返回适应度值最高的个体作为问题的解。
遗传算法具有全局搜索能力、自适应性和鲁棒性,适用于各种优化问题,尤其在复杂、多模态和高维的问题中表现出色。它在工程、运筹学、人工智能等领域都有广泛应用,并衍生出许多变种算法和改进方法,如遗传编程、进化策略等。
3.文件结构

crossover.m % 交叉育种
elitism.m % 精英化
GeneticAlgorithm.m % 遗传算法
initialization.m % 初始化
Main.m % 主函数
mutation.m % 变异
selection.m % 选择
Sphere.m %
4.详细代码及注释
4.1 crossover.m
function [child1 , child2] = crossover(parent1 , parent2, Pc, crossoverName)
switch crossoverName
case 'single'
Gene_no = length(parent1.Gene);
ub = Gene_no - 1;
lb = 1;
Cross_P = round ( (ub - lb) *rand() + lb );
Part1 = parent1.Gene(1:Cross_P);
Part2 = parent2.Gene(Cross_P + 1 : Gene_no);
child1.Gene = [Part1, Part2];
Part1 = parent2.Gene(1:Cross_P);
Part2 = parent1.Gene(Cross_P + 1 : Gene_no);
child2.Gene = [Part1, Part2];
case 'double'
Gene_no = length(parent1);
ub = length(parent1.Gene) - 1;
lb = 1;
Cross_P1 = round ( (ub - lb) *rand() + lb );
Cross_P2 = Cross_P1;
while Cross_P2 == Cross_P1
Cross_P2 = round ( (ub - lb) *rand() + lb );
end
if Cross_P1 > Cross_P2
temp = Cross_P1;
Cross_P1 = Cross_P2;
Cross_P2 = temp;
end
Part1 = parent1.Gene(1:Cross_P1);
Part2 = parent2.Gene(Cross_P1 + 1 :Cross_P2);
Part3 = parent1.Gene(Cross_P2+1:end);
child1.Gene = [Part1 , Part2 , Part3];
Part1 = parent2.Gene(1:Cross_P1);
Part2 = parent1.Gene(Cross_P1 + 1 :Cross_P2);
Part3 = parent2.Gene(Cross_P2+1:end);
child2.Gene = [Part1 , Part2 , Part3];
end
R1 = rand();
if R1 <= Pc
child1 = child1;
else
child1 = parent1;
end
R2 = rand();
if R2 <= Pc
child2 = child2;
else
child2 = parent2;
end
end
4.2 elitism.m
function [ newPopulation2 ] = elitism(population , newPopulation, Er)
M = length(population.Chromosomes); % number of individuals
Elite_no = round(M * Er);
[max_val , indx] = sort([ population.Chromosomes(:).fitness ] , 'descend');
% The elites from the previous population
for k = 1 : Elite_no
newPopulation2.Chromosomes(k).Gene = population.Chromosomes(indx(k)).Gene;
newPopulation2.Chromosomes(k).fitness = population.Chromosomes(indx(k)).fitness;
end
% The rest from the new population
for k = Elite_no + 1 : M
newPopulation2.Chromosomes(k).Gene = newPopulation.Chromosomes(k).Gene;
newPopulation2.Chromosomes(k).fitness = newPopulation.Chromosomes(k).fitness;
end
end
4.3 GeneticAlgorithm.m
function [BestChrom] = GeneticAlgorithm (M , N, MaxGen , Pc, Pm , Er , obj, visuailzation)
cgcurve = zeros(1 , MaxGen);
%% Initialization
[ population ] = initialization(M, N);
for i = 1 : M
population.Chromosomes(i).fitness = obj( population.Chromosomes(i).Gene(:) );
end
g = 1;
disp(['Generation #' , num2str(g)]);
[max_val , indx] = sort([ population.Chromosomes(:).fitness ] , 'descend');
cgcurve(g) = population.Chromosomes(indx(1)).fitness;
%% Main loop
for g = 2 : MaxGen
disp(['Generation #' , num2str(g)]);
% Calcualte the fitness values
for i = 1 : M
population.Chromosomes(i).fitness = Sphere( population.Chromosomes(i).Gene(:) );
end
for k = 1: 2: M
% Selection
[ parent1, parent2] = selection(population);
% Crossover
[child1 , child2] = crossover(parent1 , parent2, Pc, 'single');
% Mutation
[child1] = mutation(child1, Pm);
[child2] = mutation(child2, Pm);
newPopulation.Chromosomes(k).Gene = child1.Gene;
newPopulation.Chromosomes(k+1).Gene = child2.Gene;
end
for i = 1 : M
newPopulation.Chromosomes(i).fitness = obj( newPopulation.Chromosomes(i).Gene(:) );
end
% Elitism
[ newPopulation ] = elitism(population, newPopulation, Er);
cgcurve(g) = newPopulation.Chromosomes(1).fitness;
population = newPopulation; % replace the previous population with the newly made
end
BestChrom.Gene = population.Chromosomes(1).Gene;
BestChrom.Fitness = population.Chromosomes(1).fitness;
if visuailzation == 1
plot( 1 : MaxGen , cgcurve);
xlabel('Generation');
ylabel('Fitness of the best elite')
end
end
4.4 initialization.m
function [ population ] = initialization(M, N)
for i = 1 : M
for j = 1 : N
population.Chromosomes(i).Gene(j) = [ round( rand() ) ];
end
end
4.5 Main.m
clear
clc
%% controling paramters of the GA algortihm
Problem.obj = @Sphere;
Problem.nVar = 20;
M = 20; % number of chromosomes (cadinate solutions)
N = Problem.nVar; % number of genes (variables)
MaxGen = 100;
Pc = 0.85;
Pm = 0.01;
Er = 0.05;
visualization = 1; % set to 0 if you do not want the convergence curve
[BestChrom] = GeneticAlgorithm (M , N, MaxGen , Pc, Pm , Er , Problem.obj , visualization)
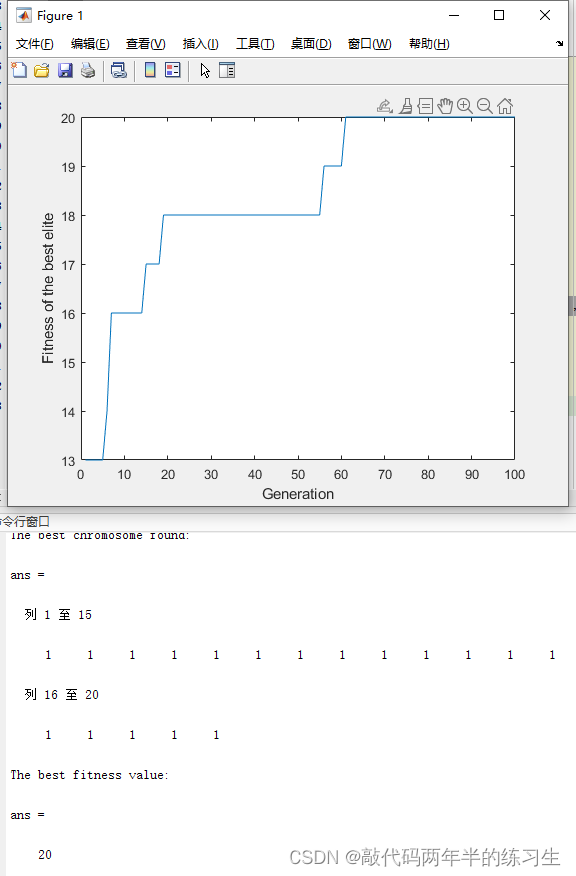
disp('The best chromosome found: ')
BestChrom.Gene
disp('The best fitness value: ')
BestChrom.Fitness
4.6 mutation.m
function [child] = mutation(child, Pm)
Gene_no = length(child.Gene);
for k = 1: Gene_no
R = rand();
if R < Pm
child.Gene(k) = ~ child.Gene(k);
end
end
end
4.7 selection.m
function [parent1, parent2] = selection(population)
M = length(population.Chromosomes(:));
if any([population.Chromosomes(:).fitness] < 0 )
% Fitness scaling in case of negative values scaled(f) = a * f + b
a = 1;
b = abs( min( [population.Chromosomes(:).fitness] ) );
Scaled_fitness = a * [population.Chromosomes(:).fitness] + b;
normalized_fitness = [Scaled_fitness] ./ sum([Scaled_fitness]);
else
normalized_fitness = [population.Chromosomes(:).fitness] ./ sum([population.Chromosomes(:).fitness]);
end
%normalized_fitness = [population.Chromosomes(:).fitness] ./ sum([population.Chromosomes(:).fitness]);
[sorted_fintness_values , sorted_idx] = sort(normalized_fitness , 'descend');
for i = 1 : length(population.Chromosomes)
temp_population.Chromosomes(i).Gene = population.Chromosomes(sorted_idx(i)).Gene;
temp_population.Chromosomes(i).fitness = population.Chromosomes(sorted_idx(i)).fitness;
temp_population.Chromosomes(i).normalized_fitness = normalized_fitness(sorted_idx(i));
end
cumsum = zeros(1 , M);
for i = 1 : M
for j = i : M
cumsum(i) = cumsum(i) + temp_population.Chromosomes(j).normalized_fitness;
end
end
R = rand(); % in [0,1]
parent1_idx = M;
for i = 1: length(cumsum)
if R > cumsum(i)
parent1_idx = i - 1;
break;
end
end
parent2_idx = parent1_idx;
while_loop_stop = 0; % to break the while loop in rare cases where we keep getting the same index
while parent2_idx == parent1_idx
while_loop_stop = while_loop_stop + 1;
R = rand(); % in [0,1]
if while_loop_stop > 20
break;
end
for i = 1: length(cumsum)
if R > cumsum(i)
parent2_idx = i - 1;
break;
end
end
end
parent1 = temp_population.Chromosomes(parent1_idx);
parent2 = temp_population.Chromosomes(parent2_idx);
end
4.8 Sphere.m
function [fitness_value] = Sphere( X )
fitness_value = sum(X.^2);
end
5.运行结果
6.参考文献
[1]Hayes-Roth F. Review of “Adaptation in Natural and Artificial Systems by John H. Holland”, The U. of Michigan Press, 1975[J]. ACM SIGART Bulletin,1975,53(53).