教程和练习文件从软件官网下载
内容:
1、拷贝教程提供的练习文件素材:
在软件的help中选择需要的教程,点击Copy to,可以将教程需要的文件拷贝到指定的文件夹里。点击Browse可以进行预览。
2、建立一个数值型的QSAR模型评估结合亲和力:
这个练习演示了如何用AutoQSAR面板来设置、建立、介绍和应用与FactoXa结合的一系列同源配体的QSAR模型。
FactoXa(也称为Factor Xa)是一种蛋白酶,属于凝血系统中的血浆蛋白。
它是凝血级联反应中的重要组成部分,起着关键的生理功能,
主要功能是将凝血级联反应中的凝血因子X(Factor X)转化为凝血酶(thrombin)。
凝血酶进一步将纤维蛋白原转化为纤维蛋白,使血液凝固。2.1、准备QSAR建模的数据:
在使用数据之前,Maestro Project Table将会被使用来处理以uM为单位结合结合亲和力数据。
- 单位的转换,IC50、EC50这些数据,多是以uM为单位,需要取对数转换为自由能标度。
- 建议将数据集分隔成75%的训练集和25%的验证集。
- 确保数据集大小合理。
- 配体应该提供2D或3D结构,每个配体具有单一电离和互变异构状态。训练集、验证集和预测集中分子的状态应该保持一致。
操作:
点击File>Import Structures

导入Fxa_1z6e+train.maegz文件
点击window>project Table
这里IC50,半数抑制率的单位是浓度uM(1uM\L),教程里将其转化为mol\L并取对数。
点击Execute执行,获得pIC50的新结合能属性,点击close关闭Calculator。
2.2、建立QSAR模型:
点击Tasks>AutoQsar
点击run运行 ,任务开始后,run按钮左侧的图标开始旋转。
在window>job Monitor里可动态监测任务的状态。

状态说明任务已经执行完成了。任务完成后,我们得到一个qzip文件,这个就使建立好的模型。
将其导入到AutoQSAR面板中的View model and make prediction。
2.3、对建立的模型进行检查:

这个模型名字说明,是用KPLS算法(一种偏最小二乘回归的扩展)拟合molprint2D指纹,在第38次对数据集进行分割(分为训练集和学习集)时候获得的。
molprint2D(fingerprint)是一种分子描述符(descriptor)或指纹方法,
用于表示化学分子的结构特征。molprint2D指纹是基于分子的二维结构信息生成的。
不同的软件工具和编程库提供了molprint2D指纹的计算和应用功能,
如RDKit、ChemAxon等。在Report Details中可以对模型进行可视化
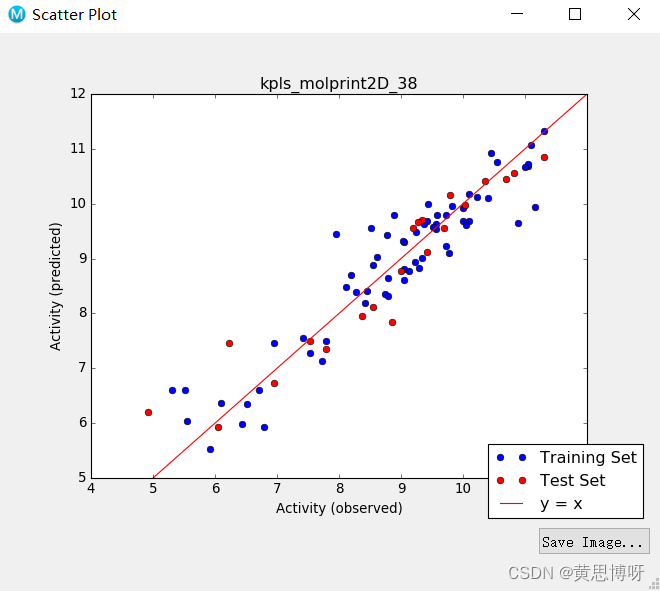
2.4、用建立的模型对未知分子进行预测:
导入预测的分子的操纵
点击File>Import Structures
Files of type选择Maestro
在Options中确保Import all structures and Replace Workspace被选择。
打开教程提供的fxa_predict.maegz选中导入的所有分子
打开Tasks里的AtuoQSAR,操作如下图:

等待任务完成。
2.5、对预测结果进行分析:
导入预测好的fxa_prediction-out.mae.gz文件,选中导入的所有小分子
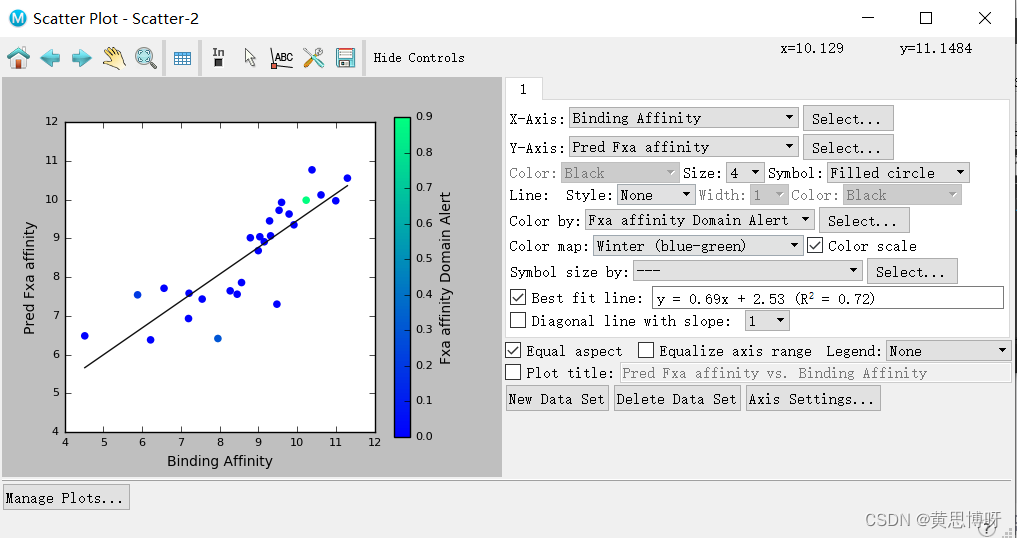
打开Project Table面板,点击Plot>New Scatter Plot
X轴选择Binding Affinity,Y轴选择Pred fxa affinity(由AutoQSAR计算得到)
设置Color by --> fxa affinity Domain Alert,Color map --> Winter(blue-green).
选择Best fit line选项,得到图片:

预警报化合物(Alert compounds)是指具有潜在毒性、危险性或不良效应的化合物
,可以通过特定的结构或功能团来进行识别和警示。
这些结构或功能团通常与已知的毒性或不良反应相关联。
预警报化合物通常基于已知的毒性数据、结构-活性关系和毒理学知识进行开发和验证。
一些常见的预警报结构或功能团包括:
烷基化合物:具有高脂溶性、高毒性或致癌性的长碳链烷基化合物。
酚类化合物:具有毒性、过敏性或肝毒性的酚类结构。
烯醇类化合物:具有不稳定性、反应性或肝毒性的烯醇结构。
亲电芳香族化合物:具有致突变性、致畸形性或致癌性的芳香族化合物。
羧酸类化合物:具有肝毒性、肾毒性或免疫毒性的羧酸结构。
需要注意的是,预警报化合物并不代表一定具有毒性或不良效应,而只是作为可能的指示物,
需要进一步评估和确认。对于具体的化学品评估和风险评估,综合考虑多种因素,
包括毒性数据、剂量效应关系、曝露情况和用途等,才能做出准确的评估和决策。去掉错误的小分子(绿色点,表示Alert compounds),再重新作图:




![[MMDetection]测试模型](https://img-blog.csdnimg.cn/4572b274a4b748e38ac3ab1f2ecf7732.png)