Rdkit|分子可视化
Github: 地址
单个展示
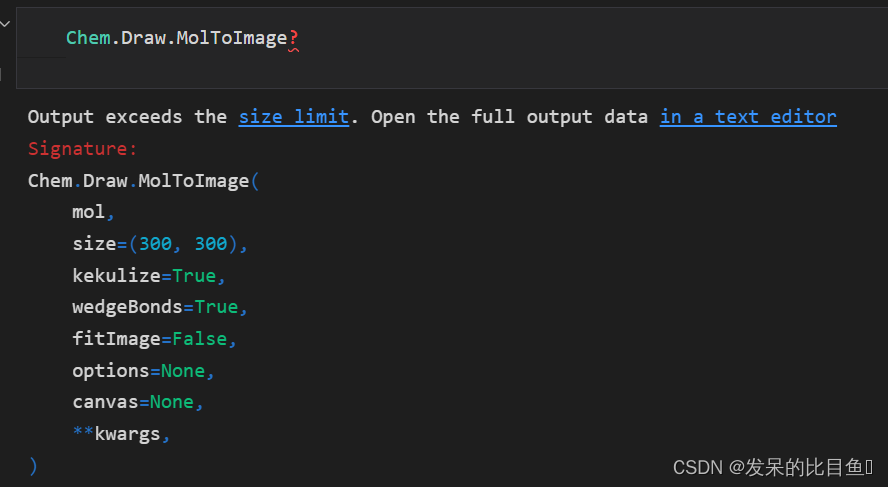
从mol对象到图片:MolToImage(mol, size, kekulize, wedgeBonds, fitImage, …)
mol:mol对象
size:图片尺寸,默认(300, 300)
kekulize:是否展示kekule形式,默认True(True:芳香系统用实线表示,False:虚线表示)
wedgeBonds:是否展示楔形键,即立体构型,默认True
import pandas as pd
from rdkit.Chem import PandasTools
from rdkit import Chem
from rdkit.Chem import Draw
from rdkit.Chem import AllChem
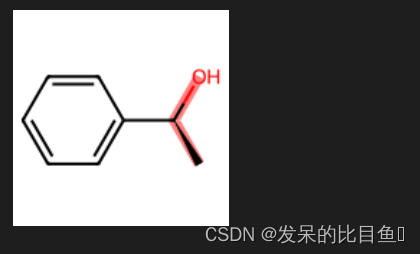
mol = Chem.MolFromSmiles("C[C@H](O)c1ccccc1")
image = Chem.Draw.MolToImage(mol, size=(150,150), kekulize=True, highlightBonds=[0,1])
## 保存
image.save("./data/im.png")
新窗口中展示图片
在新窗口中展示图片:ShowMol()
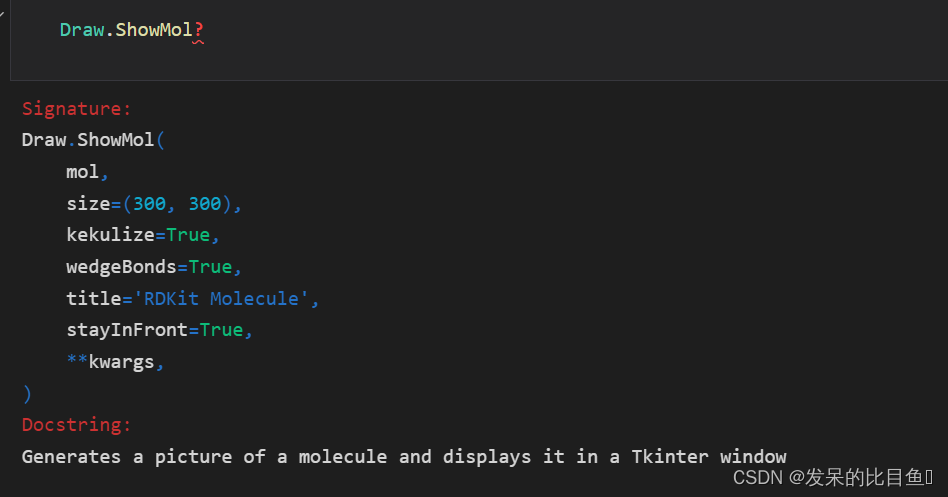
Draw.ShowMol(mol, size=(150,150), kekulize=False)
保存图片
保存图片MolToFile(mol, filename, size, kekulize, wedgeBonds, …)
参数基本同上
Draw.MolToFile(mol, 'data/output.png', size=(150, 150))
批量展示
从DataFrame中展示
从df中展示:FrameToGridImage(frame, column, molsPerRow, subImgSize, legendsCol, …)
frame:DataFrame对象
column:rdkit mol对象所在列
molsPerRow,:每行显示的分子数
subImgSize:图片大小
legendsCol:标题所在列
df = pd.read_csv('./data/batch.csv')
PandasTools.AddMoleculeColumnToFrame(df,'SMILES','mol',includeFingerprints=True)
PandasTools.FrameToGridImage(df, column='mol', molsPerRow=5, subImgSize=(200,200), legendsCol='Name')
从mol列表中展示
从列表生成分子结构:MolsToGridImage(mols, maxMols, molsPerRow, subImgSize, legends, …)
部分参数和上面的一致
mols:mol对象列表
maxMols:最多显示的分子数
molsPerRow,:每行显示的分子数
subImgSize:图片大小
legends:图题
mols = df.mol.tolist()
legends = df.Name.tolist()
legends = [str(l) for l in legends]
Draw.MolsToGridImage(mols, molsPerRow=2, subImgSize=(300,300), legends=legends)
3D展示
转换3D时,为了得到靠谱的三维构象,一般先加氢:AddHs(mol)
通过距离几何算法计算3D坐标:EmbedMolecule(mol, randomSeed, …)
mol:mol对象
randomSeed:随机种子
转换完后再进行一步力场优化,比如MMFF94:MMFFOptimizeMolecule(mol)