A Review of the State-of-the-Art on Tours for Dynamic Visualization of High-dimensional Data
Visualization of High-dimensional Data)
Lee, Stuart, et al. “A Review of the State-of-the-Art on Tours for Dynamic Visualization of High-dimensional Data.” arXiv preprint arXiv:2104.08016 (2021).
本文讨论了一种称为tour的高维可视化技术,它可以用于查看三维以上的数据。我们回顾了该技术背后的理论和历史,以及在科学和机器学习中发现的现代软件开发和应用。
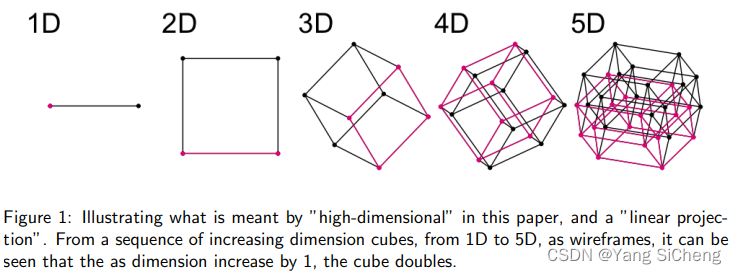
图1:说明了本文中“高维”和“线性投影”的含义。以线框图的形式,由一维到5D的维度递增的立方体序列可以看出,当维度增加1时,立方体会翻倍。
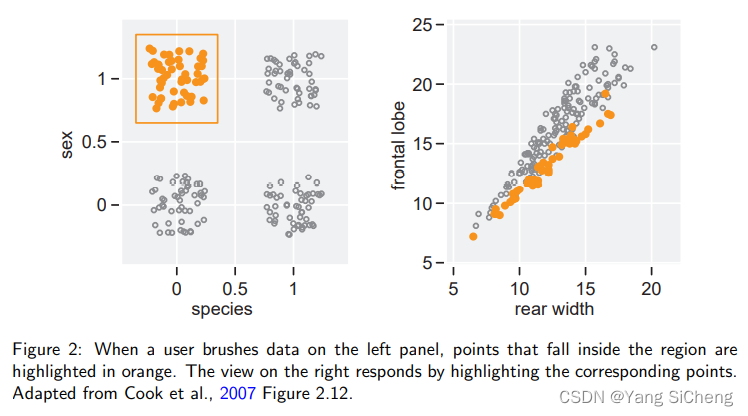
图2:当用户在左侧面板上刷数据时,该区域内的点以橙色突出显示。右边的视图通过强调相应的点来回应。摘自Cook等人,2007年图2.12。
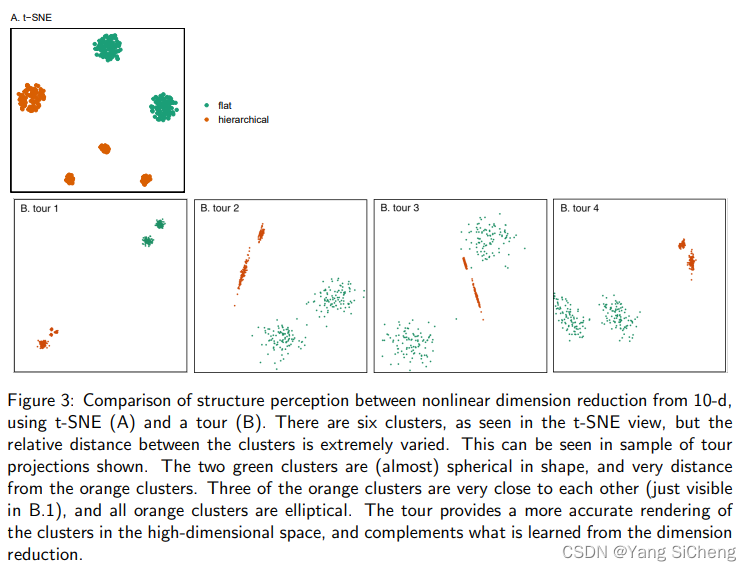
图3:用t-SNE (A)和tour (B)从10 d非线性降维的结构感知比较。在t-SNE视图中,有6个簇,但簇之间的相对距离变化很大。这可以从所示的旅游预测样本中看出。这两个绿色的星团(几乎)是球形的,与橙色星团的距离非常远。其中三个橙色星团彼此非常接近(仅在B.1中可见),所有橙色星团都是椭圆形的。该指南提供了高维空间中集群的更准确的呈现,并补充了从降维中学到的内容。
Dynamic visualization of high-dimensional data
降维(DR)是高维数据分析中的一种标准方法。除了降低噪声和促进下游计算分析,DR方法被广泛用于在二维或三维可视化数据。事实上,已经开发了许多DR方法来生成高维数据的可视化信息。目前比较流行的数据可视化DR方法包括线性主成分分析(PCA)、非线性t分布随机邻居嵌入(t-SNE)[1]和统一流形逼近与投影(UMAP)[2]。已经开发了许多其他DR方法来解决常用的t-SNE和UMAP方法的缺点[3-5]。可视化的DR方法已经在广泛的不同学科中找到了特定的用途。一些例子包括在单细胞生物学中验证细胞类型身份[6,7],从深度学习模型探索输入嵌入,探索人类基因组[8]的地理模式,以及解剖恒星物体[9]的化学丰度。尽管DR方法在高维数据可视化方面很受欢迎,但这些方法在低维可视化质量上存在失真和异质性[6,10-14]。因此,天真地使用DR方法来验证、确认或告知研究结果和方向很容易由于这些歪曲而被误解。例如,在单细胞生物学领域,通常使用t-SNE或UMAP可视化来确认[6]簇的细胞类型身份,整合不同的单细胞数据集[15 - 17],并使用RNA速度测量来计算细胞轨迹[18,19]。对于前面提到的每一个用例,观测者变化之间的距离和DR可视化质量的异质性都存在扭曲,并可能影响最终的解释[10,13,20 - 22]。一般来说,通过这些扭曲,DR可视化可能会导致簇的验证不正确(即低簇或过簇),人工检测或缺乏对簇之间桥接连接的检测,以及人工存在沿元数据轴的观测顺序或丢失。
降维法(DR)通常用于将高维数据投影到低维数据中进行可视化,从而产生新的见解和假设。然而,DR算法必然会在可视化中引入失真,并且不能完全代表数据中的所有关系。因此,需要有评估DR可视化可靠性的方法。在这里,我们提出了DynamicViz,这是一个生成动态可视化的框架,它捕获了DR可视化对数据扰动的敏感性。DynamicVic可以应用于所有常用的DR方法。我们展示了动态可视化在诊断静态可视化的常见解释缺陷和扩展现有单细胞分析方面的效用。我们引入方差分数来量化这些可视化中观测结果的动态可变性。方差分数表征了数据的自然可变性,可以用来优化DR算法的实现。我们已经免费提供DynamicViz来帮助评估DR可视化。