需要本项目的全套环境、代码、文档、资源、数据和部署调试的私信博主!!!
本研究基于中国新冠疫情2020-01-11至2022-12-20的全国整体数据进行疫情大数据分析,通过对历史的数据进行大数据分析,可以有效的掌握过去疫情数据的特点,便于对疫情之后的防控提供更好的决策。
本次研究的数据集,来自于国内卫健委疫情官方数据集,通过对近三年的疫情数据进行大数据分析,可以有效的了解到历史疫情的一个整体的规律和特点,同时在疫情大数据可视化上有很重要的参考意义。首先,将开源疫情数据,进行预处理,清洗其中包含的空值以及对时间字段进行扩充为年、月、日、周等字段,方便后续的时间维度分析。其次将清洗好的数据集上传到Hadoop平台中,通过配置好flume和hive环境然后编写shell脚本将数据集利用Flume组件进行自动加载到hive数据库中,达到一键化的导入数据集。其次在hive中进行大数据分析,结合多维度的字段对疫情数据从时间上、空间上、横向与纵向的分析,进一步的挖掘疫情数据的特点和发生的时间上的分布情况。
最后将hive中分析出来的结果,进行可视化展示,这里结合Python的pyecharts可视化库,编写可视化代码进一步的将分析结果进行前端可视化展示,便于查看,最后利用pyecharts中的page组件将图表进行组合,搭建疫情可视化大屏,构建交互式的动态可视化展示,本研究基于Hadoop和Python的预处理、自动加载数据、大数据分析、可视化展示与构建可视化大屏,具有一体化的效果。
1.1 研究背景
随着互联网和移动通信技术的快速发展,数据在各个领域中得到了广泛应用,如金融、医疗、教育等。而在全球范围内爆发的新冠肺炎疫情更是展现了数据处理和分析的重要性。在这场疫情中,许多国家和地区都采取了不同的防控措施,包括医疗资源的分配、公共卫生政策的制定和宣传等,这些都需要大量的数据支持和分析。因此,对疫情数据进行深入研究和分析对于更好地应对疫情具有重要意义。
然而,由于疫情数据规模庞大、复杂多变,传统的数据处理和分析方法已经无法满足需求。而Hadoop作为一种新兴的大数据处理技术,具有分布式存储、并行处理、容错性高等优势,能够很好地解决传统方法所遇到的问题。因此,基于Hadoop的疫情信息分析与可视化研究成为了当今研究的热点。
Hadoop是由Apache基金会开发的一种开源的大数据处理框架,能够对大规模数据进行存储和分析。它的核心是Hadoop分布式文件系统(HDFS)和MapReduce计算模型。HDFS将数据分散存储在不同的节点上,并保证数据的冗余备份,从而保证数据的可靠性。而MapReduce则是一种基于分布式计算的编程模型,能够对分散在不同节点上的数据进行并行处理,加快数据处理的速度。
基于Hadoop的疫情信息分析与可视化研究可以从多个角度入手。首先,可以从病例数据入手,通过对各地疫情数据的分析,了解病毒的传播规律和趋势,为政策制定和疫情防控提供科学依据。其次,可以从医疗资源入手,对各地区的医疗资源进行分析,了解医疗资源的分配情况和不足之处,为优化医疗资源配置提供数据支持。再次,可以从公共卫生政策入手,对各地区的宣传政策进行分析,了解公众对政策的反应和反馈,为政策制定和宣传提供数据支持。
总之,基于Hadoop的疫情信息分析与可视化研究具有很大的研究价值和应用前景。通过对疫情数据的深入分析,可以为疫情防控提供科学依据和精准决策,从而更好地保障人民健康和社会稳定。此外,Hadoop作为一种新兴的大数据处理技术,将在更多领域中得到应用和发展,因此,基于Hadoop的疫情信息分析与可视化研究也具有推动大数据技术发展的作用。
在研究中,需要注意数据的来源和准确性,避免因数据质量问题造成研究结果的误导和偏差。同时,需要充分利用Hadoop的分布式存储和并行处理优势,提高数据处理效率和精度,以便更好地服务于疫情防控和人民生活。此外,研究还需要结合可视化技术,将研究结果以图表、地图等形式进行展示和呈现,让数据更具可读性和直观性,帮助决策者更好地理解数据和研究结果。
综上所述,基于Hadoop的疫情信息分析与可视化研究是一个具有重要意义和广泛应用前景的研究领域,其研究成果将为疫情防控和大数据技术发展做出重要贡献。
1.2 国内外研究现状分析
随着全球新冠肺炎疫情的爆发,疫情信息分析与可视化成为热门研究领域。Hadoop作为一个开源的分布式计算框架,已经在疫情信息分析与可视化的研究中发挥了重要作用。本文将对基于Hadoop的疫情信息分析与可视化研究的国内外研究现状进行分析。
国外研究现状:
在国外,基于Hadoop的疫情信息分析与可视化研究主要集中在疫情数据的采集、处理和分析等方面。例如,一些研究使用Hadoop技术处理大规模的疫情数据,将数据存储在Hadoop分布式文件系统中,并使用MapReduce进行数据处理和分析。通过这些研究,可以快速准确地获取疫情数据的情况,并生成相应的可视化图表。
另外,一些研究利用Hadoop技术构建了疫情预测模型。这些模型可以根据历史疫情数据,预测未来的疫情走势,并提供相应的预警。这对于政府部门和医疗机构制定相应的应对措施非常重要。
国内研究现状:
在国内,基于Hadoop的疫情信息分析与可视化研究也开始得到关注。一些研究利用Hadoop技术处理和分析新冠肺炎疫情数据,生成相应的可视化图表。例如,一些研究使用Hadoop技术对新冠肺炎疫情数据进行实时处理,并生成相应的地图展示疫情的分布情况。
此外,一些研究利用Hadoop技术构建了疫情知识图谱。这些图谱可以帮助用户快速准确地获取疫情相关的知识,并为政府部门和医疗机构提供相应的参考。
总结:
基于Hadoop的疫情信息分析与可视化研究在国内外都有较为广泛的应用。国外研究主要集中在疫情数据的处理和分析,以及疫情预测模型的构建。而国内研究则主要集中在疫情数据的可视化和知识图谱的构建。未来,基于Hadoop的疫情信息分析与可视化研究还有很大的发展空间,可以进一步提高对疫情的监测和预警能力,为政府部门和医疗机构提供更加准确的数据支持和决策依据。另外,未来可以将Hadoop技术与人工智能、机器学习等技术相结合,开发更加智能化、自适应的疫情信息分析和预测模型。
总之,基于Hadoop的疫情信息分析与可视化研究在全球范围内都具有重要的应用价值。未来,随着技术的不断发展和完善,相信这一领域的研究将会取得更加深入和广泛的成果。
1.3 研究目的
随着全球新冠疫情的爆发,疫情信息的收集、整理、分析和可视化变得越来越重要。基于Hadoop的疫情信息分析与可视化研究的研究目的在于利用大数据技术分析全球疫情数据,探究疫情传播规律,为疫情防控提供科学依据。
本研究的具体目的如下:
(1)收集和整理疫情数据
本研究将从全球各个国家和地区收集新冠疫情相关数据,包括确诊病例数、死亡病例数、治愈病例数、新增病例数、疫苗接种情况等,利用Hadoop集群进行数据处理和存储。
(2)分析疫情传播规律
本研究将利用Hadoop平台上的分布式计算和数据挖掘技术,对收集的疫情数据进行分析,探究疫情的传播规律。具体来说,可以通过疫情时间序列分析、疫情空间分析等手段,研究不同地区、不同时间段的疫情传播情况,并探究疫情传播的影响因素,如人口密度、气候、经济发展水平等。
(3)实现疫情可视化
本研究将利用Hadoop集群上的可视化工具,将研究结果呈现出来,以便疫情信息的传播和理解。研究人员可以通过地图、折线图、柱状图等多种可视化形式展示疫情数据,从而更加直观地了解疫情传播情况。
总之,基于Hadoop的疫情信息分析与可视化研究的研究目的在于利用大数据技术分析全球疫情数据,探究疫情传播规律,为疫情防控提供科学依据。通过本研究,我们希望能够提高人们对疫情的认识和了解,为全球疫情防控做出贡献。
为了实现上述研究目的,本研究将采用以下方法:
(1)数据采集和处理
本研究将使用Python等程序语言编写网络爬虫,从各种公开数据源收集新冠疫情数据。采集到的数据将进行清洗和整理,并使用Hadoop平台上的HDFS和HBase进行存储和管理。
(2)疫情传播规律分析
本研究将使用Hadoop上的MapReduce、Spark等分布式计算和数据挖掘技术,对疫情数据进行分析。具体来说,可以使用时间序列分析、空间统计分析、聚类分析等方法,探究不同地区、不同时间段的疫情传播规律。通过分析疫情传播规律,可以为疫情防控提供科学依据和指导。
(3)疫情可视化展示
本研究将使用Hadoop平台上的可视化工具,将疫情数据呈现为地图、折线图、柱状图等多种可视化形式,帮助人们更加直观地了解疫情传播情况。同时,疫情可视化还可以帮助疫情信息的传播和理解,提高公众的疫情意识和防控意识。
综上所述,基于Hadoop的疫情信息分析与可视化研究具有重要的意义和价值。本研究将采用先进的大数据技术和方法,对全球疫情数据进行分析和可视化展示,探究疫情传播规律,为疫情防控提供科学依据。本研究的研究成果将为公众、政府、医疗机构等各方提供重要的参考和指导,帮助全球共同应对疫情挑战。
1.4 研究意义
随着科技的不断发展,大数据技术在各个领域得到了广泛的应用。在当前全球范围内爆发的新冠肺炎疫情中,利用大数据技术进行疫情信息的分析与可视化已成为了研究的热点。Hadoop作为目前应用最广泛的大数据处理平台之一,在这个领域中也扮演着重要的角色。
研究利用Hadoop进行疫情信息分析与可视化,可以帮助我们更好地了解疫情发展趋势、疫情防控情况,为决策提供科学依据。具体来说,这个研究可以带来以下几个方面的研究意义:
首先,基于Hadoop的疫情信息分析与可视化研究可以帮助我们更好地了解疫情的传播趋势和空间分布情况。疫情的传播过程非常复杂,受到多种因素的影响,如人口密度、交通流动等。通过收集、整合和分析疫情数据,可以利用Hadoop的分布式计算能力,对疫情数据进行处理和挖掘,找出其中的规律性和相关性,从而得出疫情传播的趋势和空间分布情况,为相关部门提供决策参考。
其次,基于Hadoop的疫情信息分析与可视化研究可以帮助我们更好地评估疫情的风险程度和防控效果。在疫情防控过程中,需要对疫情的风险程度进行准确评估,以及对疫情防控措施的效果进行实时监测和评估。利用Hadoop的大数据处理和分析能力,可以对疫情数据进行深入挖掘和分析,从而评估疫情风险程度和防控效果。同时,通过疫情数据可视化的方式,可以将疫情数据以直观的形式展现出来,使得人们可以更加直观地了解疫情的发展和防控情况。
最后,基于Hadoop的疫情信息分析与可视化研究还可以为未来疫情防控提供重要的参考依据。当前的新冠肺炎疫情已经成为了全球关注的焦点,但未来还可能出现各种新的疫情。通过对当前疫情数据的深入分析和可视化研究,可以发现其中的规律性和变化趋势,为未来疫情防控提供重要的参考依据。例如,在新的疫情爆发时,可以借鉴前期疫情数据的分析结果,对疫情的传播规律和风险评估进行快速准确的判断,从而及时采取相应的防控措施,避免疫情扩散。
综上所述,基于Hadoop的疫情信息分析与可视化研究具有重要的研究意义,不仅可以帮助我们更好地了解当前疫情的发展情况,评估疫情的风险程度和防控效果,还可以为未来疫情防控提供参考依据。随着大数据技术的不断发展和完善,相信基于Hadoop的疫情信息分析与可视化研究会发挥越来越重要的作用,为全球疫情防控事业做出更加积极的贡献。
2 研究总体设计
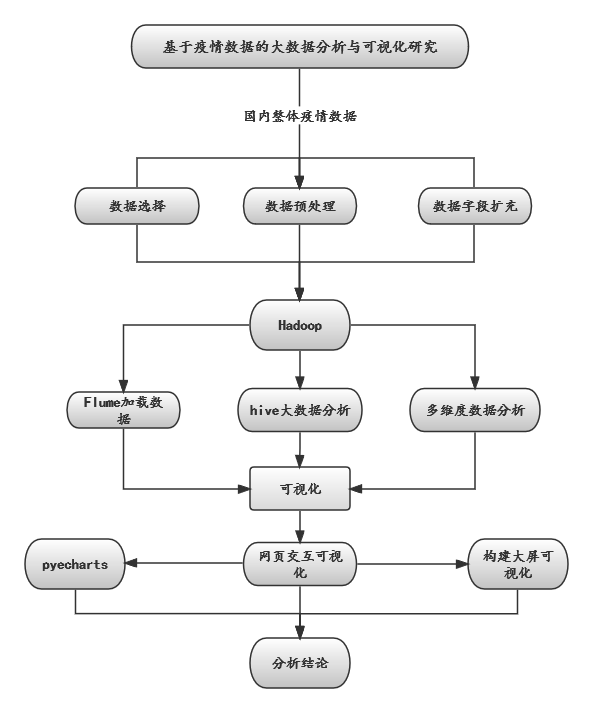
2.1 总体研究路线
本研究旨在通过基于Hadoop的疫情信息分析与可视化研究,探讨如何利用大数据技术对新冠疫情进行分析与可视化展示,为后续疫情防控提供更好的决策依据。本文使用了国内卫健委疫情官方数据集,对中国自2020年1月11日至2022年12月20日的疫情数据进行了分析。
本研究的总体设计路线包括数据预处理、数据自动加载、大数据分析和可视化展示与构建可视化大屏四个部分。首先,针对原始数据中存在的空值和时间格式不一致等问题,进行数据预处理和清洗,将时间字段扩充为年、月、日、周等字段,以方便后续的时间维度分析。其次,利用Flume组件将清洗好的数据集自动加载到Hive数据库中,实现了一键化的导入数据集,为后续的大数据分析打下了基础。
接下来,本研究利用Hive进行大数据分析,结合多维度的字段对疫情数据从时间上、空间上、横向与纵向的分析,进一步挖掘疫情数据的特点和发生时间的分布情况。在分析结果中,本研究注重探究疫情数据的规律和特点,如疫情的高峰期、疫情的传播途径、疫情的分布区域和不同地区疫情数据的对比分析等,为后续疫情防控提供参考依据。
最后,本研究通过Python的可视化库pyecharts,将Hive中分析出来的结果进行可视化展示,并使用page组件将图表进行组合,构建交互式的动态可视化展示,搭建疫情可视化大屏。通过可视化展示,本研究使得疫情数据更加直观、易于理解,并且方便各级部门和公众对疫情数据进行监测和分析,为后续疫情防控提供更好的支持。
总之,本研究基于Hadoop和Python的预处理、自动加载数据、大数据分析、可视化展示与构建可视化大屏,具有一体化的效果。通过此研究,可以更好地利用大数据技术对疫情数据进行分析与可视化展示,为后续疫情防控提供更好的支持和决策依据。
|
图 1 总体设计路线图 |
2.2 Hadoop环境部署
Hadoop是一个开源的分布式计算平台,可以用于处理海量数据。Hadoop环境主要包括HDFS、MapReduce、Hive等组件。下面是一个基本的Hadoop环境部署步骤:
(1)安装Java环境:Hadoop需要Java环境才能运行。可以从Oracle官网下载Java安装包,安装Java环境。
(2)安装Hadoop:从Apache官网下载Hadoop的二进制包,解压到指定目录。在hadoop-env.sh文件中配置JAVA_HOME环境变量。
(3)配置Hadoop集群:修改Hadoop的配置文件,包括core-site.xml、hdfs-site.xml、mapred-site.xml等文件,指定Hadoop集群的各项配置,如文件系统地址、副本数量、数据块大小等。
(4)启动Hadoop集群:在Hadoop安装目录下运行命令行工具,使用start-all.sh脚本启动Hadoop集群。
(5)安装Hive:从Apache官网下载Hive的二进制包,解压到指定目录。在hive-env.sh文件中配置Hadoop的地址和相关环境变量。
(6)配置Hive:修改Hive的配置文件,包括hive-site.xml等文件,指定Hive的相关配置,如数据存储路径、Hadoop的地址等。
(7)启动Hive:在Hive安装目录下运行命令行工具,使用hive命令行工具连接到Hive服务器,进行SQL查询和数据操作。
(8)安装Flume:从Apache官网下载Flume的二进制包,解压到指定目录。在flume-env.sh文件中配置相关环境变量。
(9)配置Flume:修改Flume的配置文件,包括flume.conf等文件,指定Flume的数据源、目的地和数据处理方式等。
(10)启动Flume:在Flume安装目录下运行命令行工具,使用flume-ng命令行工具启动Flume,开始数据流处理。
(11)安装MySQL:从MySQL官网下载MySQL安装包,安装MySQL数据库服务。
(12)创建Hive表:在Hive中创建需要使用的表,指定相关字段和数据类型。可以使用HiveQL语言进行表的创建和操作。
本研究通过搭建Hadoop伪分布式系统,对其进行大数据分析。可以学习Hadoop的基本原理和架构,对Hadoop的运行机制有更深入的了解。能够在单机上模拟多节点的分布式环境,可以更好地测试和开发分布式应用程序。可以充分利用自己的计算资源,提高数据处理效率。
由于Hadoop的部署和安装各类组件比较繁琐,这里就不作具体的详细的安装部署描述了。本研究通过前期花费时间对其进行各类组件安装,并将其结果图展示如下所示:
|
图 2 安装界面部分展示 |
2.3 前期知识准备
2.3.1 Hadoop相关知识介绍
Hadoop是一个基于Java语言开发的分布式计算框架,由Apache开发和维护。它主要用于处理大规模数据集的分布式计算,具有可靠、高效、高扩展性等特点,可以在集群中的数千个计算机节点上并行处理数据。Hadoop分为两个主要模块:Hadoop Distributed File System(HDFS)和MapReduce。其中,HDFS用于存储和管理大规模数据集,MapReduce则用于并行计算数据集。
HDFS是Hadoop的分布式文件系统,它的主要特点是高可靠性、高可用性、高扩展性和高吞吐量。HDFS将大文件分割成多个数据块,存储在不同的节点上,这些数据块可以进行备份以提高可靠性和可用性。HDFS采用主从架构,由一个NameNode和多个DataNode组成,NameNode负责管理和控制文件系统元数据信息,DataNode负责存储和处理数据块。HDFS提供了一套丰富的API和命令行工具,方便用户进行文件的读写、复制、删除等操作。
MapReduce是Hadoop的计算模型,它的核心思想是将数据划分为多个小块,然后并行计算这些小块,最后将结果汇总。MapReduce任务包含两个阶段:Map和Reduce。Map阶段用于将输入数据划分为多个小块,然后在不同的节点上并行计算这些小块,生成中间结果。Reduce阶段用于将Map阶段产生的中间结果进行合并和汇总,得到最终结果。MapReduce的优点在于它可以有效地处理大规模数据集,实现了任务的并行处理和分布式计算。
Hadoop生态系统还包括一系列相关的工具和组件,如Hive、Pig、HBase、ZooKeeper、Sqoop等。这些工具和组件可以与Hadoop集成,扩展其功能和应用场景。例如,Hive和Pig可以使用类似于SQL的语法来查询和处理数据,HBase提供了一个分布式的NoSQL数据库系统,ZooKeeper用于协调分布式应用程序,Sqoop用于在Hadoop和关系型数据库之间进行数据传输。这些工具和组件的加入使得Hadoop生态系统更加完整和强大,为用户提供了更多的选择和灵活性。
2.3.2 Hive相关知识介绍
Hive是一个基于Hadoop生态系统的数据仓库工具,它提供了类SQL的查询和数据管理功能。它允许用户将结构化数据存储在Hadoop分布式文件系统(HDFS)中,并使用SQL语言进行查询和分析。以下是关于Hive的一些重要知识点:
(1)数据仓库:Hive是一个基于数据仓库的工具。数据仓库是一种用于存储和管理大型数据集的系统,其中的数据可以被用于业务智能和数据分析。Hive支持将数据存储在HDFS中,并使用HiveQL进行数据查询和分析。
(2)HiveQL:HiveQL是Hive的查询语言,它类似于SQL语言。HiveQL允许用户使用SQL语法进行数据查询、过滤、聚合和分析操作。HiveQL还支持用户自定义函数和查询语句。
(3)数据类型:Hive支持大部分SQL数据类型,包括整数、浮点数、字符串、日期和时间等。此外,Hive还支持复杂数据类型,例如结构体、数组和映射。
(4)数据存储格式:Hive支持多种数据存储格式,包括文本、序列化、Avro、ORC和Parquet。每种格式都有其自身的优缺点,用户可以根据自己的需求选择适合的格式。
(5)分区和桶:Hive允许用户将数据按照特定的列进行分区。分区可以提高查询性能,减少数据扫描的数量。此外,Hive还支持数据桶,可以进一步提高查询性能。
(6)Hive扩展:Hive提供了许多扩展和插件,包括用户自定义函数、存储处理器和输入/输出格式。用户可以根据自己的需求编写自定义代码,将其添加到Hive中,以扩展其功能。
在疫情信息分析和可视化研究中,Hive可以用于处理和分析大量的疫情数据。通过使用HiveQL进行数据查询和分析,用户可以获得有关疫情的有用信息,并将其可视化以便更好地理解。由于Hive是基于Hadoop的,因此它可以处理大量的数据,并具有可扩展性和高可用性,这使得它成为处理疫情数据的理想工具之一。
2.3.3 Flume相关知识介绍
Flume是一个分布式的、可靠的、高可用的服务,用于收集、聚合、移动和传输大量的日志和事件数据。它是Hadoop生态系统中的一个组件,主要用于将数据从各种不同来源移动到Hadoop集群中进行分析和处理。
Flume的核心理念是将数据从源头推送到目标地,同时允许对数据进行简单的转换和处理。这种推送的方式使得Flume非常适合大规模数据采集和处理的场景。
Flume支持多种数据源,包括本地文件系统、网络协议、社交媒体等。它还提供了多种数据输出方式,包括HDFS、HBase、Kafka等。Flume还提供了丰富的数据过滤和处理功能,如正则表达式匹配、数据格式转换、数据采样等。
Flume的架构分为三个主要组件:Source、Channel和Sink。Source负责从数据源收集数据,Channel负责缓存和传输数据,Sink负责将数据输出到目标地。这种架构使得Flume可以轻松地适应不同的数据源和目标地,并能够进行高效的数据处理和传输。
Flume还支持多种模式,包括单机模式、多代理模式、层次结构模式等。这些模式可以根据具体需求进行选择,以实现最佳的性能和可靠性。
总的来说,Flume是Hadoop生态系统中非常重要的一个组件,它为大规模数据采集和处理提供了可靠、高效的解决方案。同时,Flume的简单易用性和灵活性也使得它成为了数据采集和传输领域的主流工具之一。
2.3.4 Python相关知识介绍
Python是一种高级编程语言,由Guido van Rossum于1991年创建。它是一种通用语言,非常适合用于数据处理、科学计算和Web开发等领域。Python具有简单易学、开发效率高、代码可读性强等特点,因此成为了数据科学和机器学习领域的热门语言之一。
在本文的研究中,Python被用于预处理数据和可视化。以下是Python中一些常见的数据预处理和可视化的库:
(1)NumPy: NumPy是Python中的数值计算库,支持矩阵运算和数值计算。它提供了高性能的数据结构,如ndarray、ufunc等,以及数值计算的函数,如矩阵乘法、线性代数运算等。
(2)Pandas: Pandas是Python中的数据处理库,用于处理和分析数据。它提供了高性能的数据结构,如DataFrame和Series,以及数据处理和分析的函数,如数据清洗、数据聚合、数据统计等。
(3)Matplotlib: Matplotlib是Python中的数据可视化库,支持绘制各种类型的图表,如折线图、散点图、柱状图等。它提供了丰富的图形选项,可实现高质量的可视化效果。
(4)Seaborn: Seaborn是基于Matplotlib的数据可视化库,提供了更高级别的绘图接口和美观的图形风格。它支持各种类型的统计图表,如热力图、密度图、箱型图等。
(5)pyecharts: pyecharts是Python中的可视化库,支持各种类型的图表,如折线图、散点图、地图等。它提供了丰富的图形选项和交互功能,可实现高质量的可视化效果。
本研究就是利用Python的预处理模块和pyecharts可视化模块进行研究。
2.4 数据集介绍
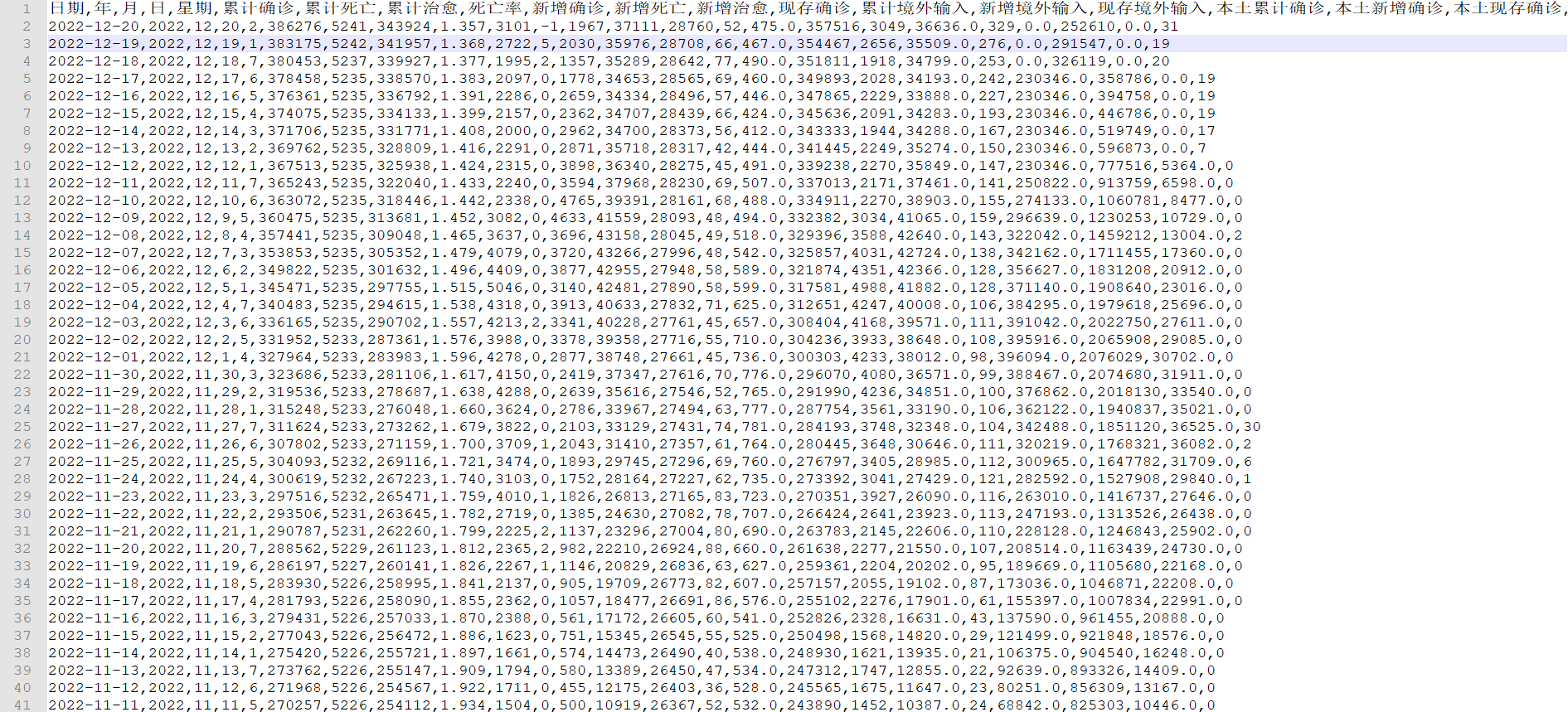
数据集包括多个字段,这些字段可用于对疫情情况进行详细分析。
日期:每天的日期
年:年份
月:月份
日:日期
星期:星期几
累计确诊:自疫情爆发以来,累计确诊的病例数量
累计死亡:自疫情爆发以来,累计死亡的病例数量
累计治愈:自疫情爆发以来,累计治愈的病例数量
死亡率:累计死亡病例数与累计确诊病例数之比
新增确诊:每天新增的确诊病例数
新增死亡:每天新增的死亡病例数
新增治愈:每天新增的治愈病例数
现存确诊:目前仍在治疗中的确诊病例数
累计境外输入:自疫情爆发以来,累计的境外输入病例数
新增境外输入:每天新增的境外输入病例数
现存境外输入:目前仍在治疗中的境外输入病例数
本土累计确诊:自疫情爆发以来,累计的本土确诊病例数
本土新增确诊:每天新增的本土确诊病例数
本土现存确诊:目前仍在治疗中的本土确诊病例数
现存重症:目前仍在治疗中的重症病例数
现存无症状:目前仍在隔离治疗中的无症状病例数
现存密切接触者:目前仍在隔离观察中的密切接触者数量
新增无症状:每天新增的无症状病例数
现存疑似:目前正在进行检测或观察的疑似病例数
数据集源自国内卫健委开放的数据集,并通过下载获得。这些数据对于深入研究新冠疫情的发展和趋势非常重要。通过分析数据集,可以深入了解疫情的传播情况、治愈率、死亡率、新增病例数等方面的趋势。使用Hadoop技术来处理这些数据,从而帮助更快速、更高效地完成数据处理和分析。
基于数据集,可以进行许多有趣的研究。例如,可以比较不同地区或国家的疫情数据,从而了解不同地区或国家的疫情情况和对策。还可以对数据进行可视化处理,从而更加生动地呈现疫情数据的趋势和分析结果。这有助于向公众传达疫情发展的情况,促进公众的防疫意识和采取有针对性的防疫措施。
总之,该数据集提供了许多有用的信息,可以用于深入了解疫情的情况和趋势,以及为制定有效的防疫策略提供支持。使用Hadoop技术进行数据处理和分析,将有助于更高效地利用这些数据,为疫情防控工作作出贡献。
|
图 3 源数据展示 |
2.5 数据预处理
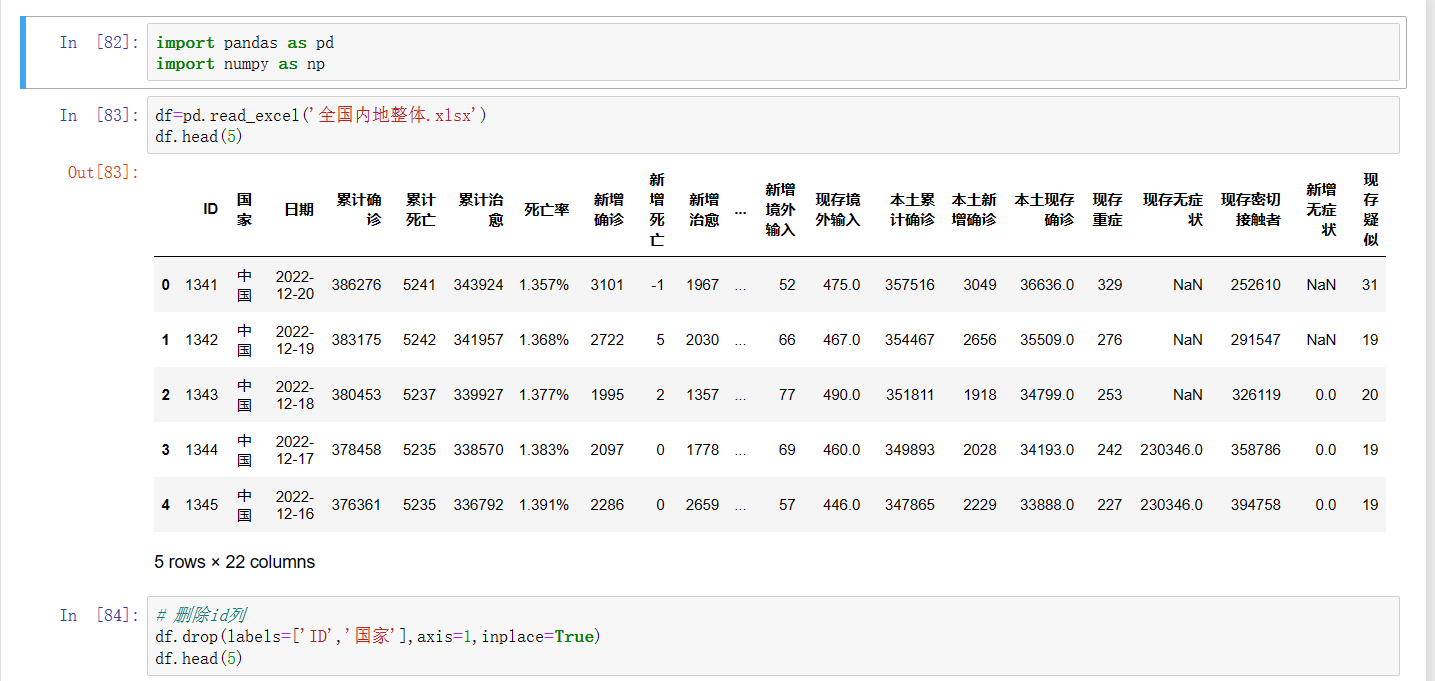
首先,对于公开提供的数据,我将其用Python的pandas导入到内存中,查看是否存在空值,由于保证数据的真实和有效性,并且结合时间上不能有太多的缺失值,所以这里我们采取填充的方法,将存在的空值填充为0即可,然后删除不重要的列,比如本数据就是研究国内的疫情数据,所以不需要该字段。此外,我们将时间字段进行扩充,将其转换为年、月、日、周的单列字段,便于后续在Hadoop中的hive里面进行大数据分析。
|
图 4 数据预处理展示 |
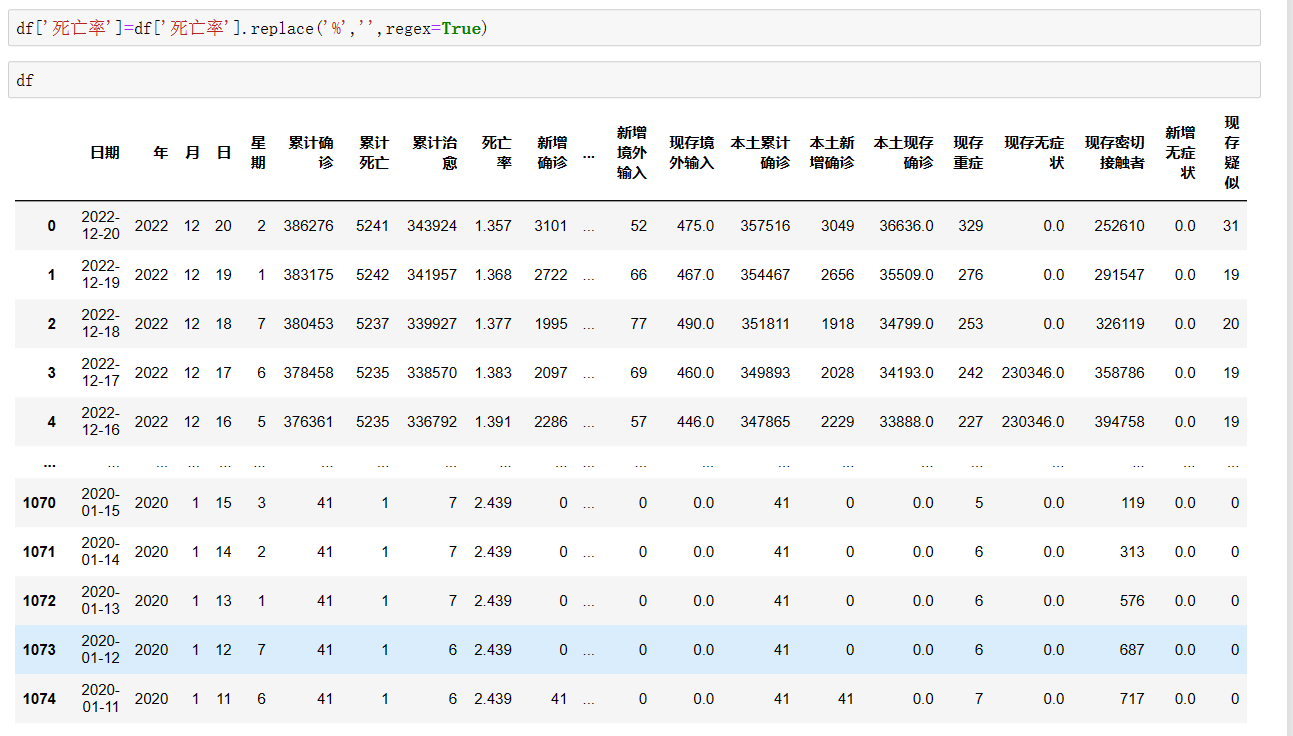
其次,我们将死亡率这个字段的百分比替换到,因为在hive中分析,是不允许出现这样的格式的,所以这里进行一步处理,采用正则表达式进行百分号的替换,使得数据变得有结构化。
|
图 5 预处理之后的数据 |
之后,为了在hive中创建合适的表,我们将所有的中文字段名,对应为英文,最后完成数据的预处理工作。
|
图 6 字段对应 |

2.6 Hive中创建表
在进行数据的导入之前,首先需要在hive中创建一个表,用于后续的flume的数据源接收。
|
图 7 Hive中创建表格 |
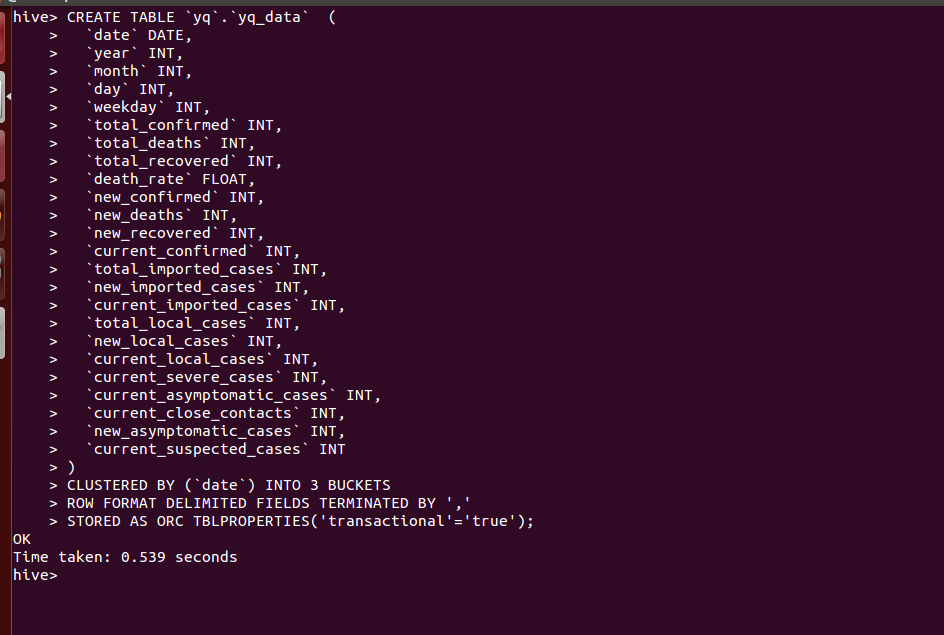
在Hive数据库中创建名为“yq_data”的表,并定义了该表的字段结构、分区方式、存储格式等属性。
首先,代码中使用CREATE DATABASE命令创建了名为“yq”的数据库,并使用USE命令将该数据库设置为当前默认数据库。接下来,使用CREATE TABLE命令定义了一个名为“yq_data”的表,包含了24个字段,这些字段分别对应疫情数据集中的不同指标,例如日期、年、月、日、累计确诊、累计死亡、累计治愈、死亡率、新增确诊、新增死亡、新增治愈、现存确诊、累计境外输入、新增境外输入、现存境外输入、本土累计确诊、本土新增确诊、本土现存确诊、现存重症、现存无症状、现存密切接触者、新增无症状、现存疑似等。
接下来,使用CLUSTERED BY命令将该表的数据按照日期进行分桶,分为3个桶,这有助于提高Hive查询数据的效率。然后,使用ROW FORMAT DELIMITED命令指定数据表中的字段分隔符为逗号,并使用STORED AS ORC命令指定该表的数据存储格式为ORC格式。最后,使用TBLPROPERTIES命令设置表属性,指定该表为事务表,支持Hive的ACID事务机制,确保数据的完整性和一致性。
这段代码的作用是在Hive数据库中创建一个名为“yq_data”的表,用于存储疫情数据集中的各个指标的数据。通过定义该表的字段结构、分区方式和存储格式等属性,可以帮助用户更方便地对疫情数据进行查询、统计和分析。同时,由于该表支持Hive的ACID事务机制,可以确保数据的安全性和完整性。
2.7 导入数据到hive中
接下来就是数据的导入,通过编写shell脚本一键化执行的,包括配置环境和配置参数变量。
首先,我们将配置flume需要的文件,达到一键化导入的效果
|
图 8 Flume配置文件 |
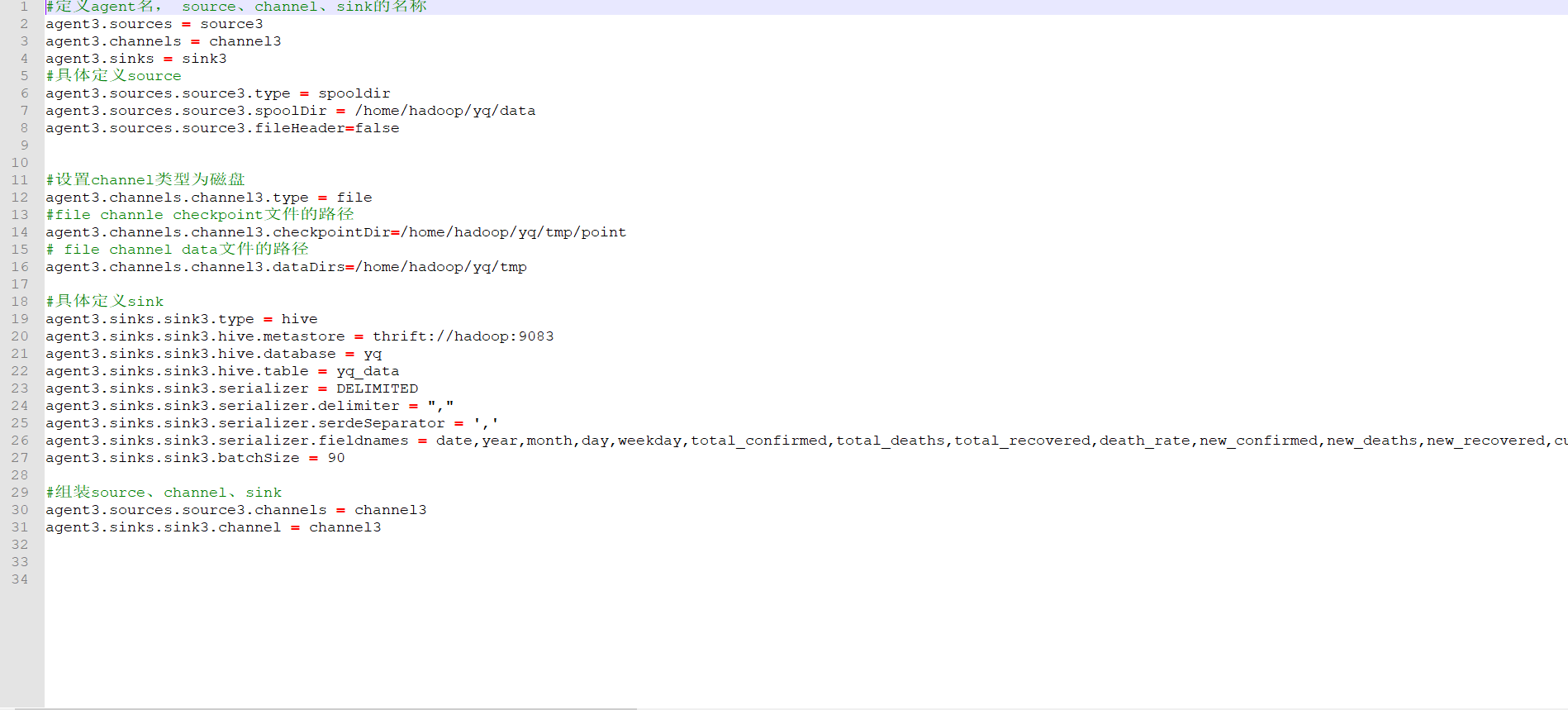
该文件用于将从指定目录下获取的文件数据写入到Hive表中。
在该配置文件中,首先定义了Flume的agent名称为agent3,其中包括了数据源source3、数据通道channel3以及数据汇sink3。然后,具体定义了source3数据源类型为spooldir,即从指定目录下获取数据,同时禁用了文件头的读取。接下来,定义了数据通道channel3类型为file,checkpointDir和dataDirs指定了通道checkpoint文件和数据文件的存储路径。最后,定义了数据汇sink3类型为hive,指定了Hive的元数据存储位置以及需要写入的数据库和表名,同时使用DELIMITED序列化方式,以逗号分隔字段,指定了需要写入表的字段名称,以及每批次写入的最大数据量。
最后,在组装source、channel、sink中,将source3和channel3绑定,并将sink3和channel3绑定,从而形成完整的数据流管道。
该配置文件的作用是为Flume提供了数据源、数据通道和数据汇的定义,以便从指定目录获取数据,并将数据写入到指定的Hive表中,实现数据的采集和处理。使用这个配置文件可以帮助用户快速搭建数据流管道,完成数据的采集和处理,提高数据处理效率和准确性。
然后我们配置好文件之后,新建两个文件夹便于后续的数据存放的位置,当我们将数据源移动到目标文件夹下之后,就会自动完成数据的导入。
前提是我们需要开启flume和hive监听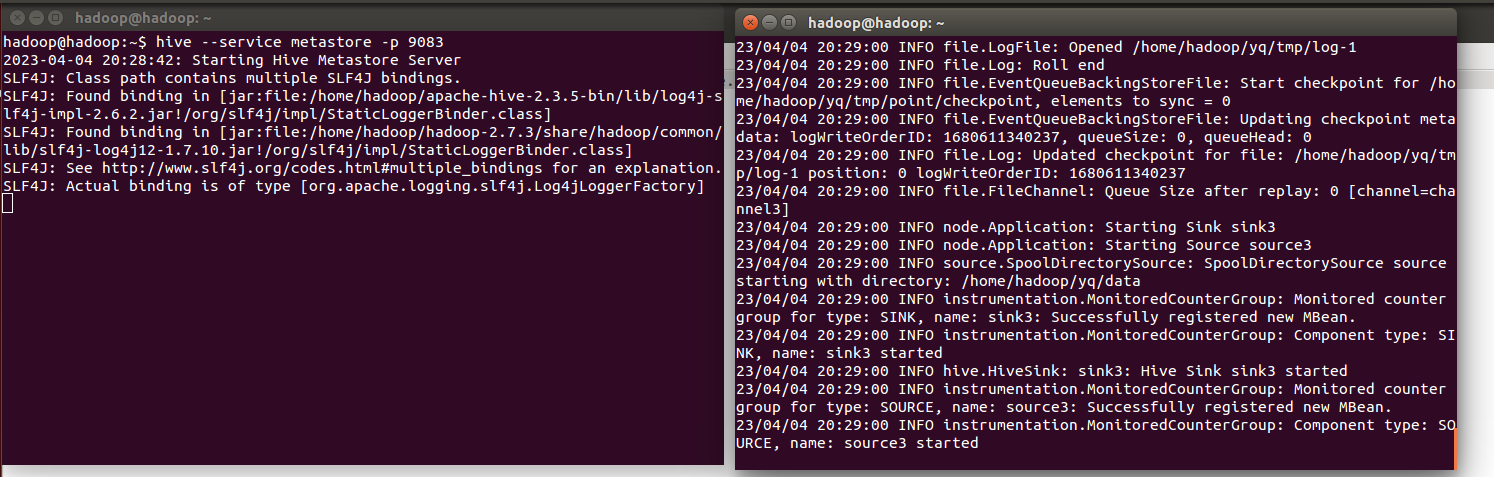 ,该命令行的Hive Metastore服务启动命令。
,该命令行的Hive Metastore服务启动命令。
Hive Metastore是Hive的元数据存储服务,用于管理和存储Hive中表、分区、数据等元数据信息。在Hive Metastore服务启动之后,Hive客户端可以通过连接Hive Metastore来获取元数据信息,并进行相关的查询和操作。
该命令中的--service metastore参数表示启动的是Hive Metastore服务,-p 9083参数指定了服务启动的端口号为9083。当执行该命令后,Hive Metastore服务会在后台启动,并开始监听指定的端口号。在Hive客户端中连接Hive Metastore服务的同时,就可以获取到相关的元数据信息,方便进行表和数据的查询和操作。
需要注意的是,启动Hive Metastore服务之前需要先启动Hadoop和Hive服务,以便建立与Hadoop和Hive服务的连接,并保证元数据的正确性和一致性。
其次:
(1)flume-ng agent:表示以agent模式启动Flume,即使用一个或多个数据源、通道和数据汇构成的管道。
(2)--conf conf:表示Flume配置文件所在的目录为conf目录。
(3)--conf-file file_hive.properties:表示需要加载的Flume配置文件为file_hive.properties文件,即指定了Flume所使用的配置文件。
(4)-name agent3:表示Flume agent的名称为agent3,即指定了agent的名称。
(5)-Dflume.hadoop.logger=INFO,console:表示指定Flume的日志级别为INFO,并将日志输出到控制台。
这条命令的作用是启动Flume的agent3,加载file_hive.properties配置文件,并输出INFO级别的日志信息到控制台,以便对Flume的运行状态进行监控和管理。这条命令通常用于在命令行中手动启动Flume agent,也可以将其作为脚本的一部分,实现自动化部署和管理Flume管道。
|
图 9 Flume导入数据展示 |
2.8 Hive大数据分析
2.8.1 每年累计新增疫情分析
我们对每日新增的数据按照每年进行分组聚合,统计累计总确诊、累计总死亡、累计总治愈、累计境外输入、累计无症状、累计本土确诊等人数进行可视化分析和展示。通过对疫情数据进行分组和聚合,从而得到不同年份的疫情数据汇总统计信息,便于人们了解疫情的发展趋势和规律,进一步指导疫情的防控和治疗。
|
图 10 hive大数据分析 |
在接下来进行的所有数据来源分析结果,都是通过Hadoop的hive进行大数据分析,并且将所得到的分析结果进行可视化展示出来,进一步让我们了解到这个疫情数据的规律。
|
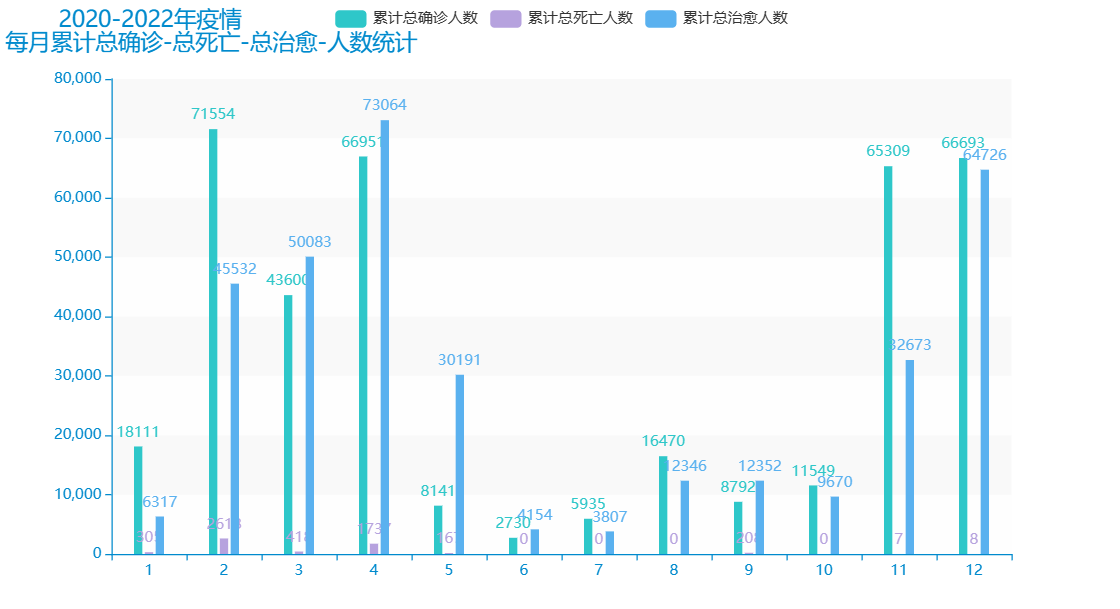
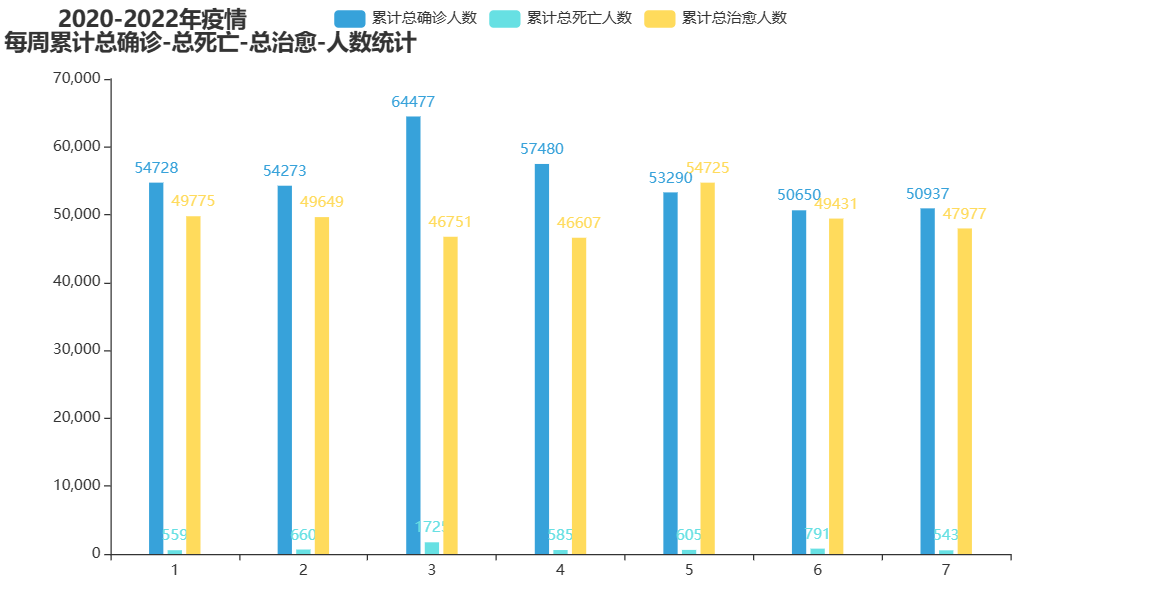
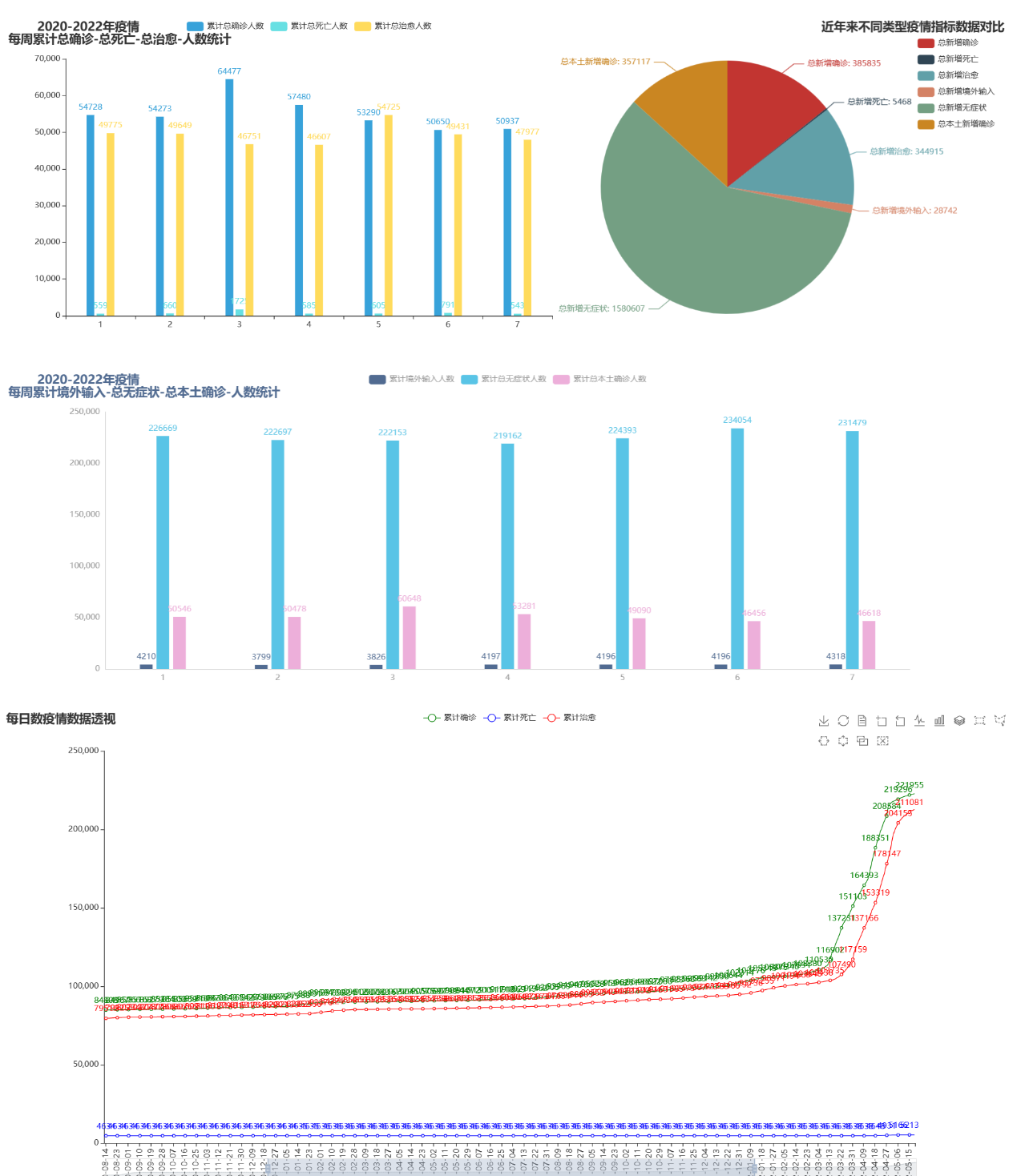
图 11 2020-2022年疫情累计总确诊-总死亡-总治愈人数统计 |
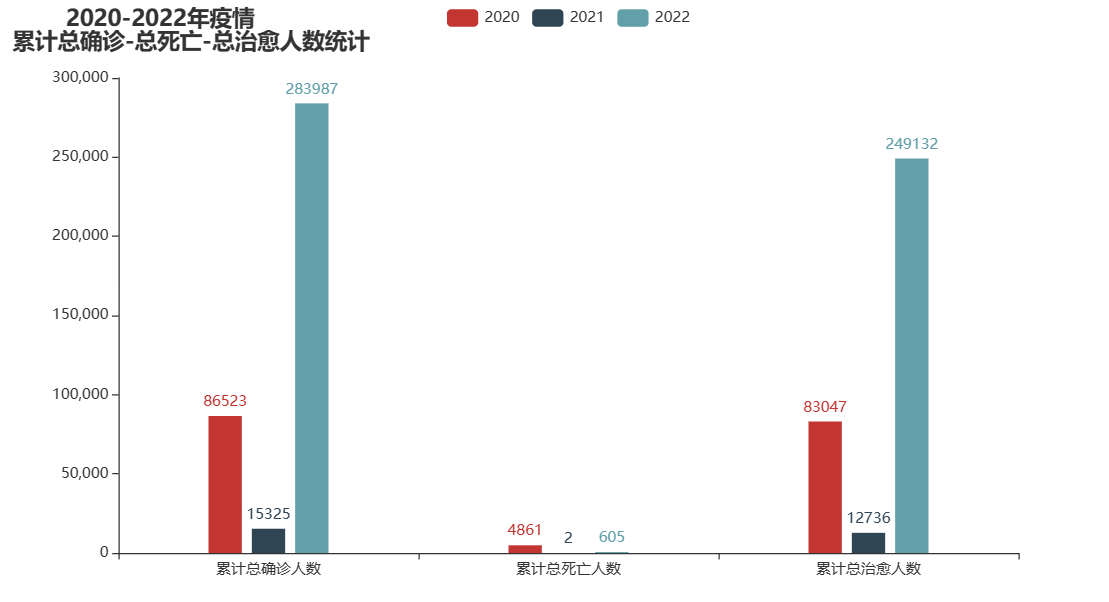
通过这里我们可以看到在2020年的时候,疫情数据相对于比较的平稳,到了2021年就呈现出比较好的一种态势,但是在2022年就出现了病毒的变异,导致国内疫情快速的上升。这里也可以看出我国在疫情防控方面做出了很大的努力,坚持以人民的生命安全为重要的,保障了我们的中国人民的生命安全。
|
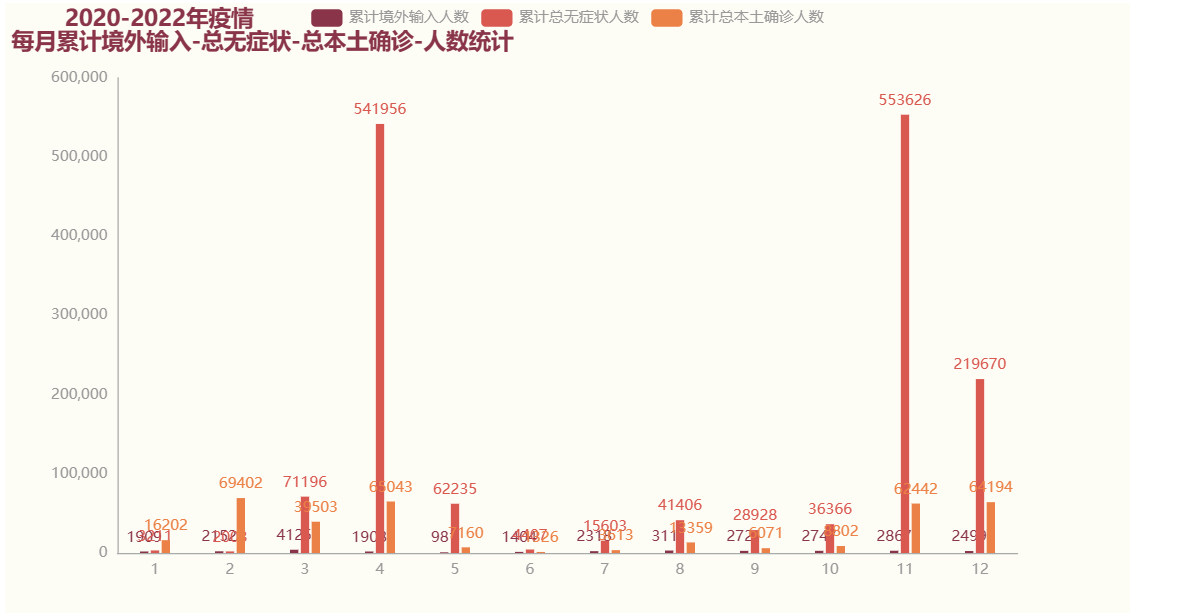
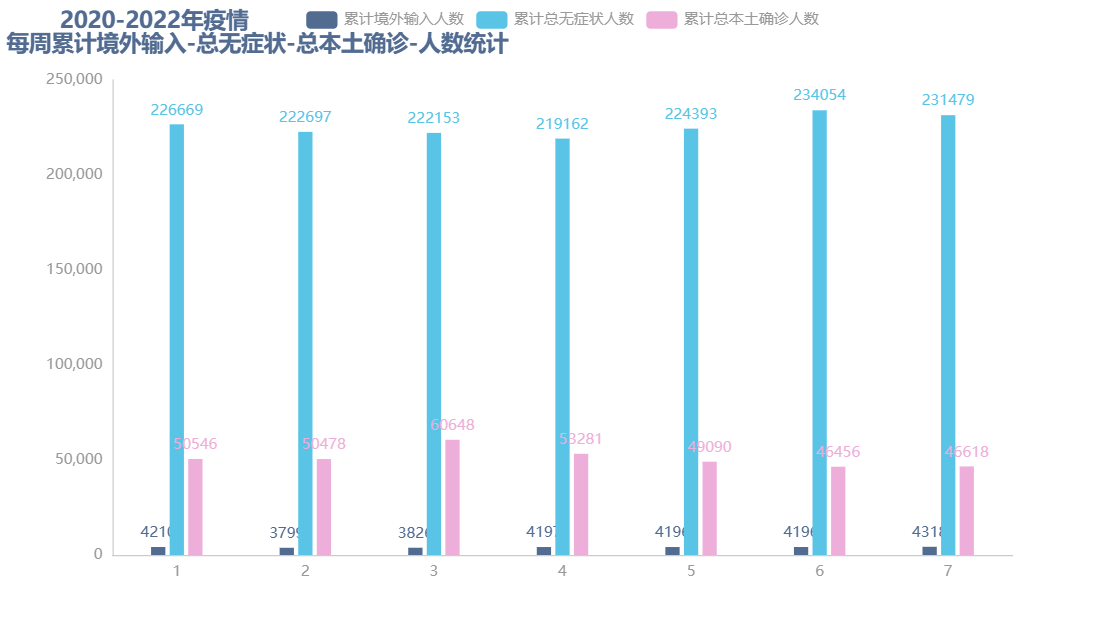
图 12 2020-2022年疫情累计境外输入-无症状、本土确诊人数统计 |
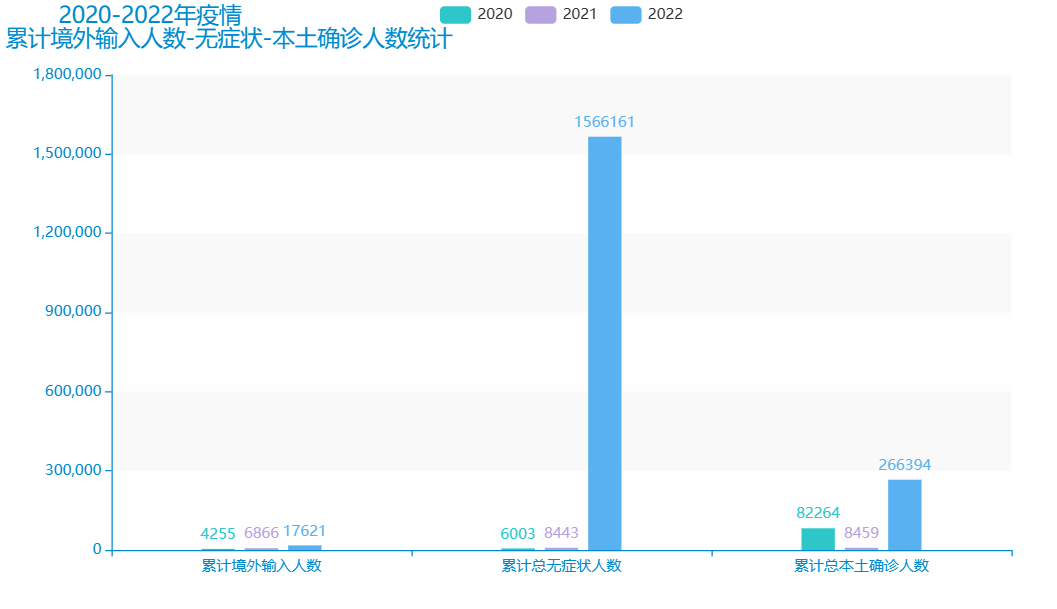
在2020年和2021年的时候境外输入的病例和无症状人数都是比较的少的,但是到了2022年的时候,国外疫情不断的反复变化,病毒也在不断的发生变异,随着而来的就是国外的疫情输入到国内了,但是我们国家在这方面做的很好,严防严控,保证在这样一个全球疫情急剧发生变化的大环境下,我们仍然可以保持一个稳定的态势,是非常不错的。对于无症状感染者,在2022年是非常多的,这也是当时国内新冠疫情的一个主要特点,无症状的人数非常的多。
2.8.2 每月累计新增疫情分析
针对国内的疫情数据,可以从每一个月来进行数据分析,找到近几年疫情高峰期和哪些月份相关,那么对应的月份包括有季节影响,时间影响、温度等其他的外在和社会因素影响,这些都是可以结合到每月的时间维度进行深入的思考和解释的。
可以用于深入了解疫情的发展和趋势,例如查看每个月的疫情情况和变化趋势,对比不同时间段的疫情数据,以及预测未来可能的疫情发展情况。这些查询结果可以用于制定更加有效的防疫策略和措施,促进公众的防疫意识,以及为医疗资源的配置提供支持。
|
图 13 2020-2022年疫情每月累计总确诊-死亡、治愈人数统计 |
从上面的数据可视化可以知道,在疫情高峰期主要是集中在春季和冬季两个时节,其中2、3、4月温度属于比较的温和的,而且也是属于开始上学和上班的一个时节,其中11和12月是属于回家过年的一个假期高峰期。那么通过上述的分析可知,在疫情发生之后,这种新增人数和我们周围的很多情况都会相关。
|
图 14 2020-2022年疫情每月累计境外输入-无症状、本土确诊人数统计 |
在4月和11月份的时候,无症状感染者数量非常多,并且在全部月份中都是占据非常大的比重的。其次就是12月份了,这也可以进一步的解释,在11和12月的时候大家都开始回家了,发生交叉感染的可能性非常大,所以需要进一步的防控这些。其次在4月的时候,需要注意佩戴好口罩,注意个人的疫情防控。
2.8.3 每日累计新增疫情分析
通过对近三年的每一天的数据进行可视化分析,找到在每个月中的每一天中有哪些时间比较容易发生疫情感染,并且可以找到这个其中的一些规律作为疫情防控的一个依据。
|
图 15 近三年平均每日疫情 |
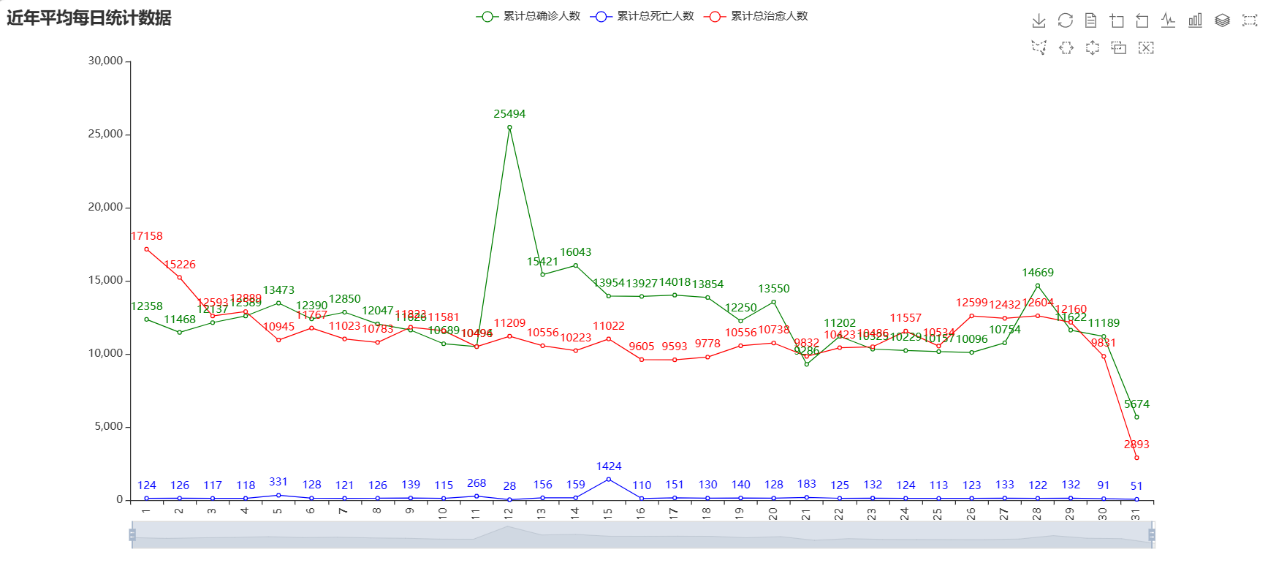
|
图 16 近三年平均每日疫情统计 |
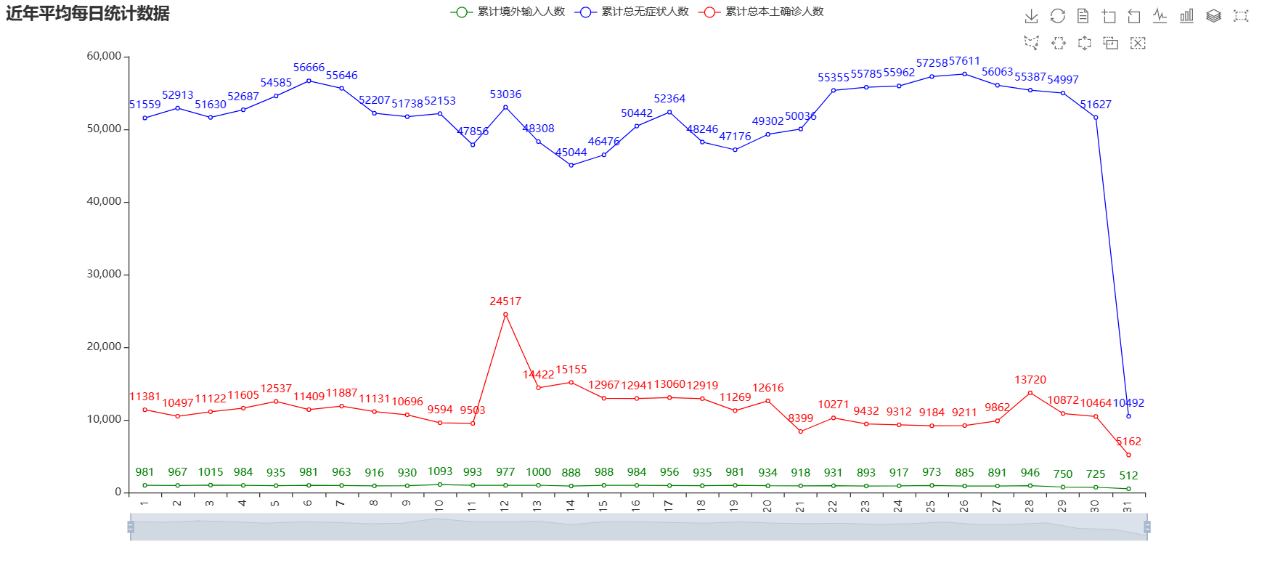
通过上述的分析和可视化,我们可以知道在一般月末的时候,这些疫情数据都有所缓和,一般是月末大家可能都在家中或者有其他的自己的事情,很少出门,这样也减少了疫情交叉感染的风险。
2.8.4 每周累计新增疫情分析
通过对近三年的每一周的数据进行分析和可视化,我们可以发现并且得出在一周之内,哪一天的感染人数是最多的,哪一天的感染人数是最少的。
|
图 17 2020-2022年疫情每周累计总确诊-总死亡-总治愈人数统计 |
从上述的分析可视化中,可以明显的看出,每周的总治愈人数都是差不多的,其中确诊人数主要是集中在工作日中,因为在工作日的时候大家经常进行核酸检测,所以对于确诊的数据有很好的监控。
|
图 18 2020-2022年疫情每周累计境外输入-无症状、本土确诊人数统计 |
对于每周中的无症状感染者人数,其中在周末的时候比较多,因为周末大家可能都想要出去玩或者去旅游,这样的情况就会增加无症状感染者的风险。
2.8.5 疫情总体对比数据分析
随着疫情的不断加剧,每天的疫情指标都在不断的上升,但是我们了解哪些指标在这几年中占据的主要的数量。
|
图 19 近三年不同疫情数据指标对比 |

通过可视化分析了解到,无症状感染者是最多的,其次就是确诊病例和治愈病例是持平的,在死亡率方面我们国家做的非常好,以及在防控境外输入的政策上都是比较的好的。
2.8.6 每日数据透视可视化
通过对每一天的疫情数据进行展示可视化可以更全面的了解疫情发展情况:通过每天的疫情数据可视化展示,可以更加全面、详细地了解疫情的发展情况和趋势,从而更好地制定相应的防疫措施。
更加直观的展现疫情数据:通过可视化展示,数据可以以图表、图形等形式呈现,更加直观、生动地展现疫情数据,有助于提高公众对疫情的关注和防护意识。
更好的发现数据规律和趋势:通过对疫情数据进行可视化,可以更加容易地发现数据之间的规律和趋势,例如新增病例数量、死亡率等变化趋势,这有助于进一步深入分析疫情发展情况,预测疫情的走向。
便于与其他数据进行对比和分析:通过可视化展示疫情数据,可以更加方便地将其与其他数据进行对比和分析,例如将疫情数据与人口数据、医疗资源数据等进行对比,有助于制定更加科学合理的防疫措施。
便于信息传播和共享:通过可视化展示疫情数据,可以更加便于信息传播和共享,例如通过社交媒体、网站等平台,向公众传达疫情发展情况和防疫措施,帮助公众更好地了解和应对疫情。
|
图 20 每天疫情数据展示 |

|
图 21 每日疫情数据透视 |
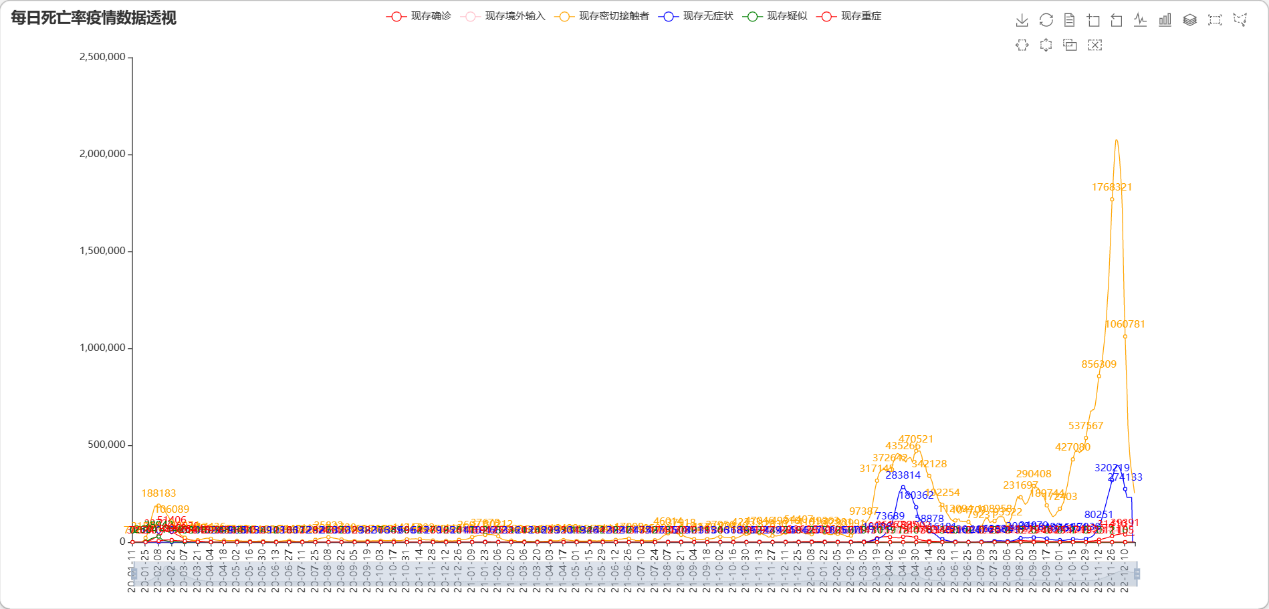
对每天的疫情数据进行时间维度的展示可以更加清晰地展现疫情的发展历程,有助于深入了解疫情的变化趋势和规律。可以通过对历史疫情数据的分析和预测模型的构建,进行未来疫情的预测。预测模型可以使用统计学、机器学习等方法来构建,从而预测未来的疫情趋势、疫情高发地区、疫情规模等方面的情况。但是需要注意的是,疫情的发展受到许多因素的影响,例如政府防疫措施、人口流动、病毒变异等,因此疫情预测结果并非完全准确,需要结合实际情况进行综合分析和判断。
2.8.7 疫情死亡率分析
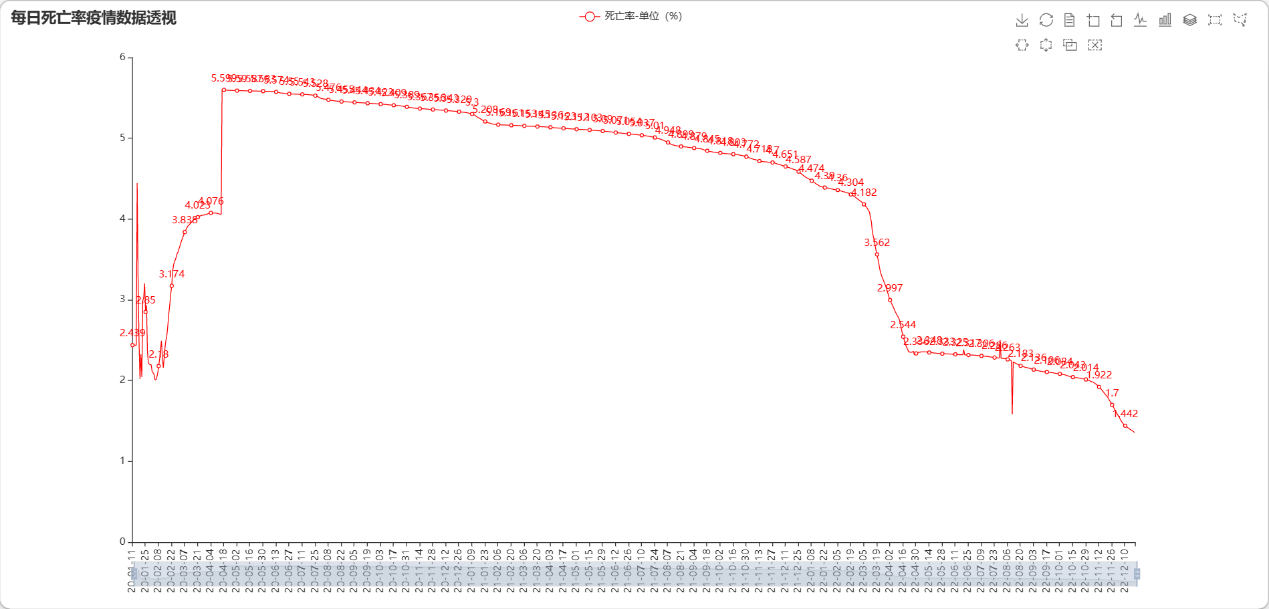
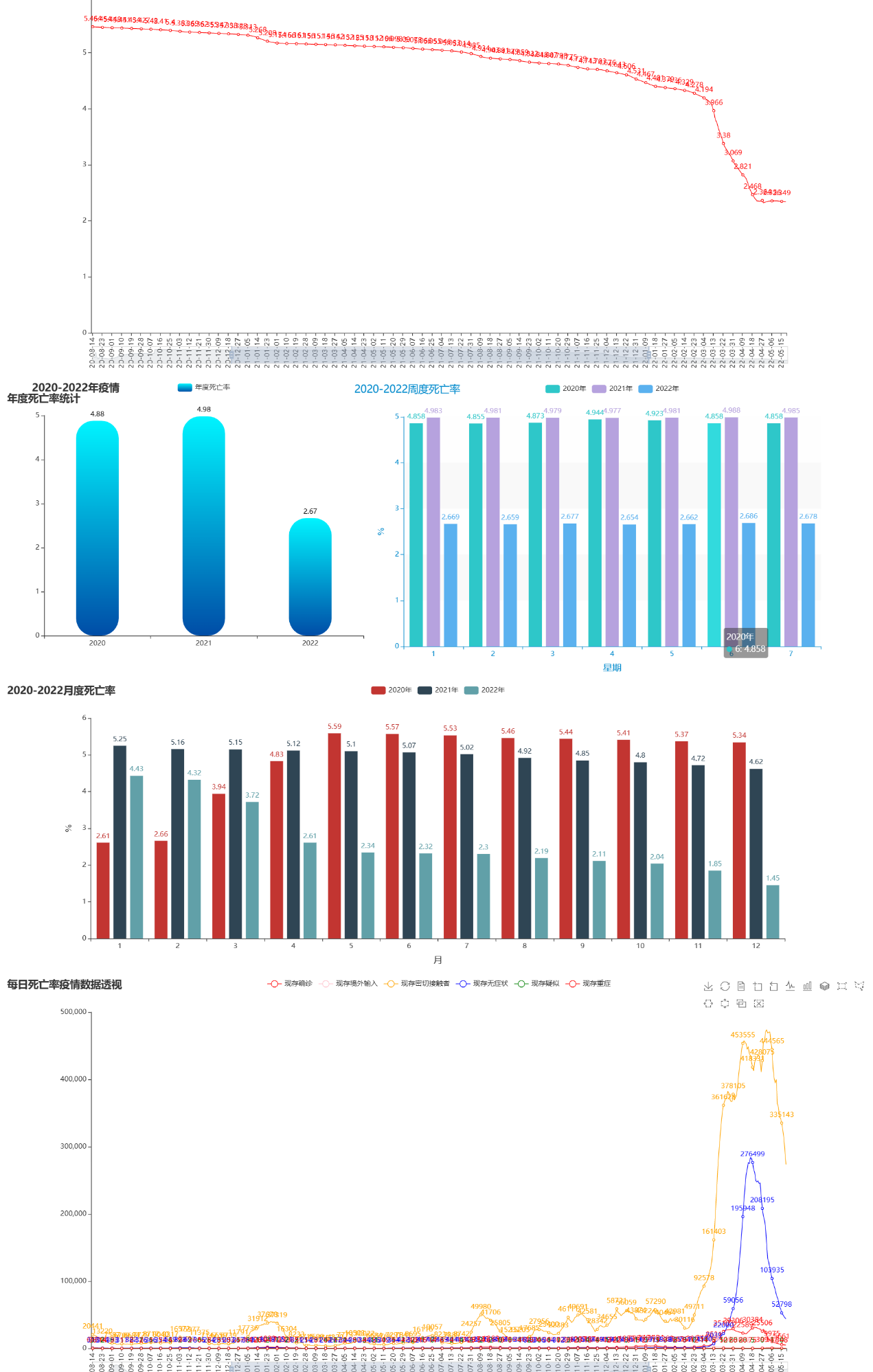
在疫情发生最初,死亡率开始上升,但是在后续的疫情防控政策越来越完善,疫情防控举措越来越大之后,死亡率逐步的开始下降,这也证明了我国在疫情防控方面做的是非常好的,也是值得其他国家借鉴的。
|
图 22 年度死亡率 |
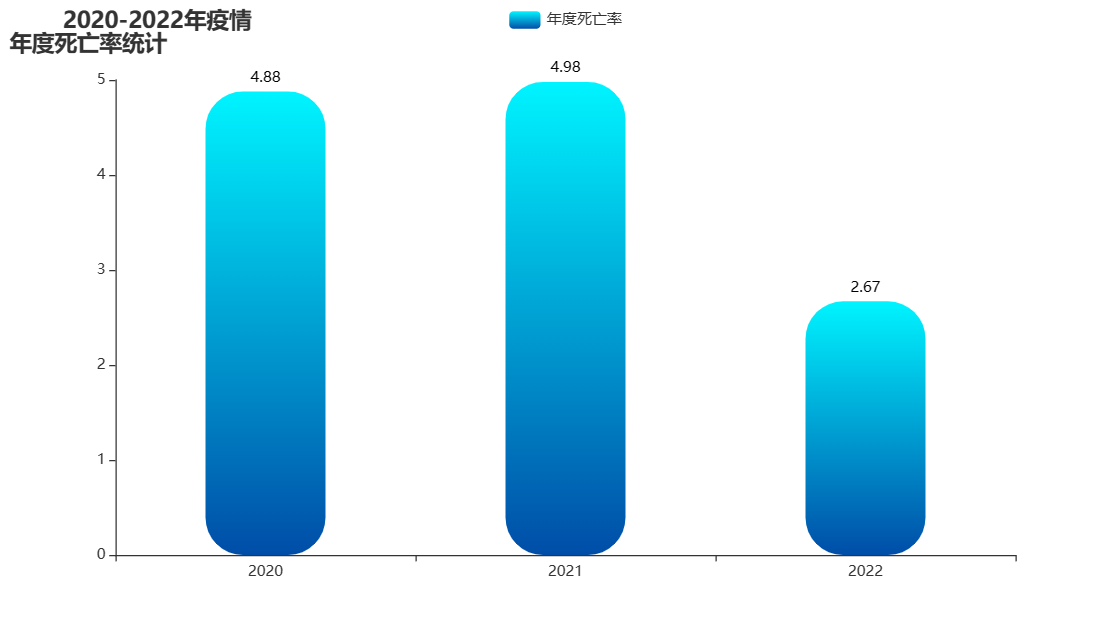
可以了解到在2021年和2020年的死亡率比较的高,但是到了2022年虽然疫情比较的严重,我们的死亡率还是下降的趋势,说明我们在防控疫情方面对人的生命安全做的比较的好。
|
图 23 月度死亡率 |
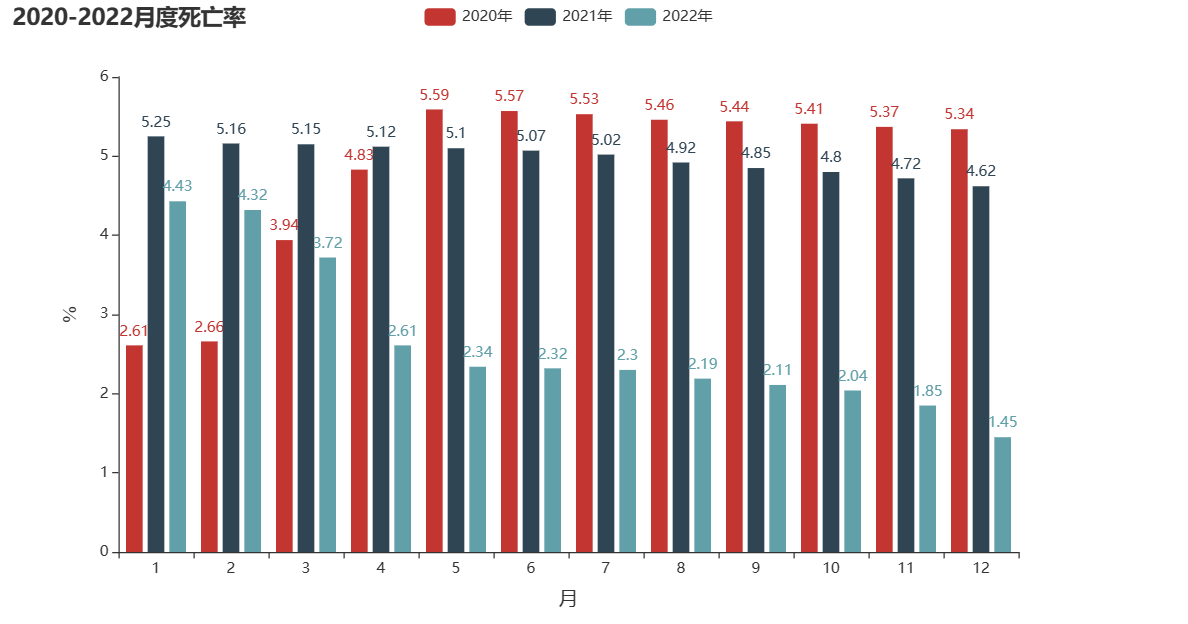
|
图 24 周度死亡率 |
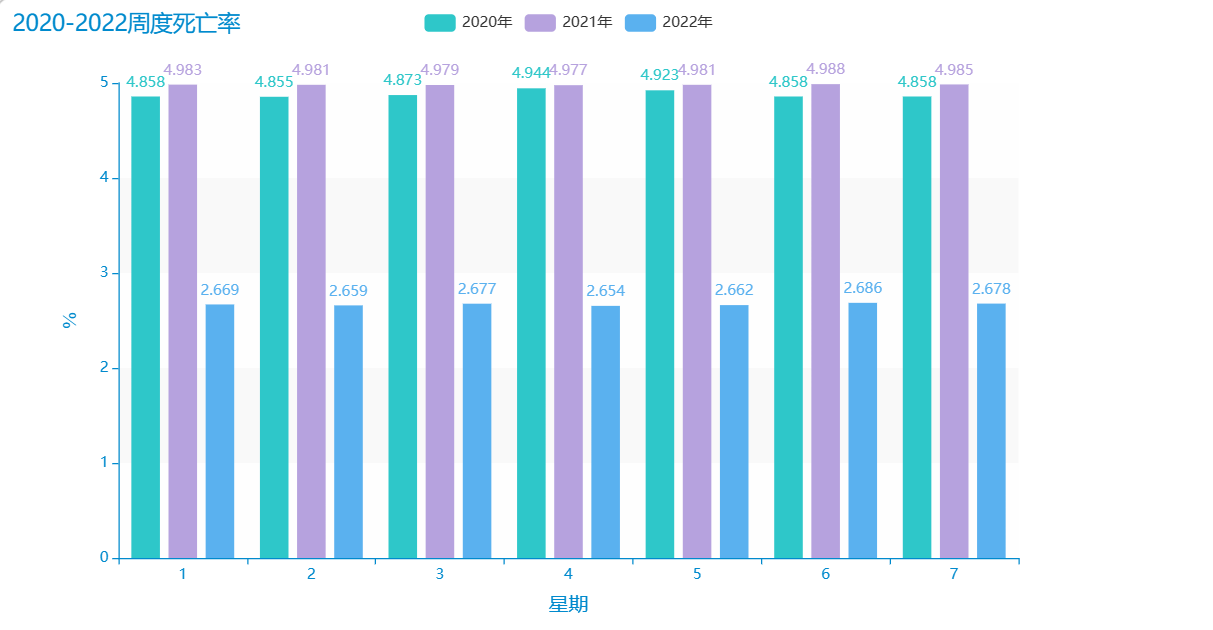
- 深入了解疫情发展情况:通过统计分析每一周的死亡率,可以更加深入地了解疫情的发展情况和趋势,从而及时采取针对性的防疫措施。
- 检验防疫措施的有效性:通过分析每一周的死亡率,可以判断之前采取的防疫措施是否有效,从而对防疫工作进行调整和优化。
- 比较不同地区的疫情发展情况:通过比较不同地区每一周的死亡率,可以了解各地疫情发展情况的差异,从而为针对不同地区的疫情制定相应的防疫措施。
- 预测疫情未来发展趋势:通过统计分析每一周的死亡率,可以预测未来疫情的发展趋势,例如疫情规模、疫情高峰期等,从而为疫情防控工作提供科学依据和指导。
促进公众防疫意识的提高:通过对每一周的死亡率进行统计分析,并向公众传递相应的信息,可以提高公众的防疫意识,增强公众的防疫自觉性和主动性,从而有效遏制疫情的传播。
2.9 大屏可视化
构建疫情大屏可视化是一种将疫情数据可视化展示的方式,它将大量的数据以图表、地图等形式呈现在屏幕上,便于人们直观地了解疫情的发展情况和趋势。疫情大屏可视化的好处如下:
首先,疫情大屏可视化可以使数据更加直观、易懂。通过大屏幕的形式将疫情数据以各种图表、地图等形式展示出来,使人们可以一目了然地看到疫情发展情况、趋势以及各地的疫情情况,从而有利于人们更好地了解疫情发展情况。
其次,疫情大屏可视化可以加强疫情信息的传播和共享。通过疫情大屏可视化,人们可以随时随地获取最新的疫情信息,便于大家及时了解疫情情况,加强对疫情的防控。此外,疫情大屏可视化还可以将数据实时同步,便于数据的共享和传播。
再次,疫情大屏可视化可以帮助决策者更好地制定防疫措施。通过疫情大屏可视化,决策者可以直观地了解疫情发展情况、趋势以及各地的疫情情况,从而更加准确地制定相应的防疫措施,有针对性地开展疫情防控工作。
最后,疫情大屏可视化还可以提高公众的防疫意识。通过将疫情数据可视化展示在大屏幕上,可以提高公众的防疫意识,增强公众的防疫自觉性和主动性,从而有效遏制疫情的传播。
总之,疫情大屏可视化是一种极具实用性和前瞻性的疫情数据展示方式。通过大屏幕的形式展示疫情数据,可以更好地了解疫情发展情况和趋势,促进疫情信息的传播和共享,有助于决策者制定相应的防疫措施,提高公众的防疫意识,为疫情防控工作提供更加有效的支持。
|
图 25 疫情可视化大屏1 |
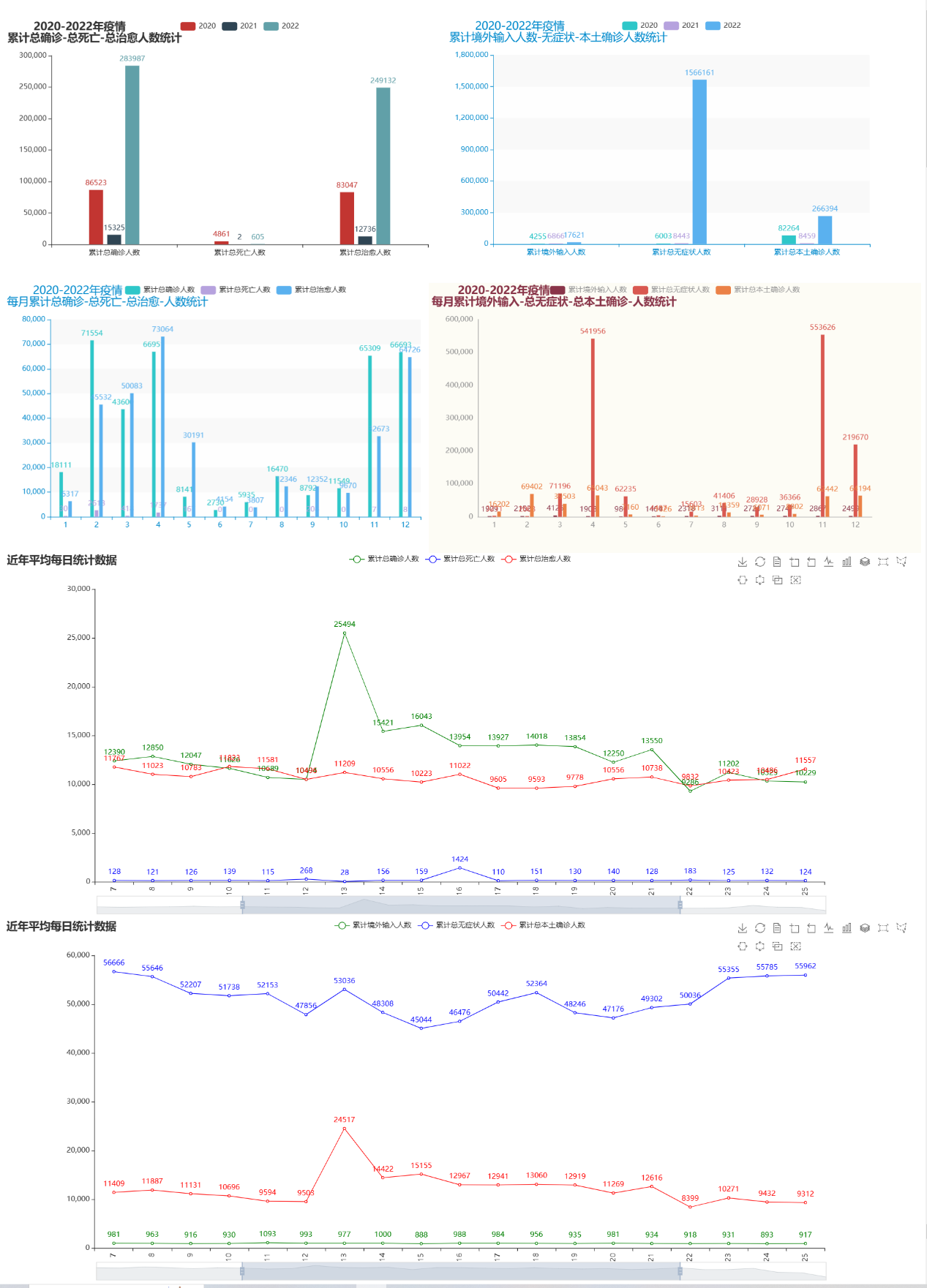
|
图 26 疫情可视化大屏2 |
|
图 27 疫情可视化大屏3 |
通过pyecharts中的page构建疫情可视化大屏的优势如下:
(1)可交互性:page提供了疫情数据可视化大屏的交互功能,用户可以通过鼠标点击或者其他操作实现对展示数据的交互,增强用户体验,让用户更加深入地了解疫情数据。
(2)丰富多样的展示效果:pyecharts中的page提供了多种图表、地图等展示效果,并且支持自定义展示效果,可以让用户选择最适合自己的展示方式,同时展示效果也很炫酷,让用户更加容易吸引眼球。
(3)可视化程度高:page提供了可视化大屏的搭建框架,可以将疫情数据以图表、地图等形式直观展示,使数据更加具有可读性和可视化,让用户更加容易理解疫情数据和发展情况。
(4)操作简便:pyecharts中的page操作简便,用户只需简单配置即可搭建一个交互式、丰富多彩的疫情数据可视化大屏,不需要具备复杂的编程技能和相关经验,即可实现数据可视化的展示。
(5)高度可定制性:pyecharts中的page支持自定义主题、背景、字体等多种风格,同时也支持多语言展示,可以根据用户的需求和偏好进行个性化设置,增加疫情数据可视化的艺术性和实用性。
总之,通过pyecharts中的page构建疫情可视化大屏,可以让疫情数据更加具有可读性和可视化,丰富多样的展示效果和交互功能可以提高用户体验,同时操作简便和高度可定制性也增加了数据展示的艺术性和实用性,为疫情防控工作提供更加有效的支持。
3 总结与分析
3.1 本研究创新之处
本研究的创新之处在于:
1、综合运用了多种大数据技术。本研究利用了Hadoop、Flume、Hive等多种大数据技术,将数据预处理、自动加载、分析、可视化等环节有机地结合起来,实现了一体化的数据分析和可视化展示,为疫情防控提供了全方位的支持。
2、结合多维度的数据进行分析。本研究从时间、空间、横向和纵向等多个维度对疫情数据进行了分析,深入挖掘了疫情数据的特点和规律,为疫情防控提供了更加全面、准确的参考依据。
3、利用Python的可视化库实现了交互式可视化展示。本研究利用Python的pyecharts可视化库,编写了交互式的可视化展示代码,将数据分析结果以图表、地图等形式进行展示,使得疫情数据的可视化展示更加生动、直观、易懂,有助于公众更好地了解疫情发展情况。
4、构建了疫情可视化大屏。本研究将可视化结果通过Python的page组件进行组合,构建了疫情可视化大屏,实现了疫情数据的实时展示和动态更新,方便决策者和公众及时了解疫情情况,为疫情防控提供了重要的参考依据和支持。
总之,本研究在数据分析和可视化展示方面具有较高的创新性,通过多维度的数据分析和交互式的可视化展示,为疫情防控提供了更加全面、准确和有力的支持,具有一定的实践意义和推广价值。
3.2 本研究不足之处
本研究虽然对近三年的疫情数据进行了大数据分析,但在分析过程中还存在一些不足之处。
首先,本研究数据集只基于中国新冠疫情2020-01-11至2022-12-20的全国整体数据进行分析,没有对各个省份、城市等地区的数据进行分析。因此,本研究在空间维度上的分析相对不足,不能深入了解各个地区的疫情情况。
其次,本研究没有针对不同的人群特征进行分析。由于不同年龄段、性别、职业等人群特征对疫情的传播和影响不同,因此分析不同人群特征的数据可以更加深入地了解疫情的传播特点和规律。
另外,本研究对预测模型的构建相对简单,没有使用更加复杂的算法来构建模型,从而可能会对预测结果的准确性产生一定的影响。
最后,本研究在可视化展示方面,只是结合Python的pyecharts可视化库进行了基本的可视化展示,并没有使用更加专业的可视化工具和技术,可能会影响可视化效果和交互性。
综上所述,本研究在空间分析、人群特征分析、预测模型构建和可视化展示等方面还存在一些不足之处,需要进一步深入研究和完善。
3.3 总结
本研究基于中国新冠疫情2020-01-11至2022-12-20的全国整体数据进行疫情大数据分析,通过对历史的数据进行大数据分析,可以有效的掌握过去疫情数据的特点,便于对疫情之后的防控提供更好的决策。
首先,在数据处理方面,本研究对原始数据进行了预处理和清洗,包括空值处理、时间字段扩充等,以便后续的大数据分析。其次,通过配置好Flume和Hive环境并编写Shell脚本,将处理好的数据集自动加载到Hive数据库中,实现了一键化的数据导入。
其次,在数据分析方面,本研究从时间、空间、横向和纵向等多个维度对疫情数据进行了分析,挖掘了疫情数据的特点和发生的时间上的分布情况。在分析过程中,本研究利用了Hive等大数据分析工具,运用了多种分析方法,包括聚类分析、时间序列分析等,以全面地了解疫情数据的特征和规律。
最后,在数据可视化方面,本研究结合Python的pyecharts可视化库,编写了可视化代码进行前端可视化展示,并利用page组件将图表进行组合,搭建疫情可视化大屏,构建交互式的动态可视化展示。通过疫情可视化大屏,可以直观地了解疫情发展趋势、地域分布情况等,为决策者提供科学依据,同时提高公众的防疫意识。
综上所述,本研究基于Hadoop和Python的预处理、自动加载数据、大数据分析、可视化展示与构建可视化大屏,实现了一体化的效果,为疫情防控提供了重要的参考和决策支持。同时,本研究还展现了大数据技术在疫情防控领域中的应用价值,具有一定的推广和应用前景。
每文一语
回首过去总会充满遗憾,向前看才会充满希望