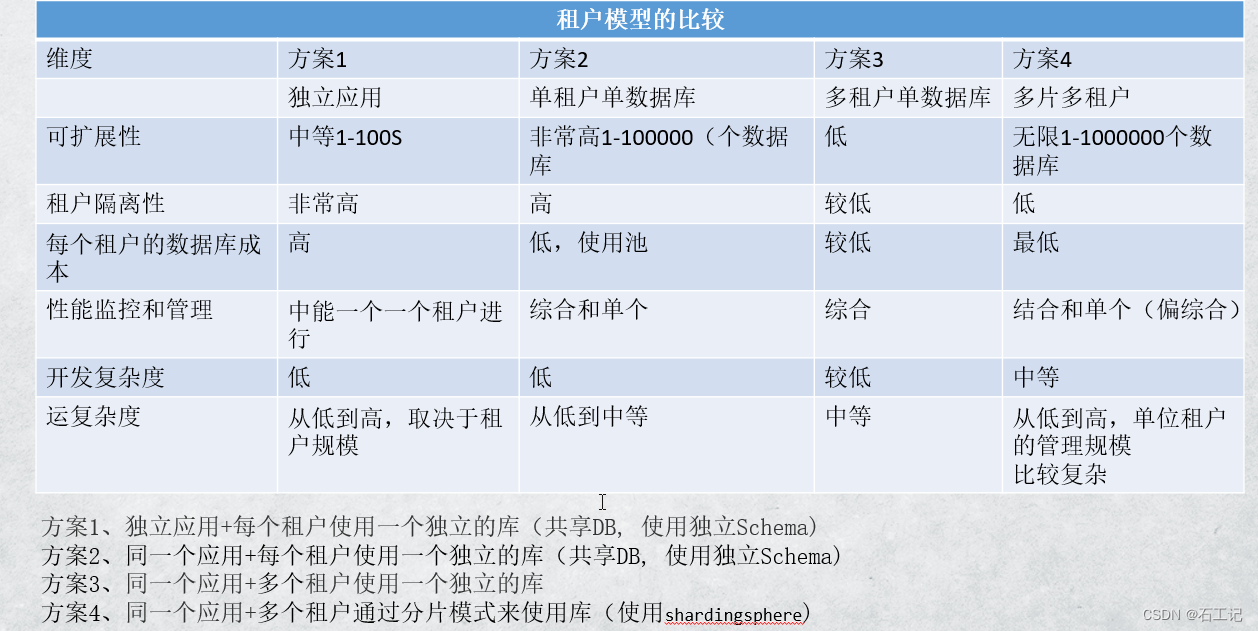
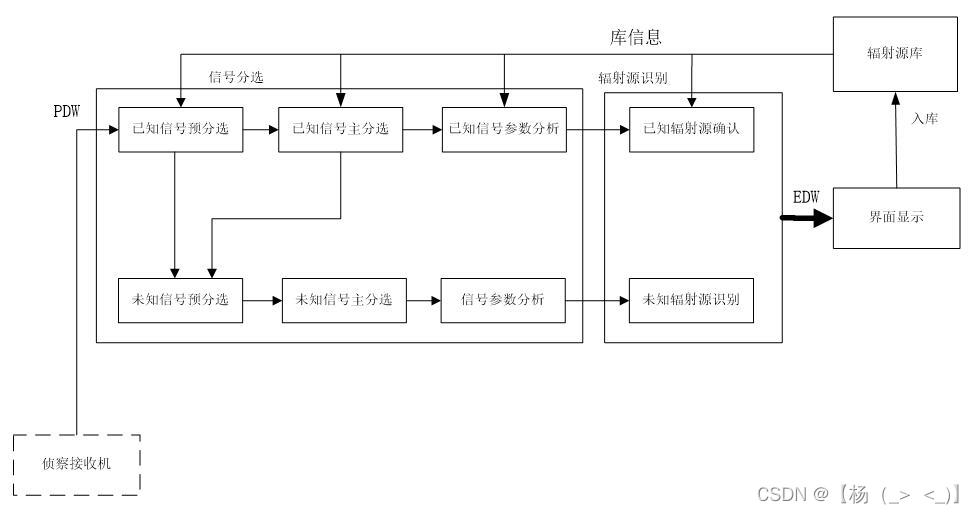
我在122服务器上测试了FullMouseBrain的数据集,发现内存爆炸
print("................................................import package ....................................")
import os
import sys
#########################################################################################################################
#########################################################################################################################
#########################################################################################################################
##########Attention1: Because I want to classify my result to different folder,so I set this ############################
#os.chdir("/Users/xiaokangyu/Desktop/tDCA_project/tDCA/")
cur_dir=os.getcwd()
sys.path.append("/DATA2/zhangjingxiao/yxk/tDCA_multi_sample/tDCA/") # you should change this path to fit your own system
#########################################################################################################################
#########################################################################################################################
#########################################################################################################################
import pandas as pd
import scanpy as sc
import numpy as np
import matplotlib.pyplot as plt
import matplotlib
import argparse
from scipy.sparse import issparse
from IPython import get_ipython
if("ipykernel" in sys.modules):
get_ipython().run_line_magic('matplotlib', 'inline')
else:
matplotlib.use('Agg')
os.system("clear")# clean screen in vscode,not os.system("clean")
from sklearn.metrics.cluster import adjusted_rand_score,normalized_mutual_info_score
from sklearn.metrics import silhouette_score
import torch
from torch.autograd import Variable
from time import time # calculate running time
##################
from preprocess import read_dataset,normalize
from tDCA import tDCA## use DCA autoencoder model
from tAE import tAE ## use normal Autoencoder model
from utils import *
from loss import *
from train import train_model
from time import time # record time
#from torchsummary import summary
# run pip install torchsummary---->print number of parameters and model like model.summary() in keras
##########################################################################################################################
if __name__ == "__main__":
##########################################set parameters in command line##################################################
parser = argparse.ArgumentParser(description="set parameters for tDCA", formatter_class=argparse.ArgumentDefaultsHelpFormatter)
parser.add_argument("--dataset", default = "dataset", type = str,help="dataset name")
parser.add_argument("--datatype",default="single",type=str,choices=["single","multi"],help="type of dataset(single or multi)")
parser.add_argument("--highly_genes", default = 1000, type=int,help="number of highly variable genes")
parser.add_argument("--pretrain_epochs", default = 50, type=int,help="maximum epochs in pretrain stage")
parser.add_argument("--batch_size",default=256,type=int,help="batch size for tDEC training")
parser.add_argument("--save_raw",default=0,type=int,help="visulize raw dataset") ## use integter
parser.add_argument("--resolution",default=0.5,type=float,help="default resolution selected in louvain algorithm")
parser.add_argument("--n_cluster",default=4,type=int,help="n cluster set for louvain algorithm in DEC clustering stage")
parser.add_argument("--K",default=20,type=int,help="K nearest neighghour choosen in calulating KNN and MNN")
parser.add_argument("--save_corrected",default=1,type=int,help="whether to save final embedding to file")
parser.add_argument("--plot_MNN",default=0,type=int,help="whether to plot MNN pair")
parser.add_argument("--batch_name",default="BATCH",type=str,help="string indicate batch information in dataset")
parser.add_argument("--interval",default=100,type=int,help="interval to visulize the embedding in pratrain stage")
parser.add_argument("--Lambda_tri",default=1.0,type=float,help="weight of Adaptived triplet loss")
parser.add_argument("--Lambda_recon",default=0.1,type=float,help="weight of reconstruction loss")
parser.add_argument("--Gamma",default=2.0,type=float,help="weight of AP(anchor positive) and AL(anchor limit) in Adapted Triplet Loss")
parser.add_argument("--do_tsne",default=1,type=int,help="Do TSNE plot in final embedding")
parser.add_argument("--do_umap",default=1,type=int,help="Do UMAP plot in final embedding")
parser.add_argument("--reproduce",default=1,type=int,help="whether to reproduce the result")
parser.add_argument("--seed",default=1,type=int,help="random seed to reproduce result")
parser.add_argument("--dec_clustering",default=0,type=int,help="whether to apply dec clustering after triplet learning")
args = parser.parse_args("")
dataset=args.dataset
datatype=args.datatype
n_hvg=args.highly_genes
num_epochs = args.pretrain_epochs
batch_size = args.batch_size #
save_raw=args.save_raw
reso=args.resolution
n_cluster=args.n_cluster
k=args.K
save_corrected=args.save_corrected
plot_MNN=args.plot_MNN
batch_name=args.batch_name
interval = args.interval
lamb_tri=args.Lambda_tri
gamma=args.Gamma
lamb_recon=args.Lambda_recon
do_tsne=args.do_tsne
do_umap=args.do_tsne
reproduce=args.reproduce
seed=args.seed
dec_clustering=args.dec_clustering
############################################################################################################################################
############################################################################################################################################
############################################################################################################################################
############################################################################################################################################
###############################################different setting for different dataset######################################################
print("...................................................................................................")
print(".........................................read data.................................................")
adata = sc.read("/DATA2/zhangjingxiao/yxk/dataset/FullMouseBrain/FullMouseBrain_raw.h5ad")
print(adata)
dataset="FullMouseBrain"
datatype="single" #
n_cluster= 13
############################################################################################################################################
############################################################################################################################################
############################################################################################################################################
############################################################################################################################################
############################################################################################################################################
if(issparse(adata.X)):
adata.X=adata.X.toarray()
adata.raw=adata ## speed is slowly when training sparse matrix withn DCA model
if(datatype=="single"):
batch_name=None
adata.obs["BATCH"]="1" # convenient for plot
else:
batch_name="BATCH"# BATCH in your adata.obs
print("..................................read data done...................................................")
print("...................................................................................................")
######################################################################################################
print(".......................Basic information about parameter setting...................................")
print("dataset={}".format(dataset))
print("datatype={}".format(datatype))
print("number of hvg={}".format(n_hvg))
print("total training epochs={}".format(num_epochs))
print("batch_size={}".format(batch_size))
print("resolution={}".format(reso))
print("wheather to plot and save visulization of raw data:{flag}".format(flag="True" if save_raw else "False"))
print("final number of cluster={}".format(n_cluster))
print("K nearest neighour={} (knn,mnn)".format(k))
print("wheather to save corrected embedding by tDEC:{flag}".format(flag="True" if save_corrected else "False"))
print("wheather to plot MNN pair of dataset:{flag}".format(flag="True" if plot_MNN else "False"))
print("{} indicate the batch id in dataset".format(batch_name))
print("interval={}".format(interval))
print("weight of triplet loss:{}".format(lamb_tri))
print("weight of reconstruciton loss:{}".format(lamb_recon))
print("weight of AP(anchor positive) and AL(anchor limit) in Adapted Triplet Loss:{}".format(gamma))
print("do tsne after final training:{flag}".format(flag="True" if do_tsne else "False"))
print("do umap after final training:{flag}".format(flag="True" if do_umap else "False"))
print("reproduce with training:{flag}".format(flag="True" if reproduce else "False")) ####
print("Do dec clustering after training:{flag}".format(flag="True" if dec_clustering else "False")) ######
##########################initialization for addtional setting###############################
print("...............................initialization for addtional setting................................")
method="tDCA"
print("method={}".format(method))
ae_weight_file='AE_weights.pth.tar' ## not implemented now
ae_weights=None ## not implemented now
save_dir=cur_dir+"/output/"+dataset
sc.settings.figdir=save_dir
seed_torch(1)# for reproduce
device = torch.device("cuda" if torch.cuda.is_available() else "cpu")
print("device={}".format(device))
if not os.path.exists(save_dir):
os.makedirs(save_dir)
filelist = [ f for f in os.listdir(os.path.join(os.getcwd(),save_dir))]
for f in filelist:
os.remove(os.path.join(os.getcwd(),save_dir, f))
###############################################################################################
#########################data preprocessing ###################################################
time_start=time()
print("...................................................................................................")
print("....................................data preprocessing.............................................")
adata = read_dataset(adata,
transpose=False,
test_split=False,
copy=True)
adata = normalize(adata,
size_factors=True,
normalize_input=True,
logtrans_input=True,
select_hvg=True,
n_hvg=n_hvg,
subset=True,max_value=10.0)
结果这里就出现了问题