为什么要写这篇文章:
最近因为朋友需要用到MCScanX画两个物种的共线性点图,但是发现搜到的blog中所提供的安装方法都不太相同,且在都会出现或多或少的问题,所以来找我帮忙,我搜到的所有blog安装链接http://chibba.pgml.uga.edu/mcscan2/MCScanX.zip其实都已经不维护了。总体安装的很艰难,所以想把自己的安装经历和大家分享一下,以便其他人少走弯路。
下载安装包以及需要的环境
需要我们自行下载并解压安装的主要有三个文件 Zlib库文件、libpng文件、MCScanX以及一些依赖环境。
-
依赖环境:
以下依赖环境有的话就不用再装了,不知道有没有的话可以试着输入以下,有的话会弹出用法。
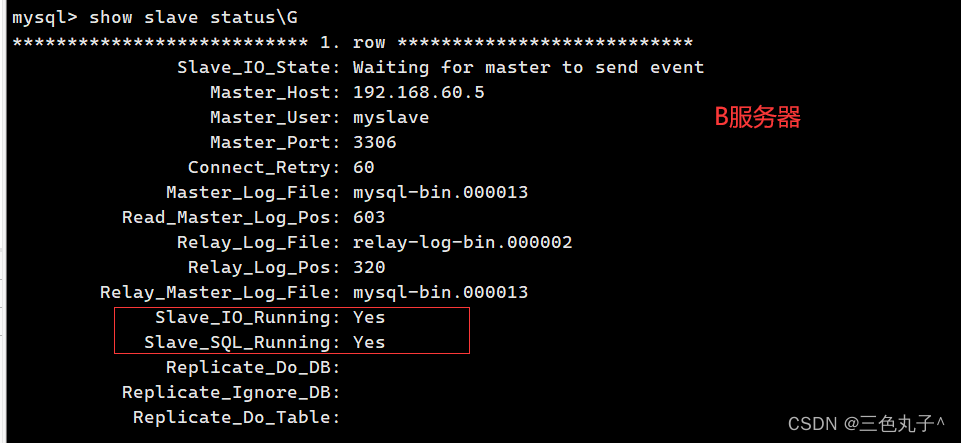
例如:(base) ght@DESKTOP-TU3M5B7:~$ makeblastdb # 如果没有的话会弹出安装方法 Command 'makeblastdb' not found, but can be installed with: sudo apt install ncbi-blast+(base) ght@DESKTOP-TU3M5B7:~$ makeblastdb # 如果安装了的话会弹出用法 USAGE makeblastdb [-h] [-help] [-in input_file] [-input_type type] -dbtype molecule_type [-title database_title] [-parse_seqids] [-hash_index] [-mask_data mask_data_files] [-mask_id mask_algo_ids] [-mask_desc mask_algo_descriptions] [-gi_mask] [-gi_mask_name gi_based_mask_names] [-out database_name] [-blastdb_version version] [-max_file_sz number_of_bytes] [-logfile File_Name] [-taxid TaxID] [-taxid_map TaxIDMapFile] [-version]安装以下环境:java、javac、blast、make。命令如下,根据自己系统的情况选择安装哪一个:
sudo apt update sudo apt upgrade # 这两步比较玄学,但是还是建议升级一下 sudo apt install openjdk-11-jre-headless # java sudo apt install openjdk-11-jdk-headless # javac sudo apt install ncbi-blast+ # blast sudo apt install make # make
-
Zlib库:
建议先去官网看看现在的版本是什么,然后以官网的下载链接为准,这里代码只做参考- 下载并解压:
wget http://www.zlib.net/zlib-1.2.12.tar.gz # 下载Zlib tar -zxvf zlib-1.2.12.tar.gz # 解压 cd zlib-1.2.12/- 安装:
./configure make make install -
libpng库
与Zlib类似,但是libpng依赖于Zlib,所以需要先下载安装Zlib。- 下载并解压:
wget https://cfhcable.dl.sourceforge.net/project/libpng/libpng16/1.6.37/libpng-1.6.37.tar.gz tar -zxvf libpng-1.6.37.tar.gz cd libpng-1.6.37/- 安装:
./configure make make install
下载并安装MCScanX
如上所述,MCScanX官网已经不维护了,但是可以从github上下载。
- 下载:
wget https://codeload.github.com/wyp1125/MCScanX/zip/refs/heads/master unzip master cd MCScanX-master # master解压出来文件名是MCScanX-master - 安装:
由于现在大都是64位系统,所以需要给以下三个文件msa.h,dissect_multiple_alignment.h,and detect_collinear_tandem_arrays.h添加库文件:#include <unistd.h>。我用的是vim:vim msa.h #然后把这三个头问题就修改了 make #然后安装 - 将MCScanX以及他的下游分析添加到环境变量:
- MCScanX
echo 'PATH=$PATH:~/yourPath/MCScanX-master/ ' >> ~/.bashrc #此处的yourPath指你装MCScanX的路径 MCScanX # 这样就可以直接命令行调用MCScanX了- 下游分析程序
export CLASSPATH='.:/yourPath/MCScanX-master/downstream_analyses' # 将绘图等需要的类添加至java的类路径 java dot_plotter -g os_sb.gff -s os_sb.collinearity -c dot.ctl -o dot.PNG # 这样就可以直接调用下游java分析程序了
举例说明
我主要用MCScanX做共线分析并画图,这里用我自己的流程作为示例。其他的我也不太会哈哈
-
处理输入文件:
下载两个物种的
se.pep.fa和so.pep.fa文件,以及其se.gff3和so.gff3文件。但是MCScanX需要的gff文件与标准版的gff3文件有所区别,只有四列,分别是染色体名称、基因名、基因起始坐标、基因结束坐标,所以需要先进行处理。注意:染色体格式为sp_name1。
sp_name gene_name starting_position ending_position # gff文件的格式这是我处理
gff3文件的python代码,大家可以参考:import os import pandas as pd def main(path): gff3_files = [] files = os.listdir(path) for file in files: if file.split('.')[-1] == 'gff3': gff3_files.append(file) x = path for gff3_file in gff3_files: with open(x+gff3_file, "r") as file: temp = pd.read_csv(file, sep='\t',comment='#', header=None) # 只保留基因 temp = temp.drop(temp[temp[2] != 'gene'].index) temp = temp[[0,8,3,4]] temp[8] = temp[8].map(lambda x: x.split(';')[0].split('=')[1]) # 丢掉ups temp = temp.drop(temp[temp[0].str.contains('ups')].index) dataFrame = temp.drop(temp[temp[0].str.contains('unplaced-scaffold')].index) dataFrame = dataFrame.reset_index(drop = True) # 排序并输出 name = dataFrame.at[0, 0] number_index = 0 for j in range(len(name) - 1, -1, -1): if name[j].isdigit(): continue else: number_index = j + 1 break dataFrame[0] = dataFrame[0].map(lambda x: int(x[number_index:])) dataFrame = dataFrame.sort_values(by=[0, 3], ascending=[True, True]) dataFrame[0] = dataFrame[0].map(lambda x: name[:number_index] + str(x)) dataFrame.to_csv(x+gff3_file[:-1], header=None, index=None, sep='\t') # 最终有几个gff3文件,就会生成几个gff文件 # 按间距中的绿色按钮以运行脚本。 if __name__ == '__main__': path = 'C/gff_file_path/' main(path) -
blastp 比对
makeblastdb -in se.pep.fa -dbtype prot -parse_seqids -out se.pep.db # 因为我画的是se和so之间的共线性点图,所以使用se建库,再和so进行比对。 blastp -query so.pep.fa -db se.pep.db -out se_so.blast -evalue 1e-10 -num_threads 12 -outfmt 6 -num_alignments 5 -
运行MCScanX
运行MCScanX之前,我们需要将gff文件、blast文件放入同一个文件夹,且gff文件是两个物种gff文件合并的gff。另外两个文件的名字需要相同,我这里是se_so.gff和se_so.blast。MCScanX se_so得到三个文件:
se_so.html、se_so.collinearity、se_so.tandem。 -
绘制共线性点图
- 控制文件
绘制共线性点图需要dot.ctl文件。文件格式为:
800 800 so1,so2,so3,so4,so5,so6,so7,so8,so9,so10,so11 se22,se21,se20,se19,se18,se17,se16,se15,se14,se13,se12,se11,se10,se9,se8,se7,se6,se5,se4,se3,se2,se1 # 从上往下分别为X轴、Y轴的像素大小,X轴、Y轴染色体顺序(染色体格式需要为物种名+条数,eg:so1 )- 绘图命令:
java dot_plotter -g se_so.gff -s se_so.collinearity -c dot.ctl -o dot.PNG
- 控制文件