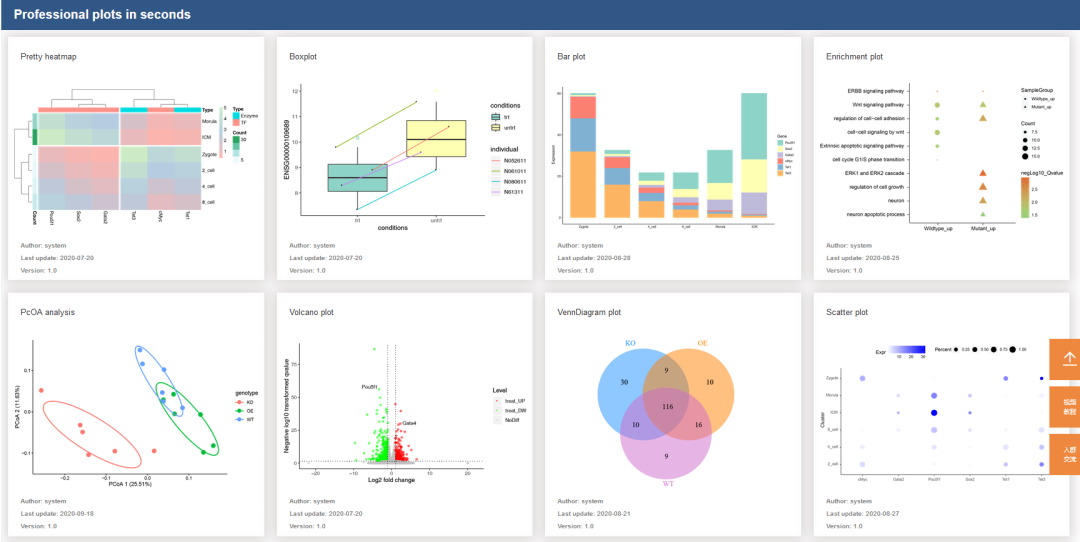
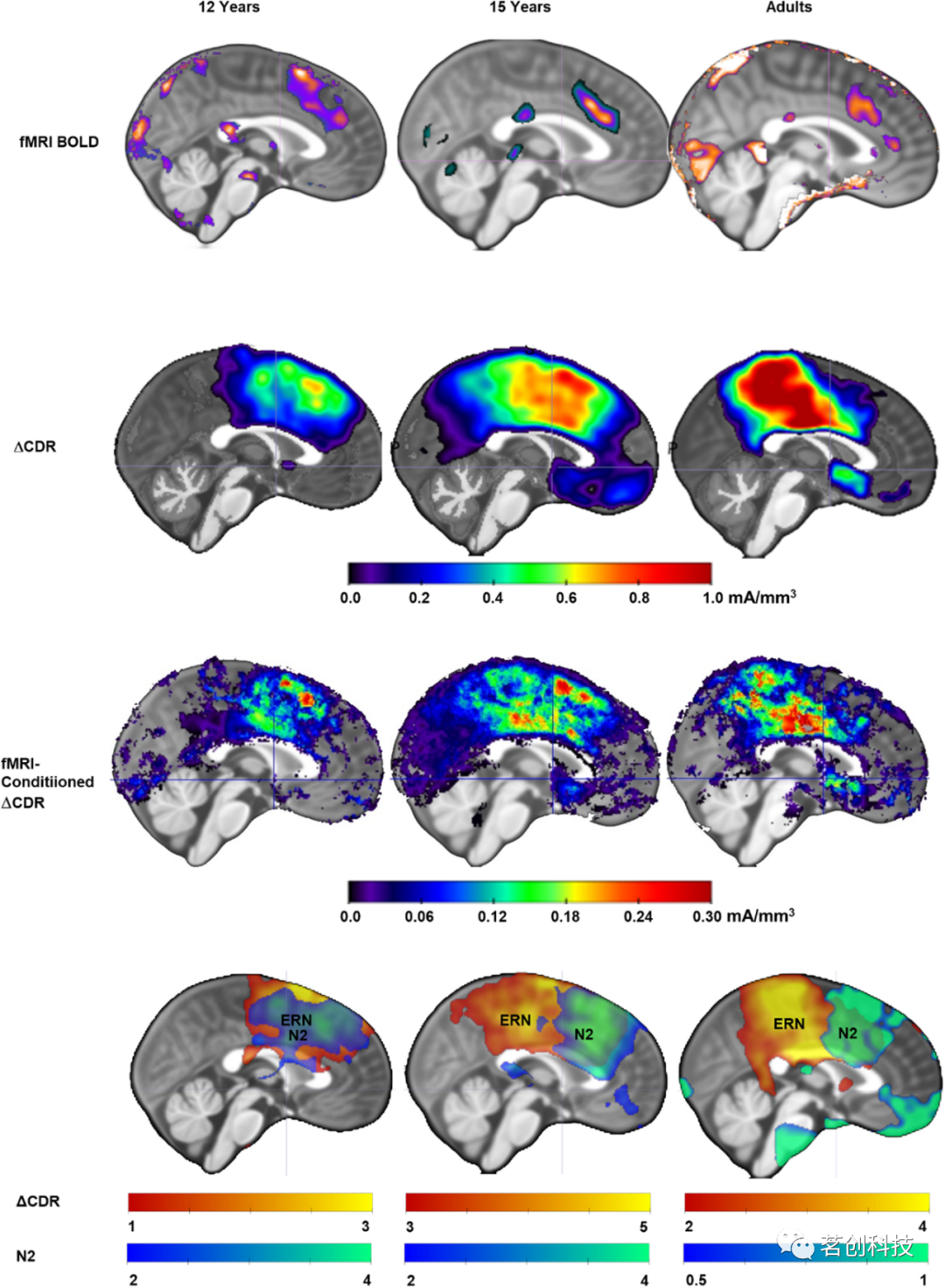
在癌症患者中,药物对肠道细菌群的影响很难确定。以往的研究主要关注抗生素对肠道微生物组的影响,而对非抗生素药物的影响在人体中了解较少,这是由于缺乏可靠的药物暴露数据和长期粪便样本的收集。
2023年6月8日发表于《Cell》的“High-resolution analyses of associations between medications, microbiome, and mortality in cancer patients”论文通过开发和应用一种新的计算方法PARADIGM(与肠道微生物组动态相关的参数)对大量癌症患者进行了长期粪便微生物组分析,并详细记录了药物使用情况,揭示了药物与微生物组之间的关联。

PARADIGM算法是一种基于微生物动力学数学模型,通过弹性网络正则化回归推断药物对聚类动态的影响,计算药物的细菌响应分数。PARADIGM可以推断药物、微生物组成和临床结果之间的关系,预测生物学与临床的相关关联信息,进而给出药物暴露是如何扰乱或保留微生物组成。
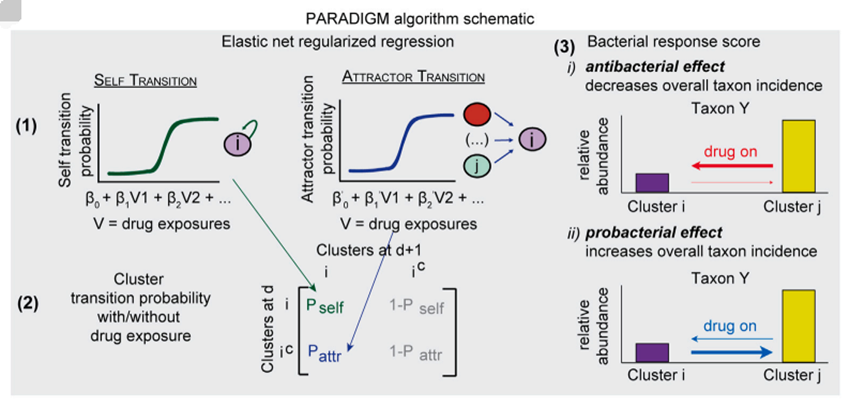
PARADIGM算法示意图
研究重点关注接受同源异体造血干细胞移植(Allo-HCT)的患者,并研究了抗生素和非抗生素药物对肠道微生物群的影响。研究人员发现,某些非抗生素药物(如泻药、止吐药和鸦片类药物)与微生物组成的改变相关,包括Enterococcus的增多和α多样性的减少。研究还证明了Allo-HCT过程中的微生物亚种竞争和遗传多样性收敛受抗生素暴露的显著影响。研究人员将药物-微生物组关联整合到两个验证队列中,结果表明这种方法可以为药物暴露如何影响微生物组成提供有价值的指导信息。
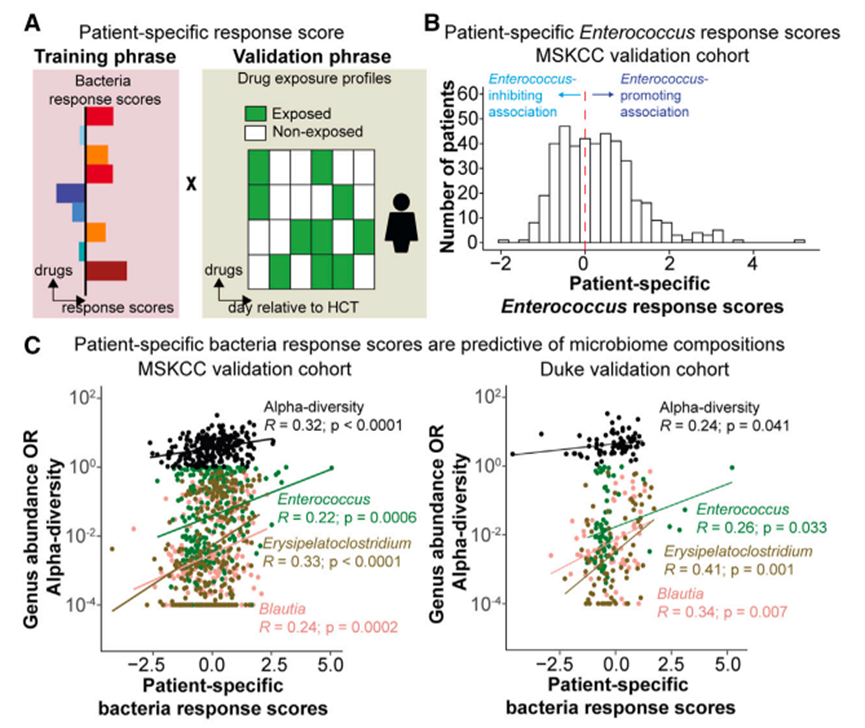
(A) 患者特异性细菌反应评分计算的示意图。(B) 在仅基于药物暴露记录的验证队列中,患者特异性的肠球菌反应评分(相对于移植前14天到14天之间的时间段)和图3C中呈现的细菌反应评分。负分表示药物暴露记录与抑制肠球菌的效应相关,而正分表示药物暴露记录与促进肠球菌的效应相关。(C) MSKCC验证队列(423名患者)和杜克队列(142名患者)中,相对于移植后的14至45天内收集的样本中,患者特异性细菌反应评分与观察到的属相对丰度或α多样性之间的Pearson相关性。p 值经过Benjamini-Hochberg校正。
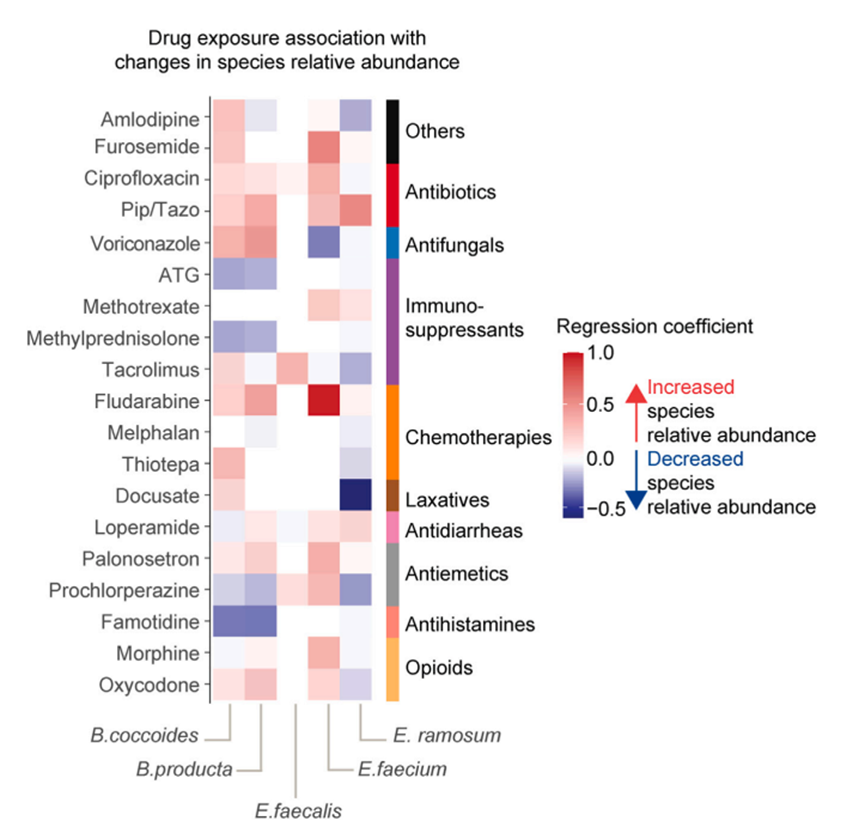
药物暴露与宏基因组测序得出的物种相对丰度变化之间的关联

在癌症患者中分辨药物暴露对肠道菌群的影响是具有挑战性的。在这里,我们通过开发和应用一种新的计算方法PARADIGM (与肠道微生物群动态相关的参数)来解析药物暴露与微生物组成的变化之间的关系。我们使用了一组包含详细用药记录的大规模纵向粪便微生物组分析数据,这些数据来自接受异体造血干细胞移植的患者。我们观察到几种非抗生素药物(包括泻药、止吐药和鸦片类药物)与肠球菌相对丰度增加和α多样性减少相关。二代测序进一步证明了亚种竞争导致Allo-HCT期间优势菌株遗传融合增加,这与抗生素暴露显著相关。我们将药物-微生物组关联整合到药物暴露数据中,仅基于药物暴露就可以预测两个验证队列中的临床结果,这表明这种方法可以生成与生物学和临床相关的见解,了解药物暴露如何扰乱或保护微生物组成。
应用一种称为PARADIGM的计算方法对大量癌症患者的纵向粪便标本和详细的日常药物记录进行分析,揭示了药物暴露与肠道微生物之间的关联。这些关联不仅重现了体外实验结果,还能预测临床结果。
阅读原文内容:
https://www.cell.com/cell/fulltext/S0092-8674(23)00526-3
往期精品(点击图片直达文字对应教程)
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
机器学习