,
桓峰基因公众号推出单细胞生信分析教程并配有视频在线教程,目前整理出来的相关教程目录如下:
Topic 6. 克隆进化之 Canopy
Topic 7. 克隆进化之 Cardelino
Topic 8. 克隆进化之 RobustClone
SCS【1】今天开启单细胞之旅,述说单细胞测序的前世今生
SCS【2】单细胞转录组 之 cellranger
SCS【3】单细胞转录组数据 GEO 下载及读取
SCS【4】单细胞转录组数据可视化分析 (Seurat 4.0)
SCS【5】单细胞转录组数据可视化分析 (scater)
SCS【6】单细胞转录组之细胞类型自动注释 (SingleR)
SCS【7】单细胞转录组之轨迹分析 (Monocle 3) 聚类、分类和计数细胞
SCS【8】单细胞转录组之筛选标记基因 (Monocle 3)
SCS【9】单细胞转录组之构建细胞轨迹 (Monocle 3)
SCS【10】单细胞转录组之差异表达分析 (Monocle 3)
SCS【11】单细胞ATAC-seq 可视化分析 (Cicero)
SCS【12】单细胞转录组之评估不同单细胞亚群的分化潜能 (Cytotrace)
SCS【13】单细胞转录组之识别细胞对“基因集”的响应 (AUCell)
SCS【14】单细胞调节网络推理和聚类 (SCENIC)
SCS【15】细胞交互:受体-配体及其相互作用的细胞通讯数据库 (CellPhoneDB)
SCS【16】从肿瘤单细胞RNA-Seq数据中推断拷贝数变化 (inferCNV)
SCS【17】从单细胞转录组推断肿瘤的CNV和亚克隆 (copyKAT)
SCS【18】细胞交互:受体-配体及其相互作用的细胞通讯数据库 (iTALK)
SCS【19】单细胞自动注释细胞类型 (Symphony)
SCS【20】单细胞数据估计组织中细胞类型(Music)
SCS【21】单细胞空间转录组可视化 (Seurat V5)
SCS【22】单细胞转录组之 RNA 速度估计 (Velocyto.R)
SCS【23】单细胞转录组之数据整合 (Harmony)
SCS【24】单细胞数据量化代谢的计算方法 (scMetabolism)
SCS【25】单细胞细胞间通信第一部分细胞通讯可视化(CellChat)
SCS【26】单细胞细胞间通信第二部分通信网络的系统分析(CellChat)
SCS【27】单细胞转录组之识别标记基因 (scran)
桓峰基因的教程不但教您怎么使用,还会定期分析一些相关的文章,学会教程只是基础,但是如果把分析结果整合到文章里面才是目的,觉得我们这些教程还不错,并且您按照我们的教程分析出来不错的结果发了文章记得告知我们,并在文章中感谢一下我们哦!
公司英文名称:Kyoho Gene Technology (Beijing) Co.,Ltd.这期分享一篇2020年10月份发表在 cell (IF:25+),作者基于单细胞 RNA-seq 揭示肝实质和非实质细胞系的早期发育。该文章使用桓峰基因公众号里面生信分享教程即可实现,有需要类似思路的老师可以联系我们!
摘 要
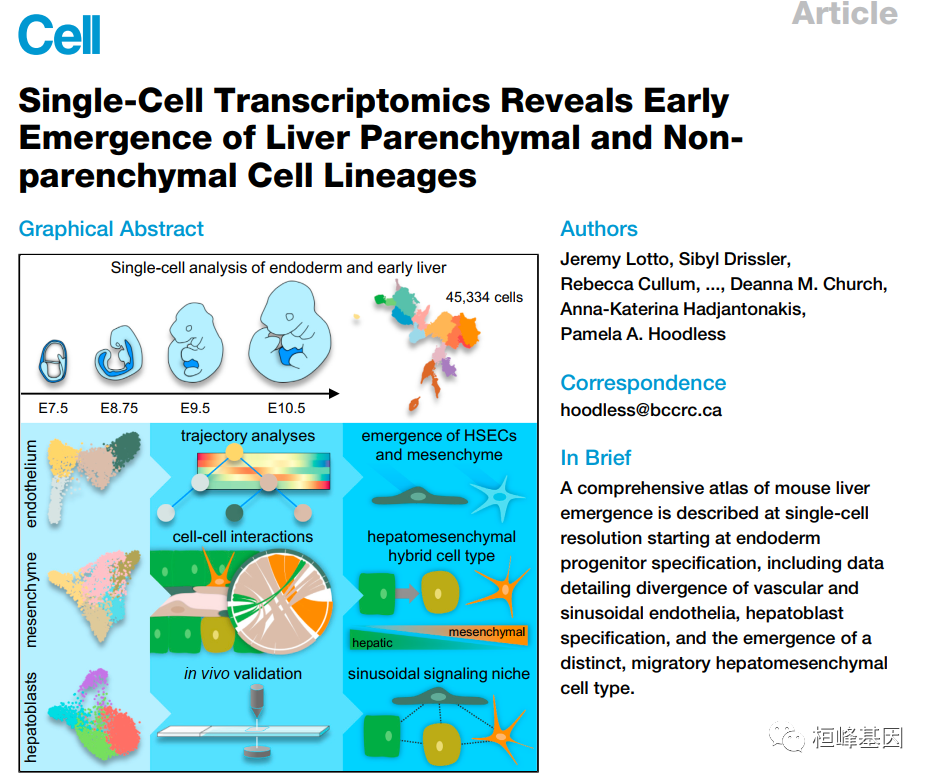
早期肝脏的细胞复杂性和规模限制了对其在器官发生过程中出现的分析。为了避免这些问题,我们分析了45,334个单细胞转录组,从胚胎日(E)7.5,当内胚层祖细胞被指定,到E10.5肝脏,当肝实质和非实质细胞系出现。我们的数据详细描述了血管内皮和窦内皮的差异,包括E8.75对窦内皮规范的独特转录谱。我们描述了两种不同的间皮细胞类型以及早期肝星状细胞,并揭示了这些群体的不同时空分布。捕获了肝母细胞特异性和迁移的转录谱,包括肝间充质细胞类型的出现和肝母细胞集体细胞迁移的证据。此外,我们在原始正弦信号的组织过程中确定了细胞间的相互作用。这项研究在单细胞分辨率上提供了从内胚层和中胚层到原始窦的肝脏谱系建立的全面图谱。
生信分析流程
相关数据集选择:
a)E7.5 and E8.75 scRNA-seq data (GEO: GSE123046)
b)Gastrulation scRNA-seq data (GEO: GSE87038)
c)E9.5 and E10.5 scRNA-seq data (ArrayExpress: E-MTAB-9334)
d)自测样本数据
e)代谢组测序结果
基因集选择:
通过单细胞数据集筛选非肝实质细胞基因并作为初筛基因,同代谢组差异基因进行筛选,最后确定标记基因
生信分析方法:
我们从文章的分析流程中提取所有的分析内容,整理出来就 15个分析条目,每个条目都包括分析的内容,这些分析构成了整个文章,本文属于生信分析的文章,下面我们就看看哪些分析可以利用桓峰基因公众号的教程来实现,点击分析条码就会跳转到对应公众号的教程,跟着教程做,您也能发轻松发高分,如下:
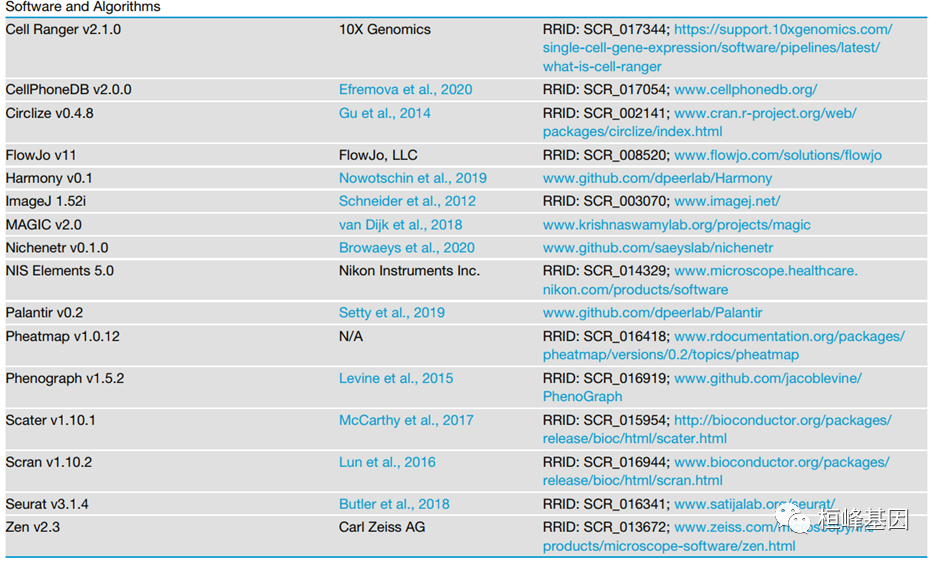
1. 10X Genomics 测序数据分析 (Cell Ranger)
2. 数据处理(Seurat)
a) 数据合并,将已测数据与文章发表的数据合并;
b) 数据过滤,归一化,降维,聚类分析;
3. 细胞类型注释 (scran)
4. 细胞间交互分析 (CellPhoneDB,Nichenetr)
5. 热图绘制(pheatmap)
6. 圈图绘制(Circlize)
文章列出来的涉及到的软件列表,从列表中可以看出来该文章虽然发在 Cell 上,但是整个文章并没有出现新的算法,思路也是中规中矩,所以学会基本的单细胞分析的各种方法,把图表做得清晰明了,颜色搭配好,还是能够发高分的,从表格上看,这里面生信分析使用的所有软件在桓峰基因公众号上都有相应的教程,欢迎大家点击免费教程进行学习,如果觉得难道过大,那就联系我们吧!
研究结果
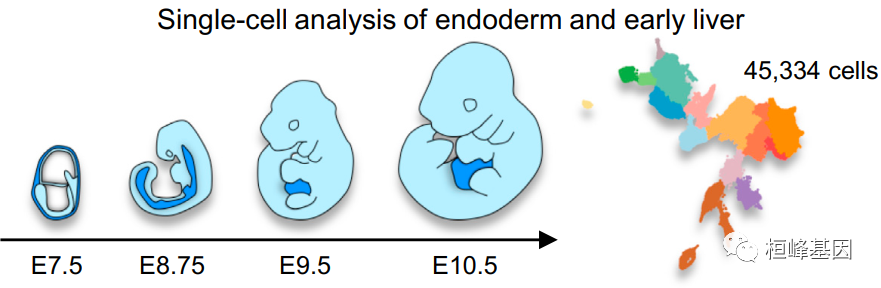
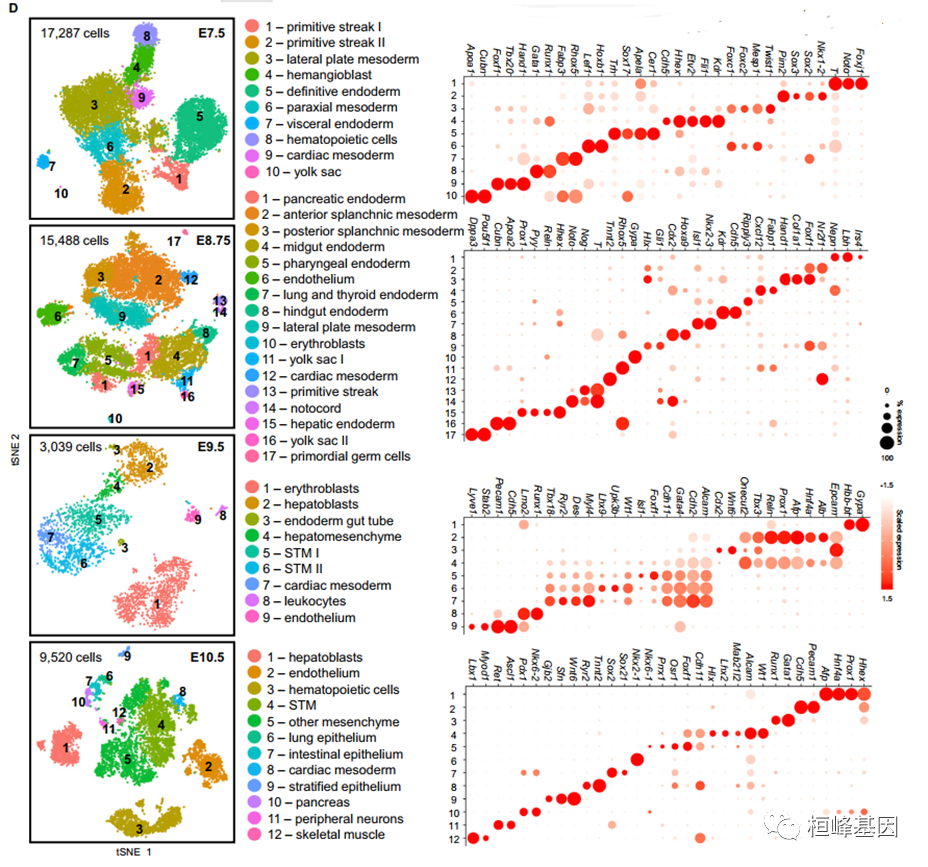
1. 利用scRNA-seq追踪突发性实质肝和非实质肝谱系
(C)所有测序细胞的UMAP可视化(Seurat);
(D)发育阶段的细胞类型组成(条形图);
(E)热图描述了所识别的所有细胞类型的差异表达基因(pheatmap)。
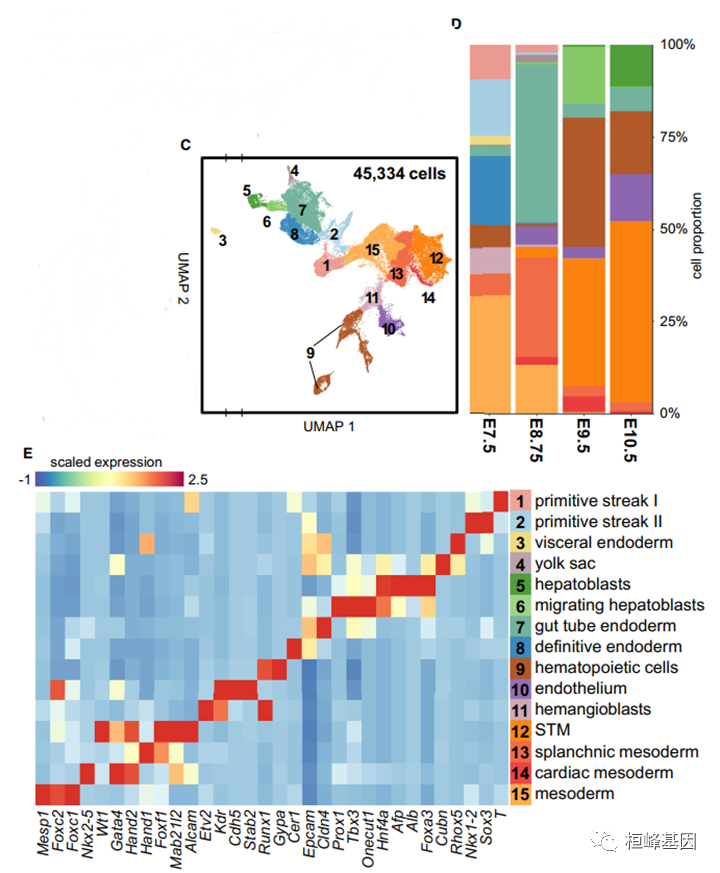
另见图S1单细胞细胞类型注释
(B) UMAP所有重复序列的可视化(Seurat)和质量控制(小提琴图);
(C)热图和umap显示相互最近邻(MNN)批量校正本文中提供的数据与Pijuan-Sala等人(2019)从整个胚胎中收集的数据之间的差异。来自两个手稿的注释簇之间的一致验证了注释策略;
(D)聚类(Seurat)和差异基因表达分析在E7.5, E8.75, E9.5处鉴定出10,17,9和12个聚类和E10.5点状图突出选择的顶级差异表达基因(气泡图)。
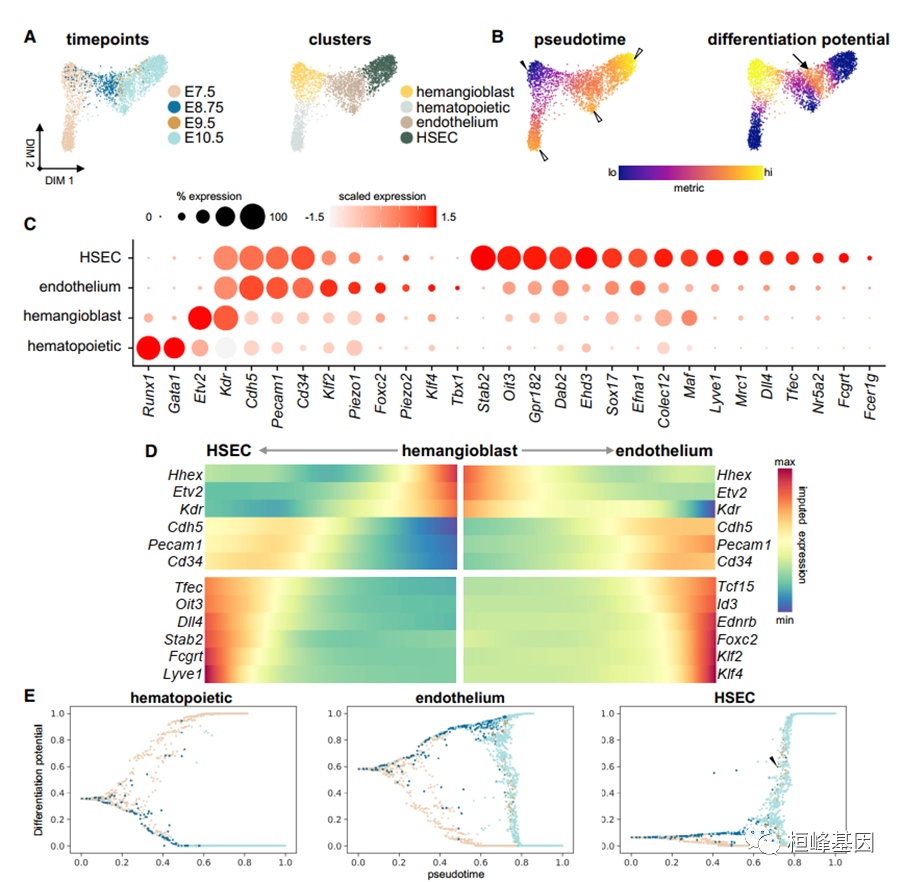
2. 窦内皮和血管内皮通过E8.75从一个共同的内皮祖细胞分化
(A)力定向布局显示时间点和内皮细胞和E7.5造血祖细胞的聚类贡献(n = 2,569)。
(B)力定向布局显示内皮细胞系从Etv2hi“起始细胞”(实箭头)到终点状态(空心箭头)的伪时间和分化潜力。箭头表示第二局部分化电位最大值,代表血管内皮的HSEC规格。(C)显示已知造血、成血管细胞和内皮细胞标记的点图,包括未成熟和成熟HSEC标记的表达。圆点的大小表示表达每种基因的种群比例。颜色表示表情的程度。
(D)热图表示从成血管细胞“起始细胞”到HSEC和内皮终端状态的伪时间基因趋势。
(E)造血、内皮细胞和HSEC轨迹的分化潜能作为伪时间的函数,其中每个点代表一个细胞,如(a)所示,由胚胎时间点彩色编码。箭头表示E8.75、E9.5和E10.5处的内皮细胞,它们被指定为HSEC终点状态。肝窦内皮细胞。
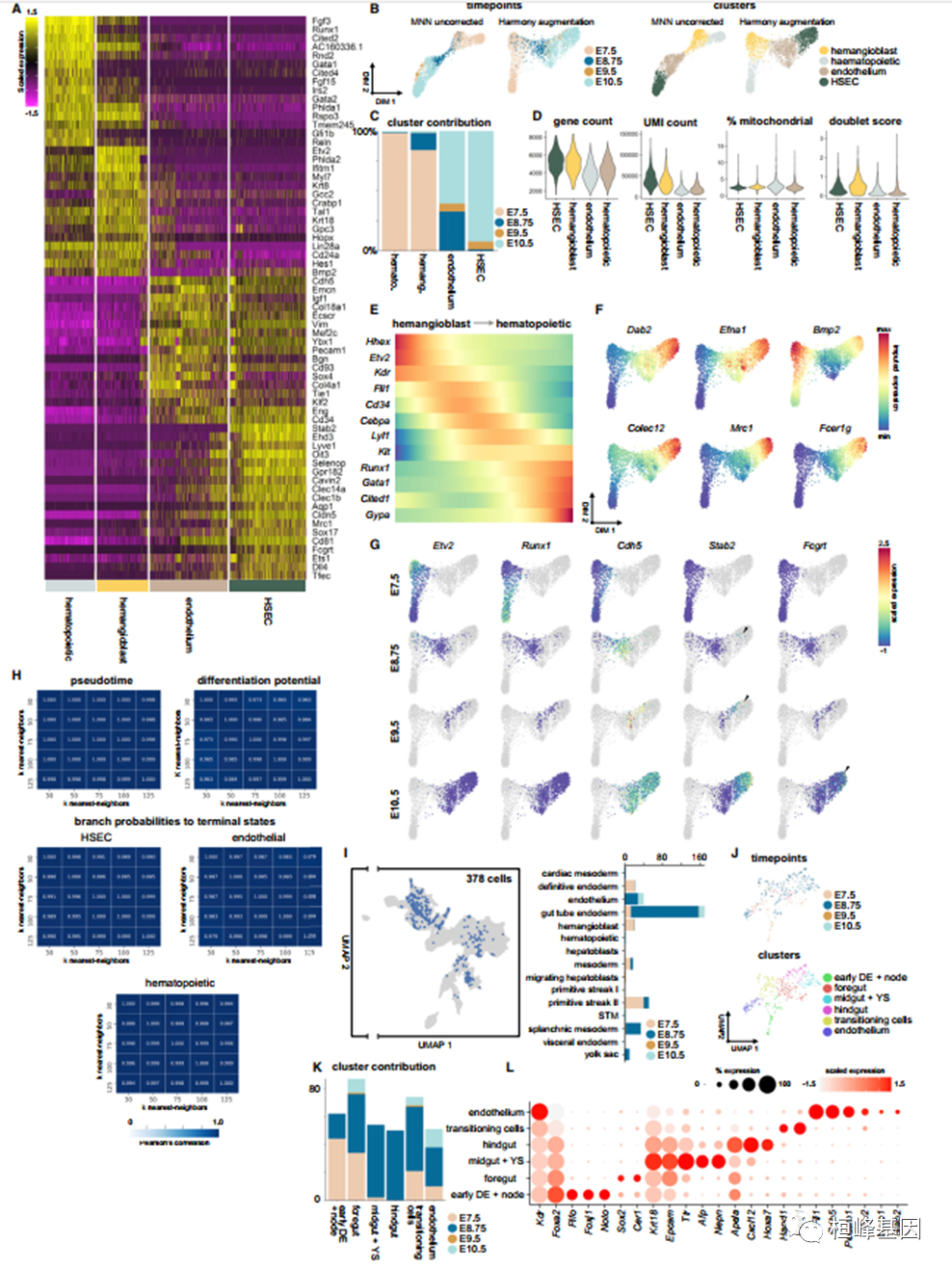
另见图S2从E7.5到E10.5捕获的内皮细胞谱系特征
(A)热图突出选择顶级差异表达基因。
(B)显示时间点和内皮细胞谱系簇分布的力向分布图和谐矫正前后。
(C)胚胎时间点在各簇间的分布,显示内皮谱系出现的时间。
(D)质量内皮细胞谱系的对照参数,按聚类排序。
(E)热图表示从Etv2hi成血管细胞“开始细胞”到伪时间的基因趋势
造血终端状态。
(F)显示HSEC集群中富集的选择基因表达的力定向布局。
(G)力定向布局,以时间点分隔,显示与内皮和造血谱系相关的基因的规范化表达。箭头表示细胞与早期HSEC标记物Stab2和Fcgrt的表达。
(H) Palantir结果是稳健的,当变化的最近邻居输入,k伪时间、分化势和成对Palantir运行之间的分支概率。
(I) Foxa2-Kdr共表达细胞的分布(>1 logcounts)在按集群完成UMAP。
(J) Foxa2-Kdr共表达细胞的重新聚类揭示了明确的内胚层和内皮细胞状态移行细胞群。
(K) Foxa2-Kdr共表达子集中胚胎时间点在聚类中的分布。
(L)显示转录的点图Foxa2-Kdr共表达子集内簇间的异质性
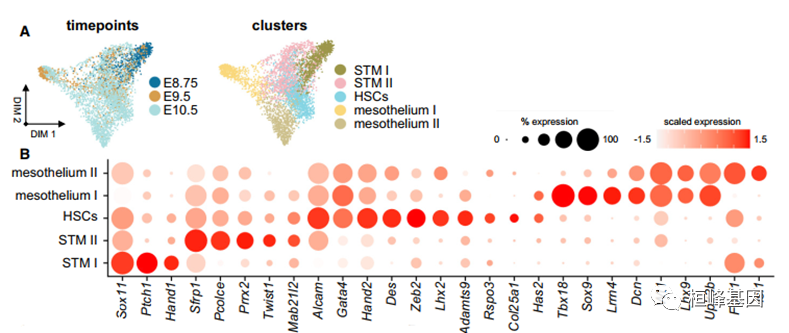
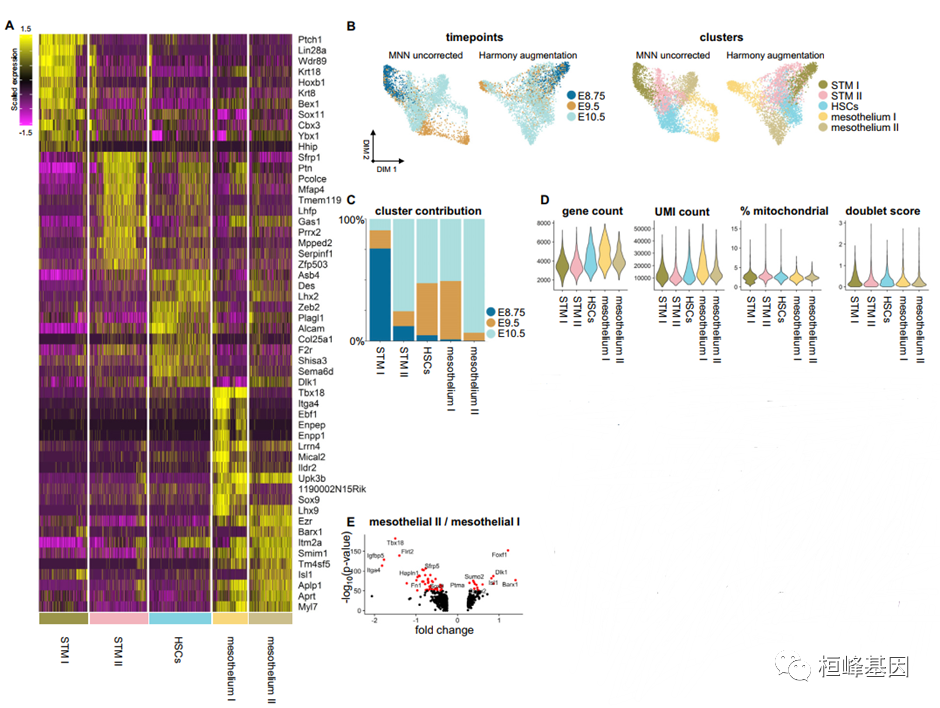
3. 早期肝脏中隔和肝间充质异质性
(A)力定向布局显示时间点和间充质祖细胞的聚类贡献(n = 3,691)。
(B)点状图显示已知和以前未鉴定的STM、间皮和HSC鉴定标记。圆点的大小表示表达每种基因的种群比例。颜色表示表情的程度。
另见图S3从E8.75到E10.5捕获的间充质谱系的特征
(A)热图突出选择顶级差异表达基因。
(B)显示Harmony校正前后间充质系时间点和簇分布的力向分布图。
(C)各簇间时间点分布,显示间充质谱系出现的时间。
(D)间充质系的质量控制参数,按聚类排序。
(E)火山图显示已鉴定的间皮簇之间的顶级差异表达基因。
4. 早期肝脏中存在一种肝间充质杂交祖细胞
(A)力向布局显示时间点和肝实质祖细胞的聚类贡献(n = 2,332)。
(B)点状图显示内胚层、迁移性肝母细胞、肝母细胞和肝间充质细胞类型的已知和以前未鉴定的标记。圆点的大小表示表达每种基因的种群比例。颜色表示表情的程度。
(C)热图和力定向布局显示捕获的所有早期肝实质细胞的平均肝、间充质和上皮基因图谱。
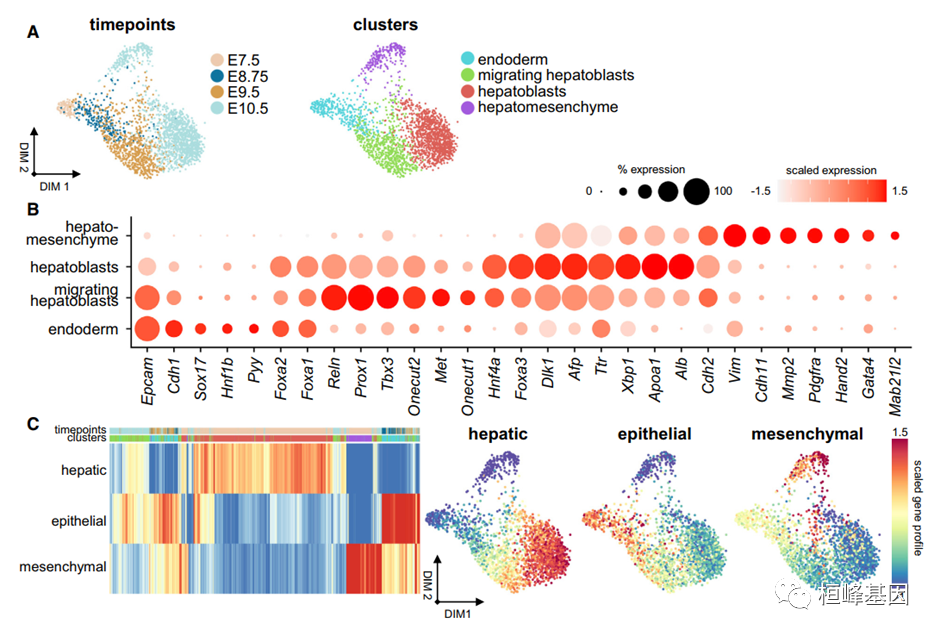
热图列根据时间点和聚类配色方案(A)进行标注,参见图S4
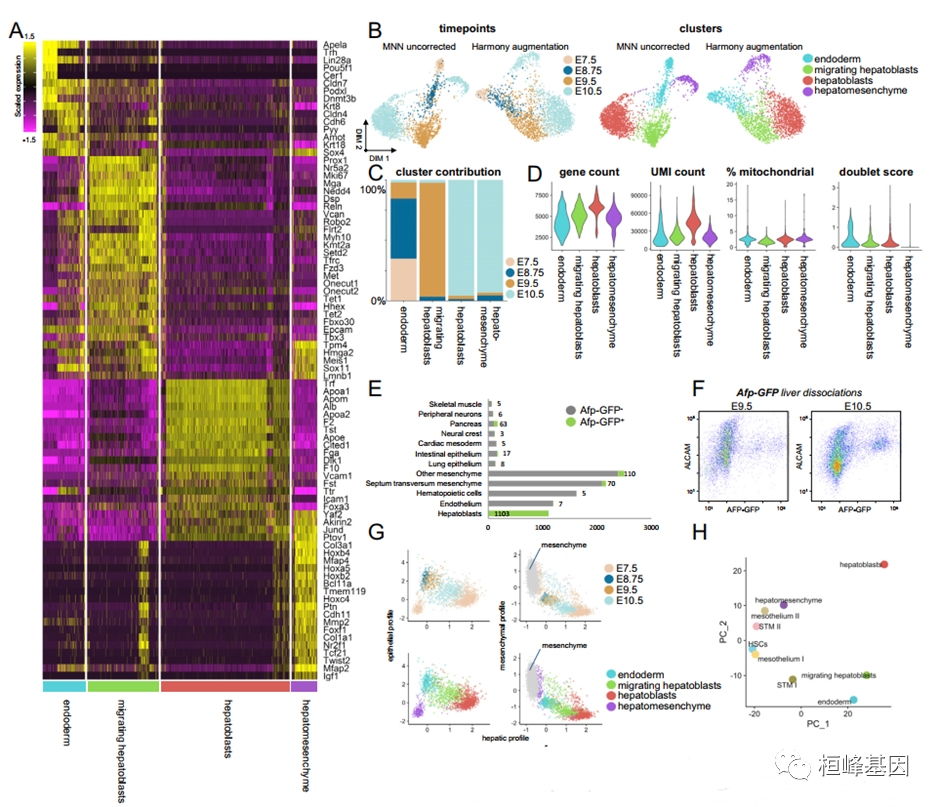
(A)热图突出选择顶级差异表达基因。
(B)显示内胚层和内胚层的时间点和簇分布的力向布局和谐校正前后的肝谱系。
(C)胚胎时间点在集群中的分布,显示肝脏谱系出现的时间。
(D)肝谱系的质量控制参数,按聚类排序。
(E) E10.5处测序的AFP-GFP+和AFP-GFP-细胞分布,显示AFP-GFP+肝实质衍生物对间充质簇的贡献。
(F)在E9.5和E10.5的离体AFP-GFP肝脏中显示AFP-GFP与stm富集标记物ALCAM的FACS图。可见E10.5处GFP+ALCAMhi细胞富集。
(G)比较单细胞肝细胞、上皮细胞和按时间点或聚类划分的间充质谱。间充质系包括间充质谱比较作为参考点。
(H)校长实质和间质细胞簇平均表达谱的成分分析显示,肝间质与间皮II和STM II簇密切共聚,提示转录平行。
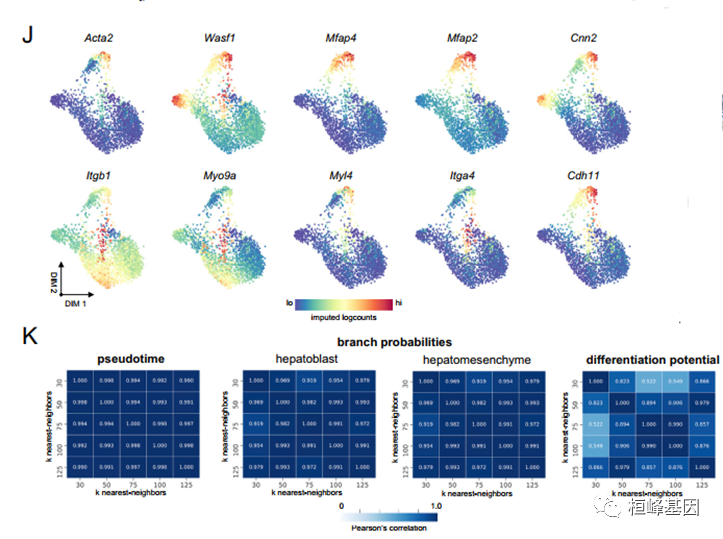
(J)显示肝间充质细胞内富集的选择基因表达的力定向布局。
(K) Palantir结果在变化时是稳健的最近邻输入,k。图显示皮尔森的相关性伪时间,分化势,和分支概率之间的对Palantir运行
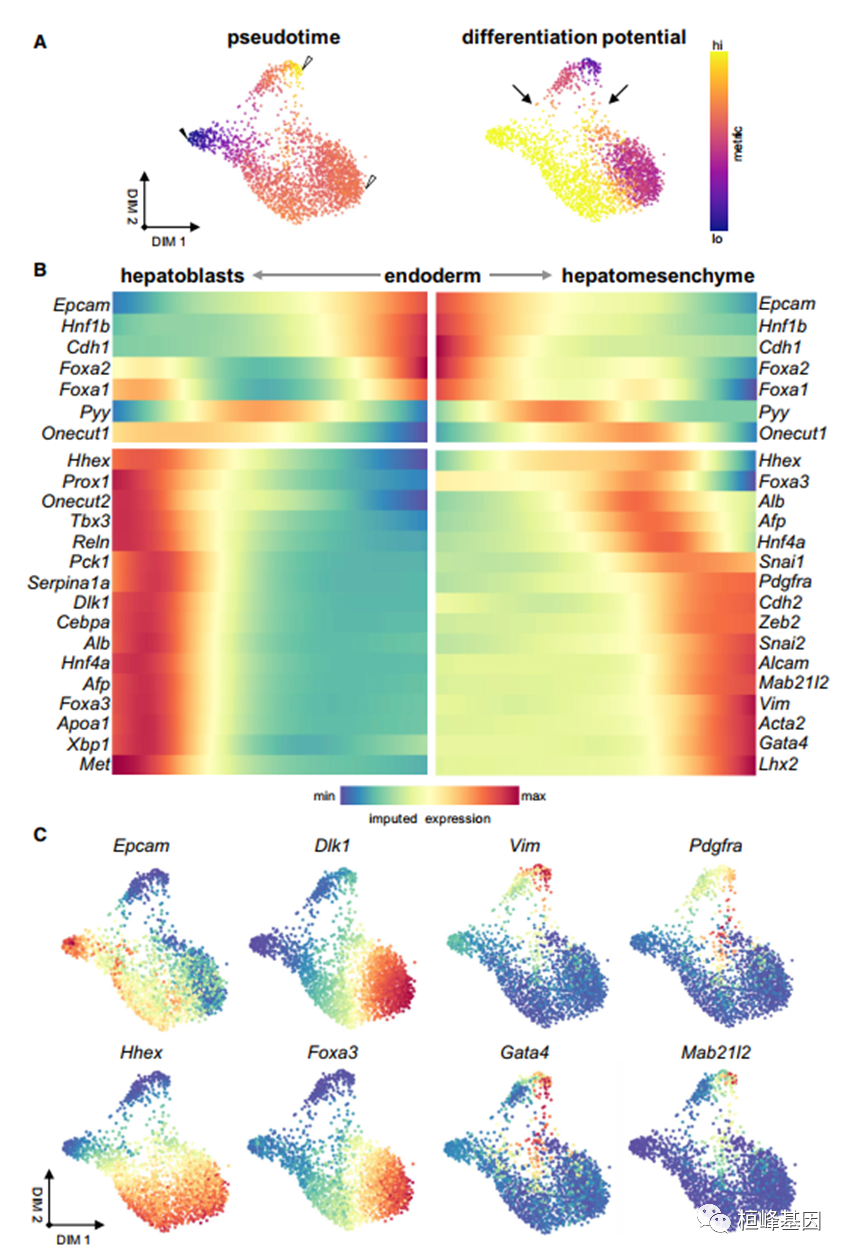
5. 肝母细胞和肝间充质细胞通过独特的迁移机制出现
(A)力定向布局显示肝实质谱系从Alblo“起始细胞”(实箭头)到终点状态(空心箭头)的伪时间和分化潜力。箭头指示达到肝间质终点状态的两个潜在轨迹。
(B)热图表示从内胚层“起始细胞”到肝母细胞和肝间充质细胞末端状态假性时间内的基因趋势。
(C)显示已识别的细胞类型标记的表达水平的力定向布局。
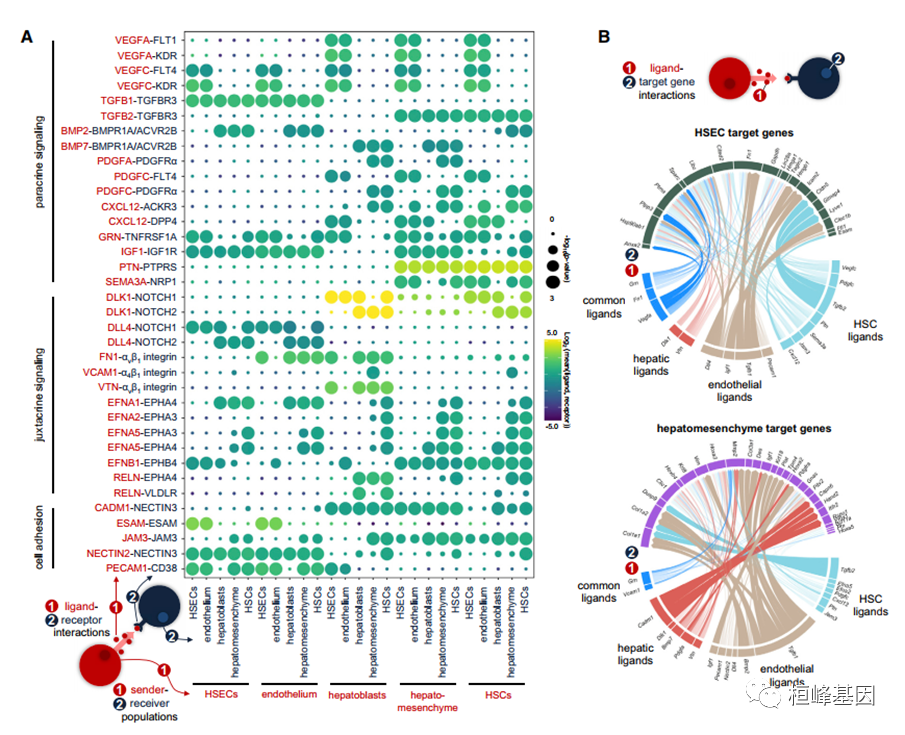
6. 原始正弦信号的信号位
(A)点图显示E10.5捕获的肝母细胞、HSEC、内皮细胞、肝间充质细胞和HSC谱系之间假定的配体-受体相互作用。圆点的大小表示所示相互作用的统计显著性。颜色表示来自簇1的配体和来自簇2的受体的平均表达水平的平均值。
(B)圆圈图描述(1)配体和(2)它们对HSECs和肝间充质细胞的预测靶基因之间的联系。链接的颜色表示配体发送体的数量,而链接的宽度和不透明度分别与配体-受体相互作用的权重和配体活性分数相关。
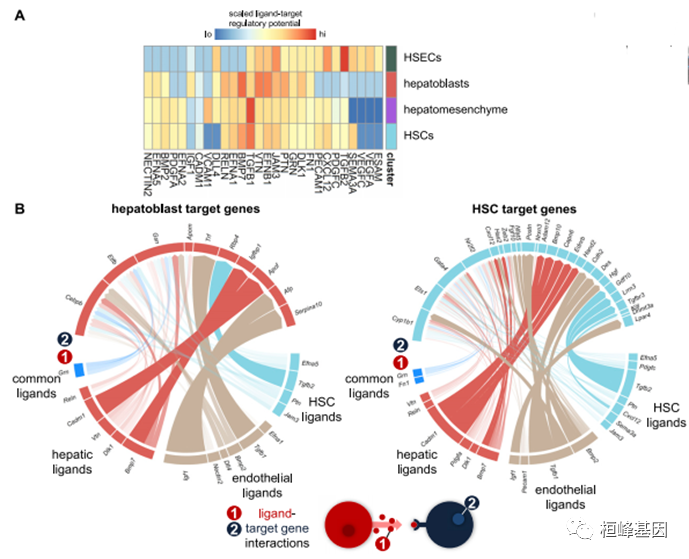
另见图S5 E10.5原始正弦波内的细胞-细胞信号
(A)热图显示了图6中突出显示的配体的标度配体-靶调控潜力。
(B)圆形图描述了(1)配体和(2)它们对肝母细胞和星状细胞的预测靶基因之间的联系。链接的颜色表示配体发送体的数量,而链接的宽度和不透明度分别与配体受体相互作用的权重和配体活性分数相关。
这篇文章利用桓峰基因公众号上的教程完全可以实现这些图表的绘制,过程需要自己细致的探究,毕竟不是很简单的分析,能发到 Cell 也需要有一定的生信分析和结果解读的能力,有需要此类分析的老师可以联系桓峰基因!
桓峰基因,铸造成功的您!
未来桓峰基因公众号将不间断的推出单细胞系列生信分析教程,
敬请期待!!
桓峰基因官网正式上线,请大家多多关注,还有很多不足之处,大家多多指正!
http://www.kyohogene.com/
桓峰基因和投必得合作,文章润色优惠85折,需要文章润色的老师可以直接到网站输入领取桓峰基因专属优惠券码:KYOHOGENE,然后上传,付款时选择桓峰基因优惠券即可享受85折优惠哦!https://www.topeditsci.com/
Reference
1.Lotto J, Drissler S, Cullum R, et al. Single-Cell Transcriptomics Reveals Early Emergence of Liver Parenchymal and Non-parenchymal Cell Lineages. Cell. 2020;183(3):702-716.e14.
2.Nowotschin S, Setty M, Kuo YY, et al. The emergent landscape of the mouse gut endoderm at single-cell resolution. Nature. 2019;569(7756):361-367. doi:10.1038/s41586-019-1127-1
3.Dong J, Hu Y, Fan X, et al. Single-cell RNA-seq analysis unveils a prevalent epithelial/mesenchymal hybrid state during mouse organogenesis. Genome Biol. 2018;19(1):31. Published 2018 Mar 14. doi:10.1186/s13059-018-1416-2
4.Pijuan-Sala B, Griffiths JA, Guibentif C, et al. A single-cell molecular map of mouse gastrulation and early organogenesis. Nature. 2019;566(7745):490-495. doi:10.1038/s41586-019-0933-9
5.Su Y, Chen D, Yuan D, et al. Multi-Omics Resolves a Sharp Disease-State Shift between Mild and Moderate COVID-19. Cell. 2020;183(6):1479-1495.e20. doi:10.1016/j.cell.2020.10.037