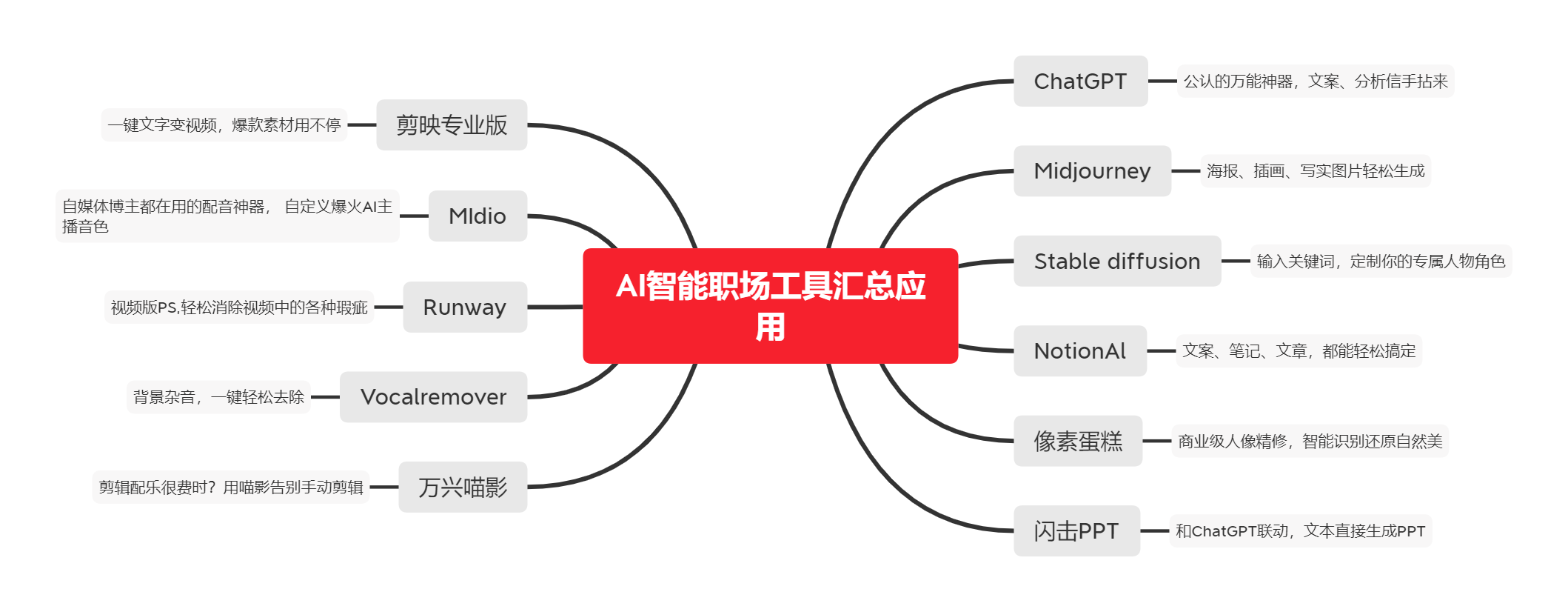
参考教程:
Etomo Tuturial for IMOD version 4.11
1. Initial Setup
- 本教程提供了一个小双轴示例数据集和Etomo的分布指南,更详细的内容参考Tomography Guide。该版本使用1k*1k的图像而不是压缩版本。
imodhelp命令可以打开帮助界面,查看各种教程。
本教程数据集下载:
https://bio3d.colorado.edu/imod/files/tutorialData-1K.tar.gz
然后进入到数据集路径,解压缩:
imoduntar tutorialData-1K.tar.gz
文件夹里有BBa.st和BBb.st两个原始的stacks(教程里写还有个finalFiles文件夹,但我下载是没有的)。
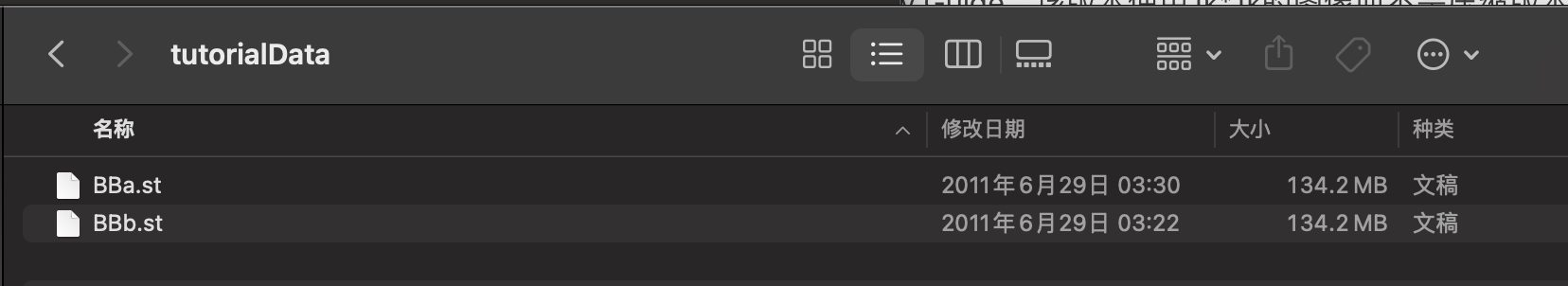
文件名反映了一个约定,原始堆栈有一个扩展名 .st,或者一个表示图像文件格式的扩展名,可以是
.tif、.mrc或.hdf。双轴堆栈有一个公共的根名称,以a和b结尾,后面跟着一个允许的扩展。.st扩展名是一种约定,并不表示图像文件的格式。
2. Etomo Setup and Viewing the Raw Tilt Series
打开 ETomo:
etomo

处理新数据集:Build Tomogram
打开以前的项目:File->Recent Projects
build tomogram后,会有Setup Tomogram面板。选择Raw Image Stack的所在路径;选择Data Type是单轴还是双轴,single frame还是Montage;Templates选择plasticSection.adoc。然后填上 Pixel size,Fiducial diameter, Image rotation,Scan Header可以自动从源文件头里得到这些信息。
本教程不涉及并行处理。
也没有涉及Image distortion field file和Mag gradient corrections。
然后指定一个或两个轴的倾斜角度。在这个例子中,倾斜角度存储在extended header中,因此应该使用默认的从数据中提取倾斜角度Extract tilt angles from data。
如果您正在重建一个从0度开始在两个方向上进行的倾斜序列,则可以将series设置为双向,这将负责设置一些适当的对齐参数。还可以选择指定从处理步骤中排除的单个投影。这个排除列表的语法是用逗号分隔的范围列表(例如1,4-5,60-70)。注意,在这个部分中还有一个按钮可以打开原始数据文件
View Raw Image Stack,以便在3dmod中查看。查看原始的tilt系列文件有助于决定预处理步骤,或者查看是否有任何特定的视图具有较差的图像质量,以及希望从对齐和重建中排除哪些视图。
要确定是否有图像质量差(聚焦差等)的视图,请按View Raw Image Stack按钮打开原始图像堆栈。通过点击鼠标中键查看原始的倾斜系列图像。注意图像是如何轻微地跳跃的。请注意您希望从对齐和重建中排除的任何特定视图。在这个样本数据集中没有需要排除的图像。
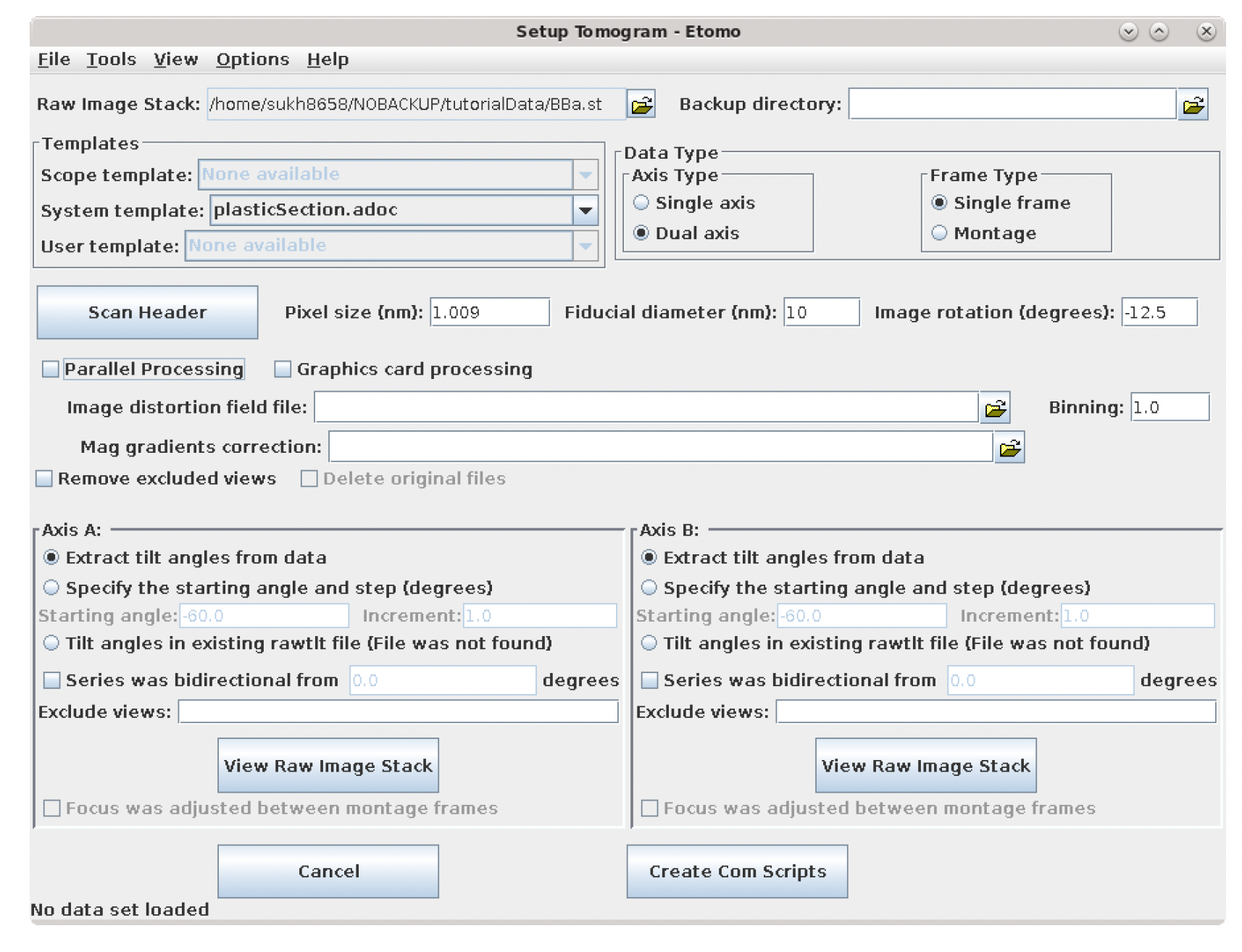
按下Create Com Scripts创建Com脚本按钮继续生成断层图。
3. Etomo Main Window
如果需要在完成本教程之前退出Etomo,可以进入tutorialData目录(该目录现在包含您的数据集),然后输入: etomo BB.edf,继续工作。
进程控制面板如下图。按钮按照建议的处理顺序从上到下排列。这些按钮用颜色编码来表示过程的阶段。

5. Pre-processing (a Detour to Axis B)
如果使用CCD相机在显微镜上收集图像,在收集初始暗参考或单个图像时,随机的X射线击中CCD相机可能会导致数据文件中出现极高或极低的像素值。因此,这些极端值会破坏对比度并可能在重建过程中引起伪影。CMOS相机也会产生类似的效果,尽管如果相机在每次曝光时多次读出,它们的效果不会像CCD相机那样极端;它们还有一个产生极值的“热像素”的倾向。本教程中的示例没有非常极端的像素值,不会导致严重问题;实际上,对于第一个轴,不需要进行任何预处理。因此,我们将跳过到第二个轴,以说明评估极端值的程度并在必要时去除它们的过程。
要查看B轴,请在Etomo窗口顶部按下Axis B按钮。这将在左侧显示另一组处理按钮,可用于执行对齐B轴倾斜系列和计算层析图的操作。为了避免混淆,A轴具有蓝色背景,而B轴具有绿色背景。
点击按钮 Pre-processing。按下Show Min/Max for Raw Stack按钮运行clip stats程序,该程序会显示原始堆栈每个部分的最小和最大密度。窗口将打开显示文本输出以及这些密度的图表。如果最小密度值是一个负数或零,那么你的数据集中可能存在一个极端的黑色像素,这是由于在采集暗参考图像期间的x射线事件造成的。如果某些部分具有较高的最大密度,则数据集中存在极端的白色像素。在这种情况下,存在一个明显的异常值和一些其他突出的极大值,尽管后者的差异很小。按下Create Fixed Stack按钮创建一个具有去除X射线的第二个堆栈。按下View Fixed Stack按钮查看该堆栈,并按下Show Min/Max for Fixed Stack按钮在修复的堆栈上运行clip stats。在图表中,注意到大的异常值已被移除,并且从一个视图到下一个的极大值通常更一致。
处理前后:
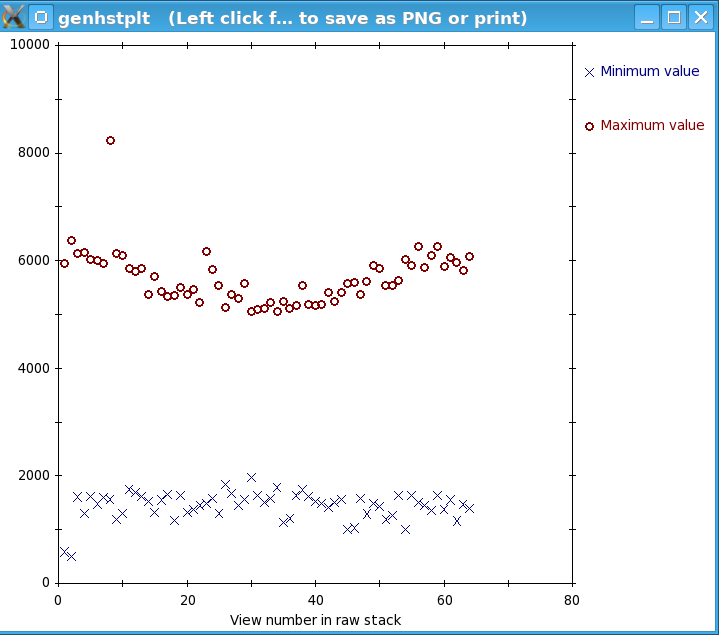
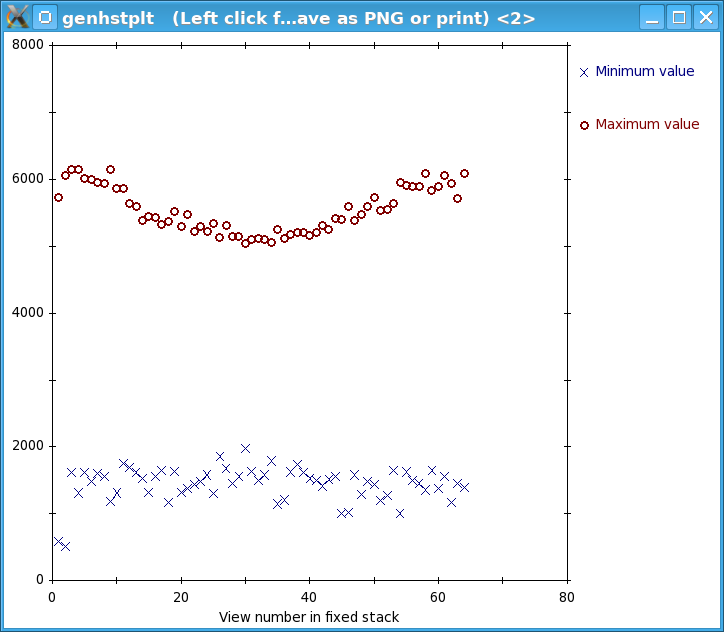
通常情况下,如果在3dmod中的黑白滑块没有非常接近,显示的对比度良好,并且修复后的堆栈中已经消除了原始堆栈输出中的异常值,则去除是足够的。由于在这里是这种情况,请按下Use Fixed Stack按钮。关闭统计和图表窗口,然后按下Axis A按钮返回到第一个轴。如果去除不够好,可以通过减小Peak criterion和Difference criterion的值来创建一个新的修复堆栈。有关此操作的详细信息,请参考《断层扫描指南》中的PRE-PROCESSING: REMOVING X-RAYS部分。
6. Coarse Alignment
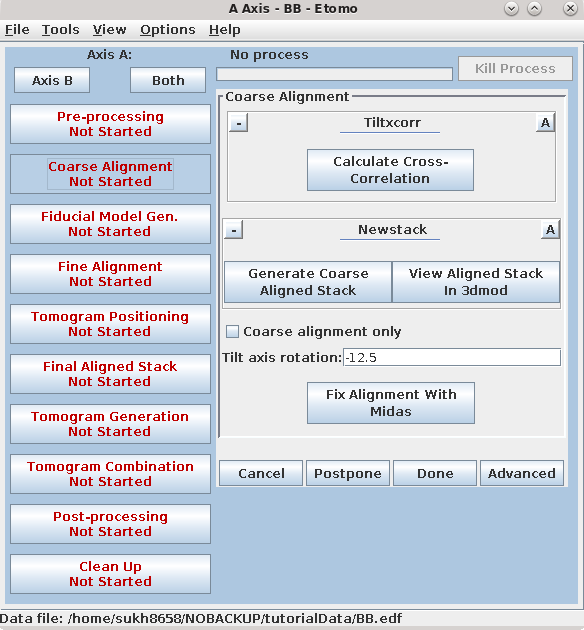
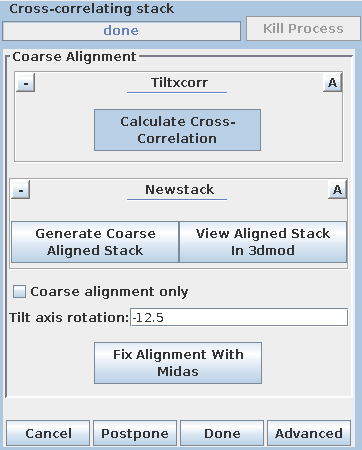
按下Calculate Cross-Correlation按钮会运行程序Tiltxcorr。该程序使用互相关法在倾斜系列的连续图像之间找到初始的平移对齐(即x和y方向上的平移)。输出文件BBa.prexf包含了一系列变换(或推荐的平移),这些变换将在下一步中应用到图像数据中。
按下Generate Coarse Aligned Stack按钮将运行两个程序。Xftoxg使用Tiltxcorr生成的变换来获得一组一致的或“全局”的对齐变换。然后,使用Newstack程序将这些新的变换应用到图像数据上。生成的输出文件是BBa.preali。您可以按下View Aligned Stack in 3dmod按钮来查看预对齐的堆栈。
如果需要,可以使用交互式程序Midas手动编辑大的图像平移。在这个数据集中,这不是一个问题;请参考Tomography Guide中的COARSE ALIGNMENT部分了解详细信息。如果运行Midas,则会使用Tilt轴旋转输入,因为Midas会旋转图像使得倾斜轴垂直。
可以选择Coarse alignment only复选框来进行没有基准对齐的快速层析图制作;请参考Tomography Guide中的Making a Quick Tomogram with Correlation Alignment部分了解详细信息。一旦对预对齐的堆栈满意,请按下"Done"按钮继续进行下一步。
7. Creating a fiducial model based on the position of gold particles
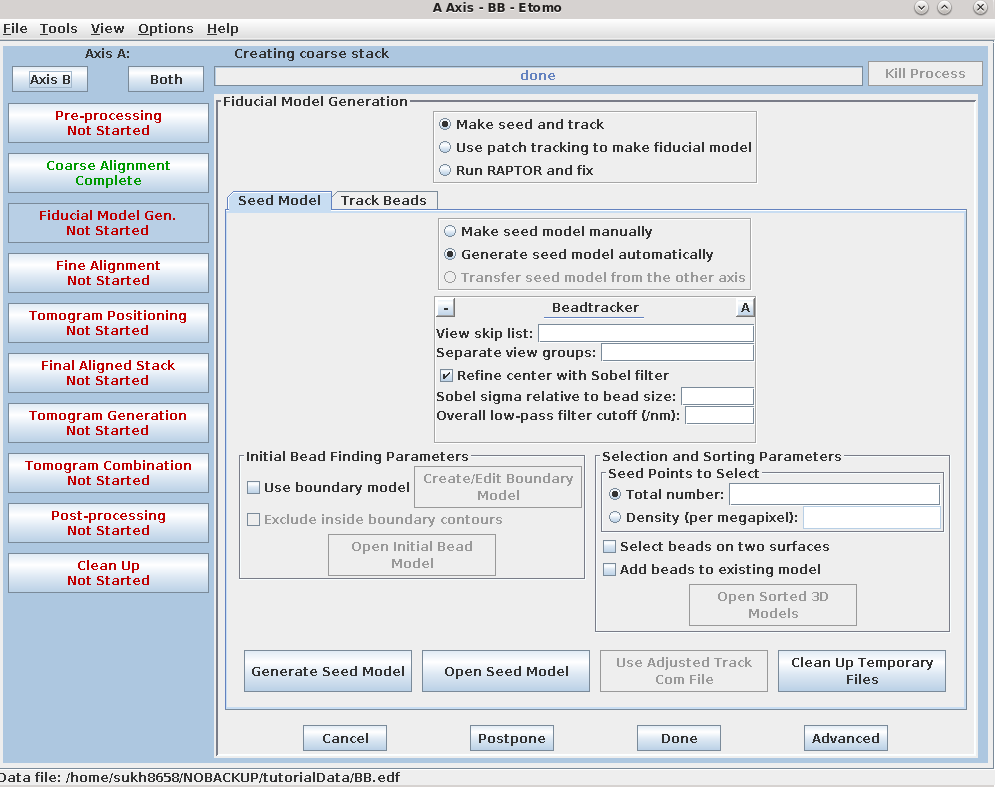
有三种选项可用于生成基准模型。最常见的方法是对一定数量的金标记点进行种子选择和跟踪Make seed and track,其中起始点可以手动或自动选择。这些起始点被称为种子模型,因为它们仅在一个截面上选择,并且从那里跟踪金标记点。另一个选项是使用补丁跟踪来生成基准模型Use patch tracking to make fiducial model,如果数据集中不包含金标记点或金标记点太少,则可以使用该选项。Run RAPTOR and fix将使用斯坦福开发的程序自动找到并跟踪倾斜系列中的金标记点。有关详细信息,请参阅Tomography Guide中的FIDUCIAL MODEL GENERATION部分。首先,我们将使用较新的功能自动选择种子点。然后,我们将返回并使用旧的方法,这对于说明在更具问题的数据集上需要遵循的步骤更好。
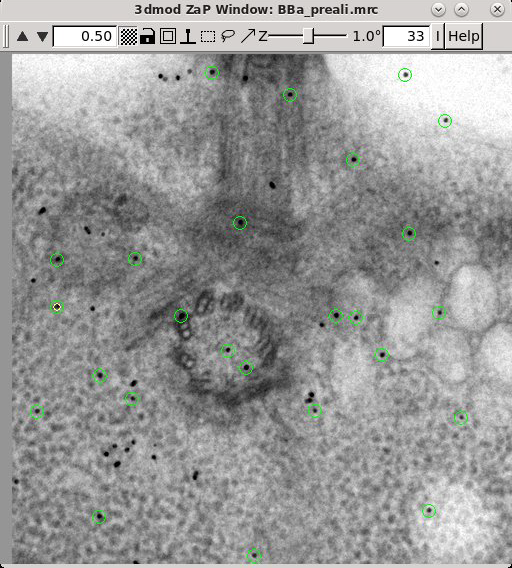
在Total number to track中输入25,并打开Select beads on two surfaces选项。按下Generate Seed Model按钮进行种子模型生成。
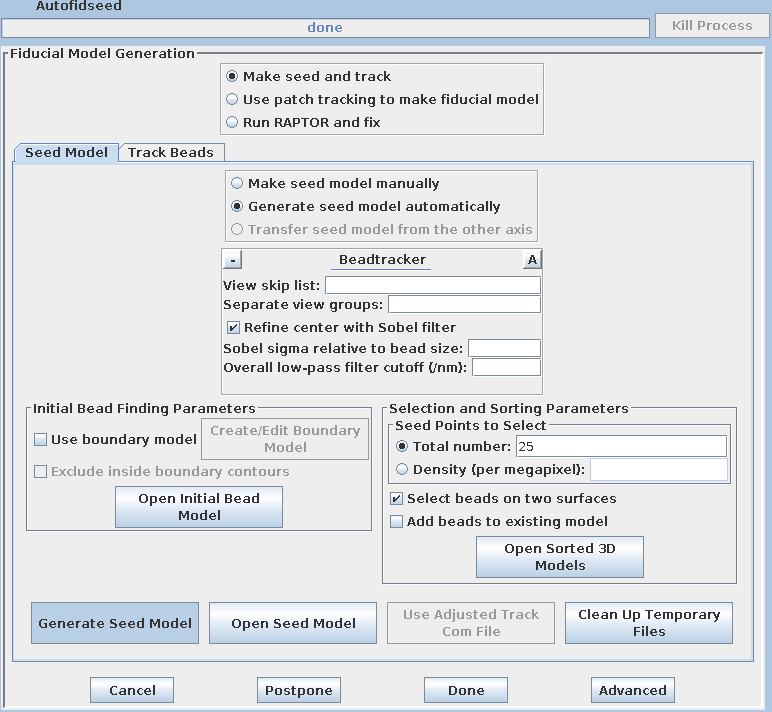
操作完成后,每个表面上选择的点数将在Project Log窗口中显示。按下Open Seed Model按钮查看所选择的点。需要向上滚动到1度(编号33)的部分才能看到这些点。绿色点表示底表面上的点,洋红色点表示顶表面上的点。下一步将使用此模型作为种子,构建完整的fiducial model。
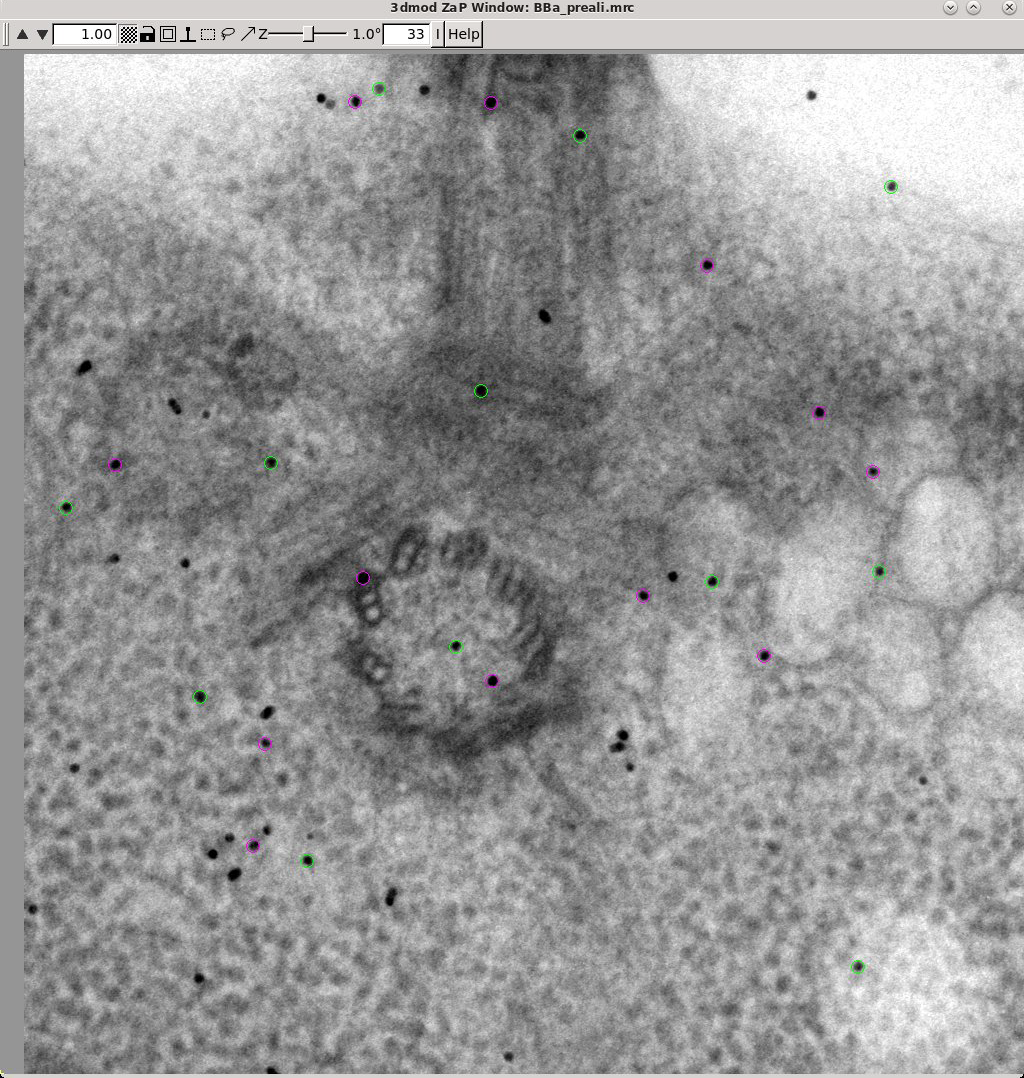
选择"Track Beads"选项卡。按下Track Seed Model处理按钮。
这将运行Beadtrack程序,以在所有其他截面上找到金粒子。由tracka.com创建的输出文件是BBa.fid,这是完成的参考模型。这个由计算机生成的模型并不完美,可能会存在参考模型中的间隙。在项目日志窗口中会显示缺失点的总数。如果缺失点较多,按下Track with Fiducial Model as Seed按钮,将使用参考模型作为种子重新运行跟踪程序。(对于这个样本数据集,不会填补更多的点。)
如果仍然存在缺失点,则下一步是一个迭代的过程,用于编辑参考模型。请注意,不需要填补所有的间隙,尤其是如果参考点数量充足的话,就像这里一样。然而,重要的是要确保绝大多数的参考点能够一直跟踪到倾斜系列的两端。
然后点击Fix fiducial model。这个过程将打开3dmod中的预对齐堆栈(BBa.preali)和fiducial model文件(BBa.fid)。
在填补间隙模式,将会弹出Bead Fixer对话框。Bead Fixer用于编辑参考模型。点击Go to Next Gap按钮(或使用空格键作为快捷键)。这将连接到一个有相邻截面上缺失模型点的点(用黄色圆圈标出)。使用Page Up键(当点上方出现向上箭头时)或Page Down键(当点上方出现向下箭头时)找到具有缺失点的截面,并使用鼠标右键将点添加到金粒子的中心。增加图像的放大倍数,可以使用**“+”键**,并调整截面的对比度,特别是在高倾斜角度时,这会很有帮助。
重复执行“Go to Next Gap”,直到在主要的3dmod窗口中出现“No more gaps found”的消息为止。通过选择File -> Save model或按下热键“s”来保存模型文件,并按下Done按钮进入精细对准步骤。
8. Alignment of serial tilts
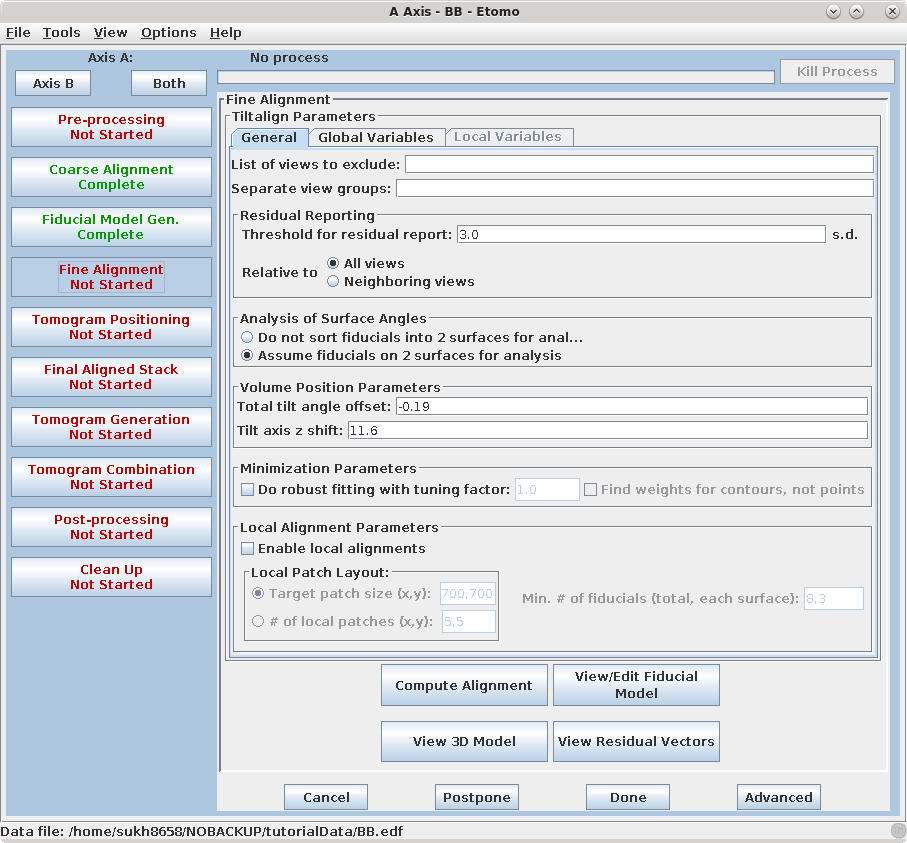
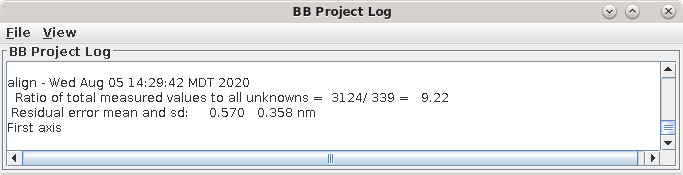
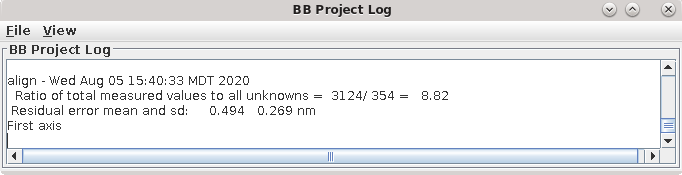
这个命令文件运行程序Tiltalign,用于求解倾斜视图中的位移、旋转、倾斜和放大差异。该程序利用金颗粒在标定模型中的位置和可变度量最小化方法找到最佳匹配。它创建一个日志文件,提供了所做操作的概要。在项目日志窗口中显示了总值与未知参数比值、残差误差的平均值和标准差的报告。要访问完整的日志文件,右键单击与该过程相关联的窗口区域。
这将打开一个菜单,分为三个或四个部分:第一部分允许您打开与当前过程相关的日志文件;第二部分允许您查看对齐参数的图表,但大多数其他面板上没有此选项;下一部分允许您打开与当前过程相关的man页面,最后一个菜单部分打开一般的帮助指南。选择“Align axis:a log file”以打开日志文件。这将打开一个带有完整日志和来自该日志文件的短节的选项卡文件。在示例中,第一个“Compute Alignment”运行的残差误差平均值为0.394 nm。
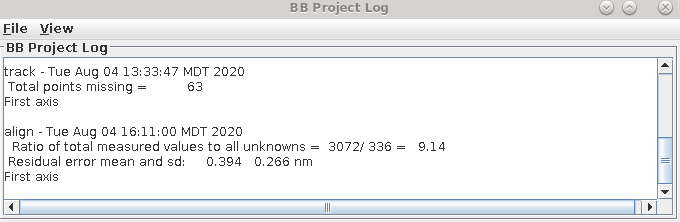
这个结果对于进一步说明下一步来说太好了,所以现在我们要进行备份。首先退出3dmod,然后转到终端窗口,并删除或重命名种子模型,例如:
mv BBa.seed BBa_auto.seed
接下来,点击左侧的Fiducial Model Gen按钮,选择Seed Model选项卡,切换回Make seed model manually。在ZaP(图像)窗口中,通过将光标置于金粒子中心并按下第二个鼠标按钮(默认情况下是中间鼠标按钮),在20-30个金粒子的中心放置一个模型点。由于选中了“自动新轮廓”,每个新的金粒子都将创建一个新的轮廓。由于选中了“自动居中”,3dmod应该确保模型点位于金粒子的中心,因此不需要像填充缺口时那样小心。在示例中(BBa.seed),我选择了25个金粒子;模型包含1个对象,25个轮廓,每个轮廓有1个点。保存这个种子模型。接下来,再次选择Track Beads选项卡。为了确保这次的结果不那么好,关闭“使用Sobel滤波器优化中心”(Refine center with Sobel filter)。
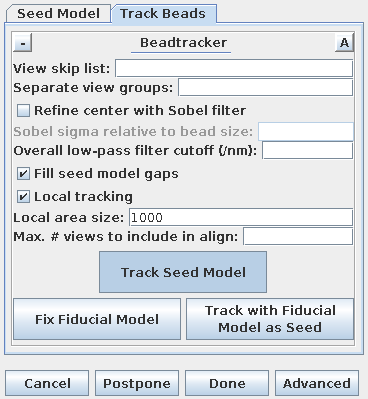
按下Track Seed Model以跟踪您的种子点。与之前一样,按下Fix Fiducial Model并填补任何间隙。按下"Down"继续进行精细对准步骤。
按下Compute Alignment。
精细对准步骤的目标是将残差误差均值降低到0.2 - 0.5纳米,适用于本教程数据集的像素大小。以纳米为单位来表示时,可以实现的残差误差均值往往与像素大小成比例关系,但不是完全成比例,特别是当像素大小变小时,各种限制因素将使误差无法降低到某个特定水平。
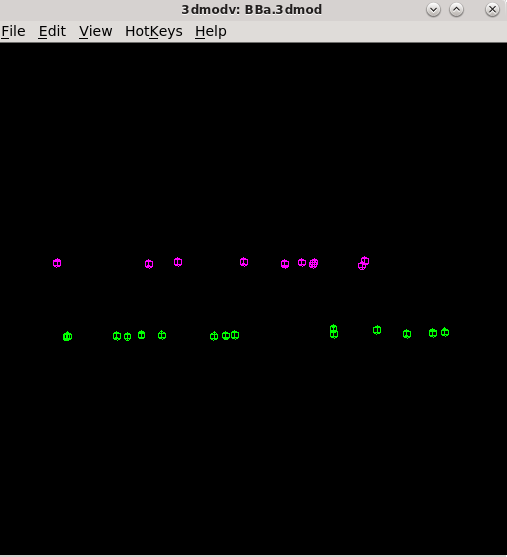
Tiltalign程序还创建了两个模型文件,提供有关标志模型的有用信息。第一个文件BBa.3dmod基于标志点的解算位置显示了标志的三维模型。在两个表面上存在的标志点被分配了两种不同的颜色;一个表面上为粉色,另一个表面上为绿色。通过点击Fine Alignment底部的View 3D Model按钮来查看该模型。
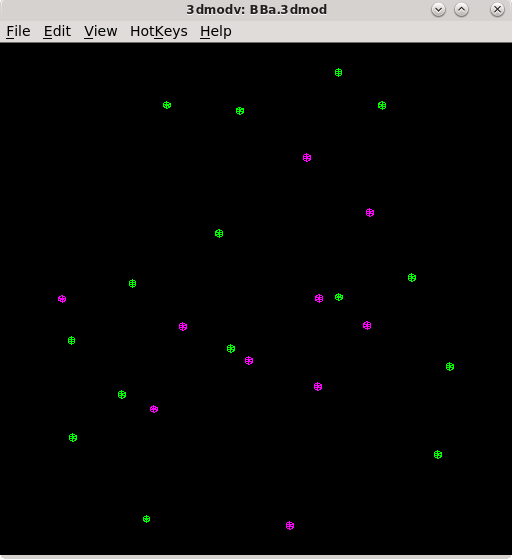
应该看到粉色和绿色球体在视野范围内均匀分布。按住小键盘上的数字8键旋转模型以侧面视角查看(在我的电脑上无效,可以通过上方HotKeys->Model Orientation and Zoom查看不同视角)。通过这个视角,你将看到两个表面的分离情况。避免使用具有在任何特定区域聚集的标志点的模型,因为这会影响对准结果。关闭3dmodv窗口。
Tiltalign生成的第二个模型是残差向量模型。点击精细对准框底部的View Residual Vectors按钮。这将在3dmod中打开预对准堆栈,并在每个切片上显示残差向量模型。
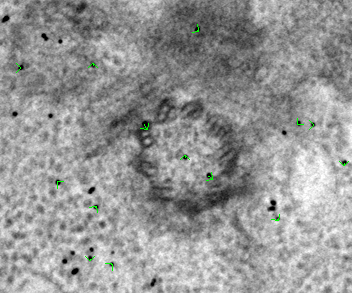
在模型中,当前模型点将显示为绿色箭头的起点,残差的位置将显示为箭头的末端。为了区分残差位移和实际模型点,残差位移被放大了10倍,因为位移通常非常小(<2像素)。在大型(>2k x 2k)图像中,残差模型通常会在某个区域显示较大的位移,而在其他区域则没有。在这些情况下,残差模型有助于决定是否需要局部对准。在此示例中,不需要进行局部对准。
保持3dmod处于打开状态,进行接下来的几个步骤,并点击View/Edit Fiducial Model按钮重新加载标志模型以进行编辑。这将打开Bead Fixer对话框,进入Fix big residuals模式,并加载aligna.log文件。接下来的迭代步骤涉及修复具有大残差的标志点。
请注意,您也可以使用robust fitting来获得几乎相同的结果,而无需修复标志点。鲁棒拟合将对残差较大的一小部分点减少或不给予权重,以避免它们对对准解决方案产生影响,因此如果剩余的点足够多以提供良好的解决方案,则对准应该是适当的。有关详细信息,请参阅Tomography Guide中的"Using robust fitting"部分。
在Bead Fixer对话框中点击Go to Next Big Residual。具有大残差的模型点将有一个红色箭头指向推荐的移动方向。您可能会看到模型点没有正确居中在金粒上。如果您在Bead Fixer对话框中点击Move Point by Residual,它将按推荐的量移动模型点。这在大多数情况下都有效,但如果推荐看起来不正确,可以通过将光标居中在金粒中央,然后点击第三个鼠标按钮来手动移动它。推荐位置是根据数学对齐模型拟合到所有标志模型点的位置预测,而不是基于对金粒在图像中实际位置的分析。如果您的样本畸变足够严重,以至于不适合对齐模型,那么箭头通常会指向实际金粒位置的相反方向。
重复选择Go to Next Big Residual和Move Point by Residual,直到没有残差为止。热键’将循环到下一个残差,热键;将按残差移动点。目标是将每个点居中在与之关联的标志上。残差箭头并不总是正确的。点也可以用鼠标右键点击移动。有时来自另一表面的标志会使当前标志的范围难以看清。使用Page Up和Page Down按钮区分标志(但始终返回到由"前往下一个大残差"选择的部分)。您可以通过按下"撤消移动"按钮来撤消按残差移动。
当没有更多残差需要修正时,保存模型文件并保持文件打开。访问aligna.log文件,并转到Surface Angles选项卡。
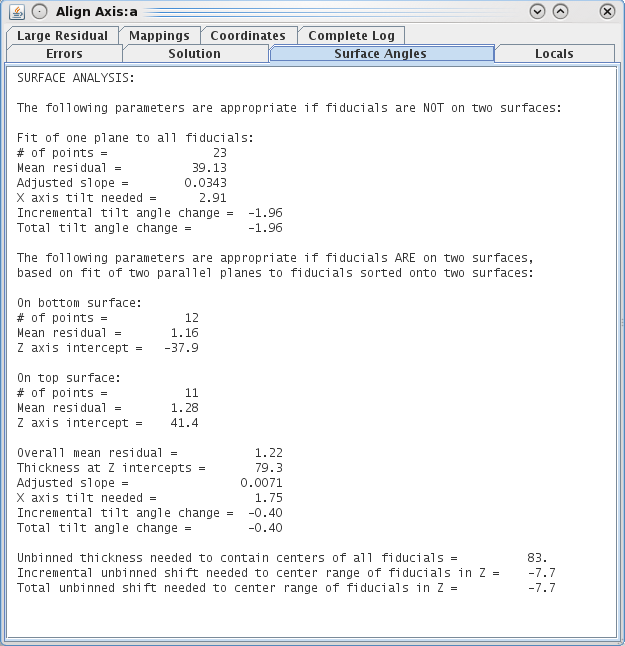
请注意底部附近的“Total tilt angle change”(在上面的例子中,该值为-0.40)。将此值放入Fine Alignment Volume Position Parameters框的Total tilt angle offset下。再次点击Compute Alignment按钮。将使用您的修正计算对齐,并且3dmod将重新读取生成的日志文件。请查看主3dmod窗口底部的文本框。再次进行修正具有较大残差的模型点。保存模型。
如果数据集在两个表面上有很好的金颗粒分布,可以解决扭曲问题。在Fine Alignment框中点击Global Variables选项卡:
在面板底部的Distortion Solution Type区域中选择Full solution选项。这将激活对两种类型的扭曲进行求解:X轴伸缩和斜变。保持默认的组大小,并点击“Compute Alignment”按钮运行对齐。当“Compute Alignment”完成后,3dmod将重新读取日志文件。
返回到3dmod窗口,重复修正Tiltalign在求解扭曲后生成的新残差。然后在Bead Fixer窗口中按下Save & Run Tiltalign按钮保存模型并计算对齐。只要您没有在Etomo中更改任何参数,您可以使用此按钮,而无需返回到Etomo。经过几次对齐和在3dmod中检查模型点后,最终的平均残差应该降低到0.2-0.4。在示例中最终的Residual error mean是0.494 nm,因为在这个版本上没有进行太多的改进。
9. Sampling the data set to create 3 small reconstructions
对数据集进行抽样以创建三个小的重建图像。
下一步的目标是通过对倾斜图像的顶部、中部和底部进行抽样,将重建图像进行平移和旋转,使其尽可能平坦并适应最小的体积。(当这些抽样不足够时,可以使用整个降采样后的重建图像来进行操作;详细信息请参阅《层析成像指南》中的“使用整个重建图像进行定位”部分。)
有两个旋转可以调整:绕倾斜轴的旋转,使切片在X-Z平面上观察时保持水平;以及绕X轴的旋转,使切片在整个重建图像的长度上保持相同的Z高度。
有一个功能可以自动检测切片的顶部和底部表面,这样就不需要绘制模型来定义表面。在这个轴上,我们将手动执行此操作;而对于第二个轴,我们将使用自动定位功能。
将Positioning tomogram thickness设置为400。这将创建一个比原始切片更厚的重建图像。按下Create Sample Tomograms按钮。
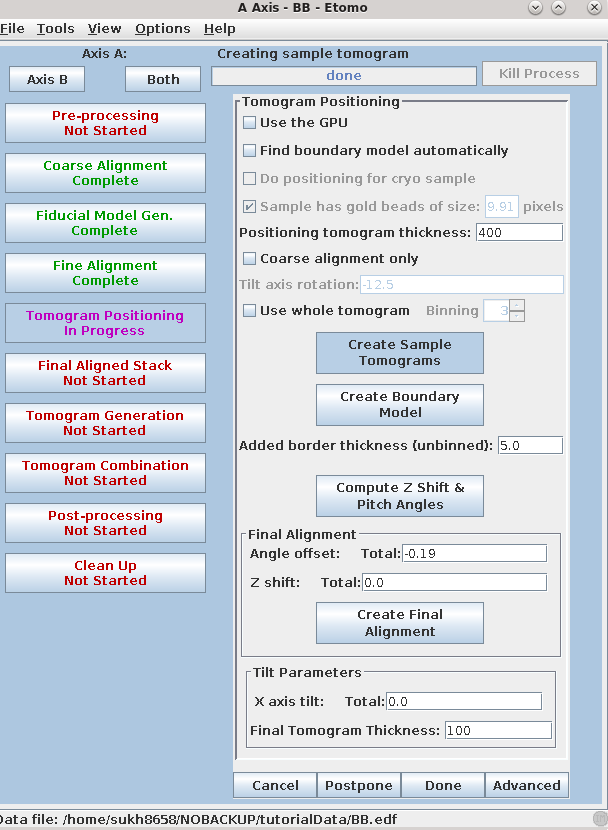
该命令文件首先从图像堆栈的顶部、中部和底部提取和对齐一个60像素的薄片。然后,程序使用这些样本从对齐的堆栈的顶部、中部和底部创建20个重建图像切片。这些输出文件的命名为topa.rec、mida.rec和bota.rec。按下Create Boundary Models按钮。这一步涉及创建一个模型来定义每个重建图像的顶部和底部表面。当按下“Create Boundary Models”按钮时,3dmod将一次读入所有三个重建图像,首先显示并从边缘视图查看topa.rec。3dmod还将从一个空模型tomopitcha.mod开始。
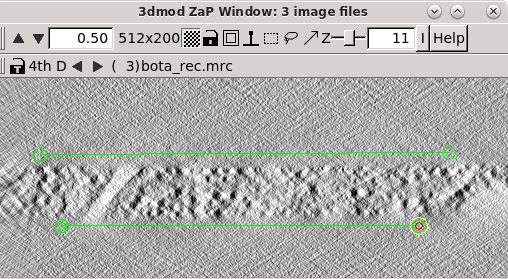
ZaP窗口的顶部工具栏上有一个’4thD’功能和一个向前和向后的箭头。如果点击向前箭头,可以依次切换到mida.rec和bota.rec重建图像。
首先从topa.rec开始。使用对比度滑块调整对比度。注意体积中心具有斑点状外观的物质。这是重建图像中含有生物材料的部分。**使用中间鼠标按钮,在顶部表面的左侧和右侧分别放置一个模型点,定义包含生物材料的区域,并在顶部表面的左侧和右侧分别放置第二个模型点。两个点之间将连接一条线。**用左侧和右侧各2个点建模切片的底部表面。通过点击Zap窗口顶部的’4D’右侧的箭头按钮,切换到mid.rec和bot.rec文件。重复对其他两个重建图像的顶部和底部表面进行建模。最终的模型应该有1个对象和6个轮廓,每个轮廓应该有2个模型点。保存这个模型文件。
通常情况下,最终的层析图的厚度应比您绘制的线之间的实际距离大10到20个像素。您可以调整"Added border thickness"的值来实现此目的。默认值为5,会使层析图增厚10个像素;将其改为10,可以增厚20个像素。
按下Compute Z Shift and Pitch Angles按钮。根据这些模型轮廓,tomopitch确定参数,使重建图像尽可能平坦,并尽可能适应最小的体积。这些偏移量会自动添加到现有的角度偏移、Z轴平移和X轴倾斜值中。最终的层析图厚度也会由Etomo根据tomopitch日志输入。
10. The final runs to create the full-size tomogram
点击Create Final Alignment。然后Done。
点击Final Alignment这个任务,Create Full Aligned Stack。该命令将对最终对齐堆栈的全尺寸图像应用对齐变换。输出文件命名为BBa.ali
按下View Full Aligned Stack可以查看完整的对齐堆栈,尽管这不是必需的。还有一些可选的步骤,如CTF校正、擦除参考点和对齐堆栈的滤波,但在这里并不需要。按下“Done”按钮,进入下一步。
Tilt Parameters部分的默认滤波参数适用于许多情况,但可以进行更改,或者可以对对齐堆栈进行滤波以减少重建图像中的噪音(详见层析图生成(TOMOGRAM GENERATION)部分的层析图指南)。
11. Generate Tomogram
Tomogram Generation 标签页按下Gnerate Tomogram,等待一会儿,然后点击View Tomogram in 3dmod查看。可以点击Delete Intermediate Image Stacks节省空间。当层析图生成完成后,按下3dmod中的“View Tomogram”按钮来查看它。可以选择在这一步中删除中间图像堆栈以节省空间。通过按下鼠标的第二个按钮,可以逐个浏览从层析图的顶部表面到底部表面的连续层面图像。
12. On to the second axis (Axis B)
Etomo窗口上方选择Axis B,前往Coarse Alignment。然后Done。
为了合并两个倾斜轴,要确保至少有一些(8-10个)被跟踪的标记颗粒在两个序列中都存在。为了实现这一点,使用Transferfid程序。
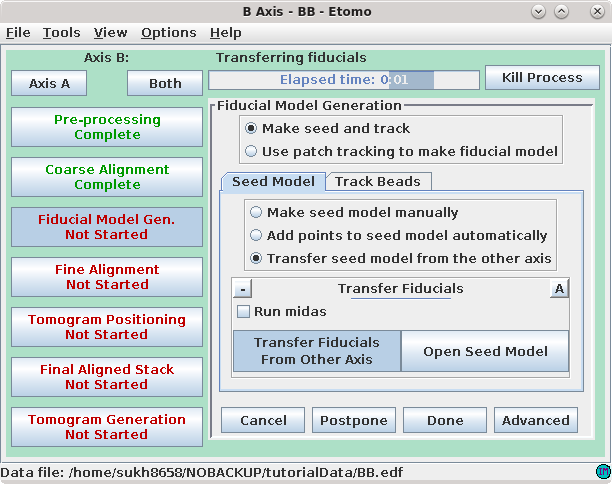
点击Transfer Fiducials From Other Axis按钮将运行Transferfid程序,该程序根据第一个轴的标记模型创建第二个轴的种子模型。程序将搜索两个序列中最匹配的一对视图,然后将第一个序列的标记传输到第二个序列以创建种子模型。最后,程序将列出第一个和第二个轴之间的标记对应关系,并在项目日志窗口中报告这些信息。
如果有一些标记没有成功传输,请不要担心。在这个示例中,有19个标记对应成功。这个数量有点少,这可能是因为两个轴的匹配程度不够好。你可以手动添加一些额外的点,但也可以使用自动添加功能。只需选择Add points to seedmodel automatically即可打开自动添加面板。点击Add beads to existing model来向由Transferfid生成的种子模型中添加更多的点,该模型命名为BBb.seed。选择Track Beads选项卡,并在Axis B窗口中进行种子模型的跟踪。
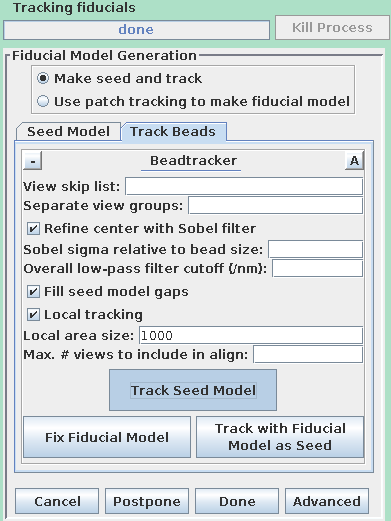
这将自动跟踪"B"集合的标记点。按照上面介绍的步骤,Fix Fiducial Model Using Bead Fixer修复BBb.fid文件中的gaps。当gaps修复完毕后,点击"Done"以继续进行精细对准和断层图计算步骤。
现在,您将按照与"A"轴设置相同的步骤和程序继续进行。简要地说:
- 在精细对准框中,按照前面介绍的步骤,分别点击"General"和"Global variable"选项卡下的
Compute Alignment按钮。 - 在这一步,您将开始进行迭代对准过程,方法与"A"轴设置相同:编辑具有较大残差的模型点,保存模型,并进行对准计算。
- 当对准完成后,点击"完成"并继续进行断层图生成步骤。进行自动断层图定位。要使用自动定位功能,请打开"Find boundary model automatically"选项,并将"Positioning tomogram thickness"设置为400。
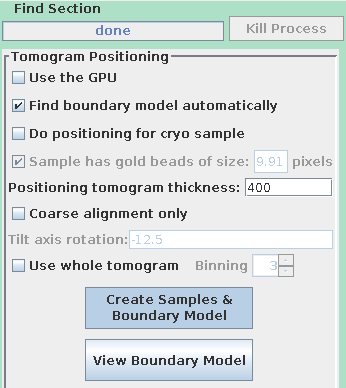
自动断层图定位可以使用样本或整个断层图进行,当标准样本位置上的材料不足时,整个断层图的方法更可靠。在这里,我们仍然使用样本,因为从整个断层图中检查边界模型需要稍微不同的方法(有关详细信息,请参阅《Tomography Guide》中关于使用整个断层图进行自动定位的部分)。
点击Create Samples & Boundary Model按钮。完成后点击查看边界模型按钮,并检查绘制的轮廓。
按照上面对A轴的描述,继续执行“Final Aligned Stack’”的步骤。
按照上面对A轴的描述,继续执行“Tomogram Generation”的步骤。
13. Combing the two axis
回到Axis A界面。点击Tomogram Combination。
Tomogram Combination 面板包括三个标签页:Setup,Initial Match,和Final Match。
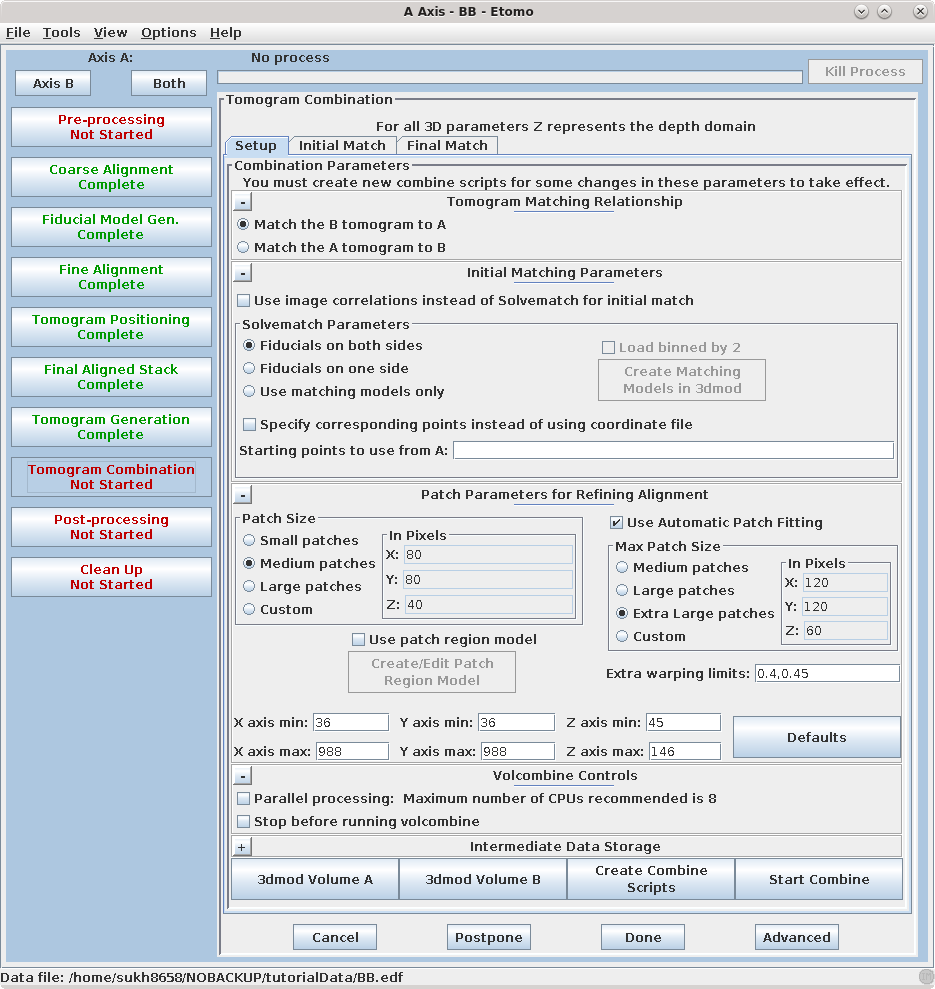
Setup窗口是提供有关特定数据集的信息的地方。第一部分描述了层析图匹配关系Tomogram Matching Relationship。通常情况下,将B层析图与A匹配。Solvematch Parameters 要求提供关于参考点分布的信息。在这个例子中,参考点分布在两个侧面上。对于这个数据集,程序可以轻松地同时拟合所有的点,因此不需要填写Starting points to use from A。
下一部分包含了用于使用局部3D互相关进行对齐的Patch参数的信息。选择Small patches。对于大多数数据集,通常需要使用中型或大型的patch,但是这个数据集已经被缩小了,所以小型patch中有足够的信息。注意选择了使用自动patch拟合Use Automatic Patch Fitting。这个功能会在找不到足够的信息时自动增加patch的大小。不会不必要地增加patch的大小。当选择自动patch拟合时,程序还会分析整个体积中存在的信息量,并尝试排除没有足够信息的patch。如果这个过程效果不好,那将不得不回到旧的方法,指示要从中提取patch的区域。为了让您对这些方法有一些经验,我们将按照设置体积的坐标限制的程序进行操作。
当需要指定从中提取patch的体积限制时,查看要匹配的层析图是很重要的。几乎从不使用整个Z轴范围而不进行自动patch拟合,即使对于X轴和Y轴也可能不适合使用默认值。要找到限制范围,请点击面板底部的3dmod Volume A按钮。这将在3dmod中打开BBa.rec。浏览图像并决定X轴、Y轴和Z轴的哪个范围包含了用于匹配体积的有用信息。在这个例子中,保留了X轴和Y轴的默认值。在这个例子中,Z轴的最小值和最大值被设置为模糊区域约占片段一半的第一和最后一张切片(45和146)。
有时,仅仅设置坐标限制并不能很好地消除“空白”区域,以使两个轴能够组合起来。在这种情况下需要创建一个patch区域模型。要创建一个patch区域模型,勾选Use patch region model的复选框,然后按下Create/Edit Patch Region Model按钮,打开正在匹配的轴。在模型模式下,在包含生物材料的区域周围绘制闭合轮廓。在层析图中每隔大约10个切片进行一次。保存模型,命名为patch_region.mod。创建patch区域模型对于重建具有大量树脂或图像中存在开放区域的数据集特别有用。 这个示例数据集不需要patch区域模型。
当设置面板的参数输入完成后,点击Create Combine Scripts以创建一系列的命令文件,以运行合并过程中的各种程序。忽略弹出的警告消息。点击Start Combine开始双轴层析图合并过程。Etomo将在运行各种程序时自动切换到初始匹配(Initial Match)和最终匹配(Final Match)选项卡。层析图合并完成后,点击Open Combined Volume以查看最终层析图。点击Done以进入最后的步骤。
14. Post-processing and Clean-up of intermediate files.
后处理包括volume trimming 和 byte scaling 步骤,然后删除中间文件。还有选项可以将体积展平和创建压缩的体积(flatten the volume and to create a squeezed down volume);在处理非常大的数据集时,这两个选项都非常有用,尤其是在重建串行切片(serial sections)时,flatting 选项尤为有帮助。关于它们的使用方法,请参阅指南中的POST-PROCESSING部分。
两个轴合并后的最终重建始终被称为sum_rec.mrc。(如果您有一个单轴数据集,则此时重建文件的名称将是数据集名称后跟_full_rec.mrc)。通过按下3dmod Full Volume打开sum_rec.mrc重建。逐步查看重建并确定最终体积的X、Y和Z范围。设置X和Y范围的一种便捷方法是在Zap窗口的工具栏上打开带有虚线矩形的橡皮筋,在所需区域的左上角按下第一鼠标按钮,然后拖动鼠标到右下角。如果需要,也可以设置Z范围;按下“Lo”设置最小Z,按下“Hi”设置最大Z。当您按下Get XYZ Volume Range from 3dmod时,Etomo将从3dmod中检索橡皮筋的X和Y值(如果设置了Z值也会检索)。
在这个例子中,X轴有一些修剪,Y轴使用了默认范围。Z轴(在翻转的重建中)的范围已设置为Z最小值为52,Z最大值为142,以排除非细胞材料。最后,设置一个缩放范围以找到不包括金颗粒的切片范围。在这个例子中,缩放是基于切片的,范围为55至140切片(使用3dmod输入数字请参见下文)。
有时候不可能找到不含金颗粒的切片范围。例如,在这里使用的样品中,一些金颗粒出现在细胞外的塑料树脂上,并出现在我们希望用于缩放的相同切片上。将X和Y缩放范围限制在不包含任何这样的金颗粒的区域将改善最终重建的对比度。按下3dmod Full Volume按钮并打开ZaP窗口。转到显示金颗粒的切片,并在一个不包含它们的区域周围放置一个橡皮筋。要做到这一点,按下Z滑块左侧的橡皮筋切换按钮。

在目标区域的左上角按下鼠标左键,将鼠标拖动到目标区域的右下角。然后将Z滑块移动到缩放范围的下部(55)并按
Lo。移动Z滑块到缩放范围的上部(140),并按Hi。

进入Etomo的Scaling框中,点击Get XYZ Sub-Area From 3dmod按钮。这将导致Etomo检索您选择的X、Y和Z值。默认的重新定向选项将围绕X轴旋转最终的体积,以便可以方便地通过3dmod和其他程序进行读取,无需特殊选项。点击Trim Volume按钮运行Trimvol。Trimvol是一个用于修剪体积并将其转换为字节的单个工具。最后,通过点击3dmod Trimmed Volume查看最终修剪的体积(命名为BB_rec.mrc)。点击"Done"继续进行文件清理。
清理中间文件:
清理文件非常重要!由于您从其中一个堆栈中删除了X射线,您可以首先进行的步骤是“存档”原始堆栈。该过程将原始堆栈替换为与当前原始堆栈的差异的小型、高度压缩的文件。点击Archive Original Stacks按钮。这个过程是可逆的:如果需要,可以使用完成时显示的命令轻松恢复原始堆栈。生成冷冻断层图的过程会产生许多大型中间文件。中间文件清理框列出了我们认为是中间的、非必需的文件,可以删除。一旦您确信最终的冷冻断层图确实是最终版本,您可以通过将中间文件突出显示并按下Delete Selected按钮进行选择删除。要突出显示所有文件,请单击一个文件,然后同时按下Ctrl和A键(在Mac OS中是Command和A键)。
最终的双轴冷冻断层图被命名为BB_rec.mrc,并可以在Etomo之外使用命令3dmod BB_rec.mrc进行查看。
有关在冷冻断层图中对许多细胞特征建模的信息,请参考《Introduction to 3dmod》。这是一个简单的数据集,您在处理自己的数据时可能会遇到更多问题,因此最好在开始处理真实数据集时阅读《Tomography Guide》。此外,如果您阅读《Using Etomo》,您将能够更有效地使用Etomo,其中解释了访问帮助和设置并行处理等功能。最后,如果您按照《Batch Reconstruction Tutorial》中的指示处理此数据集,您将看到批处理在加速重建方面的潜力。
指路:
Introduction to 3dmod
Tomography Guide
Using Etomo
Batch Reconstruction Tutorial
A u t h o r : C h i e r Author: Chier Author:Chier
![[PyTorch][chapter 40][数据增强]](https://img-blog.csdnimg.cn/bd671d65e220400fab39371017cc9c7b.png)