失踪人口回归的第二期,继续说一说用PSM处理不平衡数据。
一、啥叫PSM
PSM全称为Propensity Score Matching,翻译过来就是倾向匹配得分,为了省流,让小Chart介绍一下:

放到我们的数据就是:根据某个特征,从对类别0的1671例中挑出一部分来跟类别1来匹配,形成新的数据集,然后建模。其实就是多变少的策略。
可以1:1、1:2、1:3等进行匹配,没啥固定说哪种比例好,慢慢试吧。
多说一句,PSM需要指定匹配的一个或者多个自变量(特征),一般来说,选取人口学特征,比如年龄、性别、民族啥的。也可以根据专业实际情况选别的,反正有个说法即可。
下面来操作:
二、SPSS实现1:1的PSM匹配
高能预警:SPSS22及以上自带1:1的PSM,对于其他版本或者想要体验完整版功能,就要安装相应的软件(R、SPSS R插件、PS matching插件)。据说,超级难装。
说实话,我也是第一次用,然后我看了看我的SPSS版本,哭了:

没办法,默默装了一个:

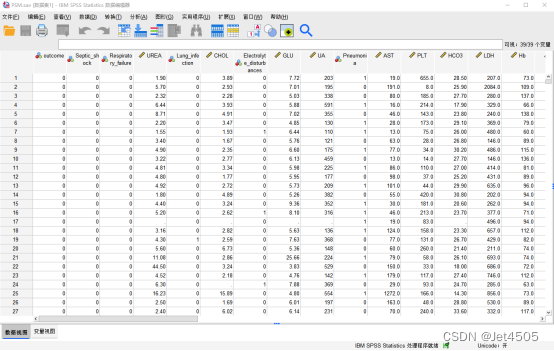
(1)数据导入:
照着点:文件→导入数据→CSV数据:

(2)倾向性评分匹配
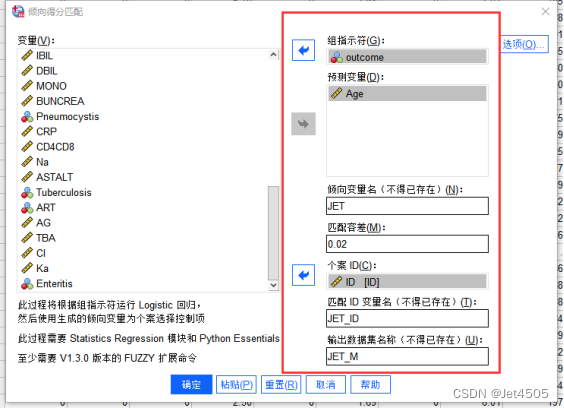
照着点:数据→倾向得分匹配→打开对话框:

组指示符:将因变量(outcome)放入;
预测变量:需要匹配的变量放进去,我这就一个,你们也可以放多个;
倾向变量随便起一个名字:JET;
匹配容差,也叫卡钳值,用来设置倾向性评分匹配标准,卡钳值设置的越小,匹配后可比性越好,卡钳值太小会导致匹配难度会加大,我们先设0.02;
个案 ID填入样本的ID,也就是排序的序号;
匹配ID变量名:随便起一个名字,用来明确匹配成功的样本ID;
输出数据集名称:把匹配的观测对象单独输出一个数据集。
(3)额外的设置
点击右上角的选项按钮,弹出对话框:
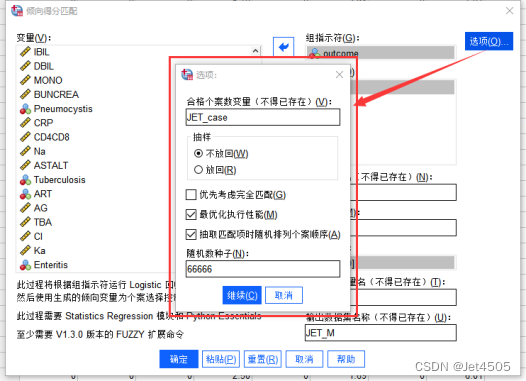
合格个案数变量:新生成一列,用来明确实验组中某一个观测对象,在对照组中有多少个观测对象满足与其匹配的条件;
抽样:不放回抽样,顾名思义;
优先考虑精确匹配:优先找实验组和对照组的JET值(倾向变量)一样的;
最优化执行性能:综合考虑精确匹配和基于设定的卡钳值范围内匹配的模糊匹配;
抽取匹配项时随机排列个案顺序:如果对照组有多个满足匹配条件的观测对象,就会随机将其与实验组观测对象匹配。注意是随机,所以为了能重复,需要设置我们熟悉的随机种子数,比如66666。
(4)看结果
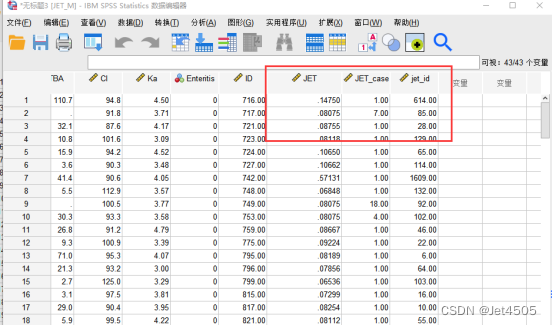
(a)点击确定以后,会生成一个新的数据集(JET_M),重点看新变量:
JET_case代表对照组中有几个符合匹配条件的观测对象(等于4,说明有4个对照组观测对象符合匹配条件);
JET是算出的倾向性评分;
Jet_id代表匹配成功的ID号。
(b)看看输出窗口的结果:
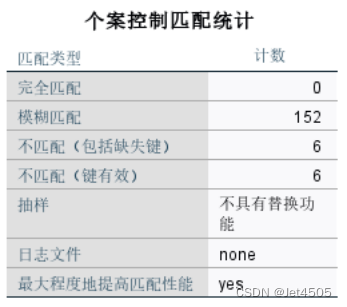
全是模糊匹配,一共152对;
(c)匹配后的数据库整理输出:
首先,把配对成功的数据撸出来:
记得是打开新的数据集(JET_M),照着点:数据→选择个案→打开新窗口:

设定条件,JET_id要大于等于1,筛选出匹配成功的对子;
挑选出来的在生成新的数据集,叫做JET_M_NEW;
记得左边选择ID:
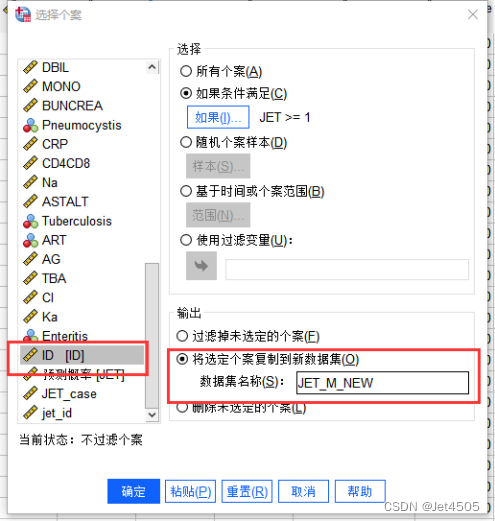
点击确定后,生成新的数据框JET_M_NEW:

然后导出数据成scv文件,转到python试试性能。
(5)Xgboost建模
无脑套代码:
#加载相关包
import xgboost as xgb
from xgboost import plot_importance
import numpy as np
import matplotlib.pyplot as plt
import pandas as pd
from sklearn.feature_selection import SelectFromModel
from xgboost import plot_importance
import math
#导入数据
dataset = pd.read_csv('wwd3.csv')
dataset = dataset.fillna(dataset.median())
X = dataset.iloc[:,1:16].values
y = dataset.iloc[:,0].values
#数据拆分成训练集、测试集
from sklearn.model_selection import train_test_split
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size = 0.30, random_state = 6588)
#数据归一化
#from sklearn.preprocessing import StandardScaler
#sc = StandardScaler()
#X_train = sc.fit_transform(X_train)
#X_test = sc.transform(X_test)
#模型构建
boost = xgb.XGBClassifier(
n_estimators=200,
max_depth=9,
min_child_weight=3,
subsample=0.9,
colsample_bytree=0.9,
scale_pos_weight=1,
gamma=0.1,
reg_alpha=7,
)
boost.fit(X_train, y_train)然后悲剧了:

显示的是格式问题,不是所有数据都是数值型,所以我们怎么看数据的每一列都是什么类型呢?无脑问小Chat即可:

我们试一下:

果然,一堆的“object”类型。所以需要把“object”全部改成“float64”,怎么搞,继续问小Chat:

复制TA提供的2行代码即可,其他不变:
#导入数据
dataset = pd.read_csv('wwd3.csv')
# 将所有能够转换为数字的object类型转换为float64类型
dataset = dataset.apply(pd.to_numeric, errors='coerce')
# 将剩余的object类型转换为float64类型
dataset = dataset.astype('float64')
dataset = dataset.fillna(dataset.median())
X = dataset.iloc[:,1:16].values
y = dataset.iloc[:,0].values看看结果:

灵敏度升高了,特异度下降了,至少没那么割裂,不过还有改进空间。毕竟样本量少了那么多,所以还得试试1:n的匹配。
三、R实现1:n的PSM匹配
说实话,我也不知道怎么做。
但是有GPT,问题不大。首先,需要写一个咒语,说清楚我们的需求:
我有一批{数据},数据包括1个因变量“outcome”和38个自变量。我希望通过R进行1:3的倾向性评分匹配,使得“outcome=1”和“outcome=0”的两组的自变量“Age”不存在显著性差异。并且整理和输出进行匹配后的数据,数据格式与{数据}一致,保存为“psm.csv”。请写出R的代码。
数据格式如下,仅列出前5行数据:
{复制你的数据,表头和前4行}

然后就给了完整代码:
# 安装并加载所需的R包
if (!require(MatchIt)) install.packages('MatchIt')
library(MatchIt)
# 加载数据
data <- read.csv('wwd3.csv', sep = '\t')
# 使用中位数填充缺失值
for (column_name in colnames(data)) {
median_value <- median(data[[column_name]], na.rm = TRUE)
data[[column_name]] <- ifelse(is.na(data[[column_name]]), median_value, data[[column_name]])
}
# 提取自变量和因变量
X_columns <- colnames(data)[-1]
y_column <- 'outcome'
# 使用逻辑回归模型进行倾向性评分计算
ps_model <- glm(outcome ~ ., data = data, family = binomial())
# 保存倾向性评分
data$propensity_score <- ps_model$fitted.values
# 1:3的倾向性评分匹配
matched <- matchit(outcome ~ propensity_score, data = data, method = "nearest", ratio = 3)
# 输出匹配后的数据
matched_data <- match.data(matched)
write.csv(matched_data, 'psm.csv', row.names = FALSE)
# 检查匹配结果
summary(matched)跑了一下,很顺序,无报错。匹配好的数据已经自动输出:

R的输出框也给出了匹配结果,不过有些乱码,我让GPT帮整理一下:

收工!后面跑分类模型大家自己弄了哈。
有了GPT,效率大大提高!