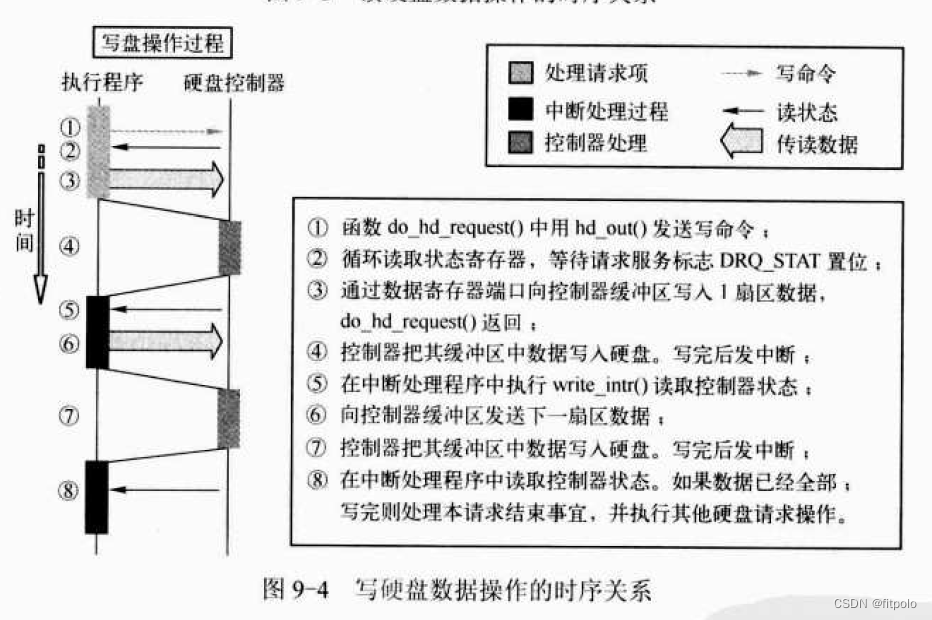
一、概述
COVID-19是当前全球面临的一项重大挑战。 本文将介绍如何使用R语言分析COVID-19相关数据,探索其感染率、死亡率和人口特征的相关性,以及使用统计建模方法预测COVID-19的死亡率。
二、数据导入与筛选
COVID-19 Data Repository by the Center for Systems Science and Engineering (CSSE) at Johns Hopkins University:由约翰霍普金斯大学的CSSE实验室创建的数据存储库,其中包括每个国家记录的每个检测样本结果。
2.1 加载数据集
# 加载必要的 R 包
# install.packages("readr")
library(readr)
# install.packages("dplyr")
library(dplyr)
# 下载数据集
confirmed_cases_url <- "https://raw.githubusercontent.com/CSSEGISandData/COVID-19/master/csse_covid_19_data/csse_covid_19_time_series/time_series_covid19_confirmed_global.csv"
confirmed_cases_df <- read.csv(confirmed_cases_url) # 读取全球每日确诊数据
deaths_url <- "https://raw.githubusercontent.com/CSSEGISandData/COVID-19/master/csse_covid_19_data/csse_covid_19_time_series/time_series_covid19_deaths_global.csv"
deaths_df <- read.csv(deaths_url) # 读取全球每日死亡数据
recovered_url <- "https://raw.githubusercontent.com/CSSEGISandData/COVID-19/master/csse_covid_19_data/csse_covid_19_time_series/time_series_covid19_recovered_global.csv"
recovered_df <- read.csv(recovered_url) # 读取全球每日康复数据2.2 对数据进行清洗、整理
接下来,我们需要对COVID-19数据进行清洗,滤除无效数据或日志中的其他纷杂信息,并对数据进行初步的整理。该过程包括对数据的缺失值进行处理,所选取的时间段和数据的筛选。
library(tidyr)
#install.packages("lubridate")
library(lubridate)
# 移除Province/State、Lat、Long列
confirmed_cases_df <- confirmed_cases_df %>%
select(-c("Province.State", "Lat", "Long")) %>%
gather(key = "date", value = "confirmed_cases", -c("Country.Region"))
deaths_df <- deaths_df %>%
select(-c("Province.State", "Lat", "Long")) %>%
gather(key = "date", value = "deaths", -c("Country.Region"))
recovered_df <- recovered_df %>%
select(-c("Province.State", "Lat", "Long")) %>%
gather(key = "date", value = "recovered", -c("Country.Region"))2.3 数据格式的转换
为了正式进入分析过程,我们还需要将数据格式转换为符合R中数据类型的格式。 在这里,我们可以使用数据框架(data.frame)和列表(list)等数据类型存储数据
# 去除重复项目,方便整合
confirmed_cases_df <- confirmed_cases_df[!duplicated(confirmed_cases_df[c("Country.Region", "date")]),]
deaths_df <- deaths_df[!duplicated(deaths_df[c("Country.Region", "date")]),]
recovered_df <- recovered_df[!duplicated(recovered_df[c("Country.Region", "date")]),]
# 汇总三个数据集
combined_data <- confirmed_cases_df %>%
left_join(deaths_df, by = c("Country.Region", "date")) %>%
left_join(recovered_df, by = c("Country.Region", "date"))
# 日期转换
combined_data$date <- as.Date(combined_data$date,"X%m.%d.%y")
# 移除有缺失的数据
combined_data <- na.omit(combined_data)2.4 数据的基本统计分析
library(ggplot2)
library(scales)
# 全球每日感染、死亡和康复情况总表 (2020/1/22 - 2023/5/14)
global_total_cases_df <- combined_data %>%
group_by(date) %>%
summarise(confirmed_cases = sum(confirmed_cases, na.rm = TRUE),
deaths = sum(deaths, na.rm = TRUE),
recovered = sum(recovered, na.rm = TRUE)) %>%
ungroup()
# 每日全球新增感染病例数以及每日新增死亡、康复数 (2020/1/22 - 2023/5/14)
global_new_cases_df <- global_total_cases_df %>%
mutate(new_cases = confirmed_cases - lag(confirmed_cases, default = 0),
new_deaths = deaths - lag(deaths, default = 0),
new_recovered = recovered - lag(recovered, default = 0)) %>%
filter(date >= "2020-02-15") # 只展示自2020/2/15以后的数据
ggplot(global_new_cases_df, aes(x = date)) +
geom_bar(aes(y = new_cases), stat = "identity", fill = "lightblue", alpha = 0.8) +
geom_line(aes(y = new_deaths), colour = "red") +
geom_line(aes(y = new_recovered), colour = "green") +
scale_y_continuous(label = comma) + # 为y轴标签添加千分位符号
labs(x = "日期", y = "人数", title = "全球每日新增确诊、死亡和康复病例数") +
theme_minimal()+ylim(0,500000)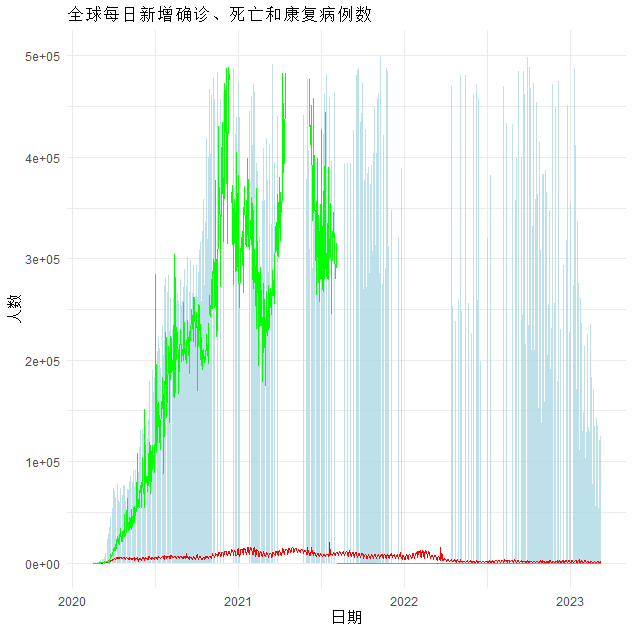
三、COVID-19统计分析
3.1 多元线性回归模型
使用多元线性回归模型分析各因素对COVID-19感染率和死亡率的影响.
世界各国人口数量统计1960-2021
# 读取人口统计数据
population_df <- read.csv("population_by_country_2020.csv")
population_df <- population_df %>%
select(-c("Country.Code", "Indicator.Name","Indicator.Code")) %>%
gather(key = "date_p", value = "population", -c("Country.Name"))
# 时间格式转换
population_df$date_p <- as.Date(population_df$date_p,"X%Y")
# 增加year参数,方便比对
combined_data$year <- year(combined_data$date)
population_df$year <- year(population_df$date_p)
# 按国家/地区和日期进行数据合并
combined_data <- combined_data %>%
left_join(
population_df,
by = c("Country.Region" = "Country.Name", "year" = "year")
) %>%
mutate(
confirmed_per_million = confirmed_cases / population * 1000000
) %>%
select(
Country.Region, date, confirmed_cases, deaths, recovered, population, confirmed_per_million
)
#移除有缺失的数据
combined_data <- na.omit(combined_data)
# 构建多元线性回归模型
model <- lm(confirmed_per_million ~ population + deaths + recovered, data = combined_data)
# 查看模型摘要
summary(model)
# 绘制模型图形
# install.packages(c("ggplot2"))
library(ggplot2)
install.packages("ggfortify")
library(ggfortify)
autoplot(model) +
scale_shape_manual(values = c(1, 19)) +
theme_bw() +
labs(title = "残差图", x = "拟合值", y = "残差值") +
stat_smooth(method = "loess", se = FALSE, color = "blue", size = 1.5) +
geom_hline(yintercept = 0, color = "red", linetype = "dashed") +
geom_text(aes(label = rownames(combined_data)), size = 2, color = "black", hjust = -0.3, vjust = 0.3)
结果展示:
Call:
lm(formula = confirmed_per_million ~ population + deaths + recovered,
data = combined_data)
Residuals:
Min 1Q Median 3Q Max
-77079 -29463 -25392 3497 387503
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 3.059e+04 1.627e+02 188.044 <2e-16 ***
population -7.821e-05 1.615e-06 -48.433 <2e-16 ***
deaths 3.271e-01 4.690e-03 69.747 <2e-16 ***
recovered 4.047e-05 1.753e-04 0.231 0.817
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
Residual standard error: 54470 on 121406 degrees of freedom
Multiple R-squared: 0.05404, Adjusted R-squared: 0.05402
F-statistic: 2312 on 3 and 121406 DF, p-value: < 2.2e-16
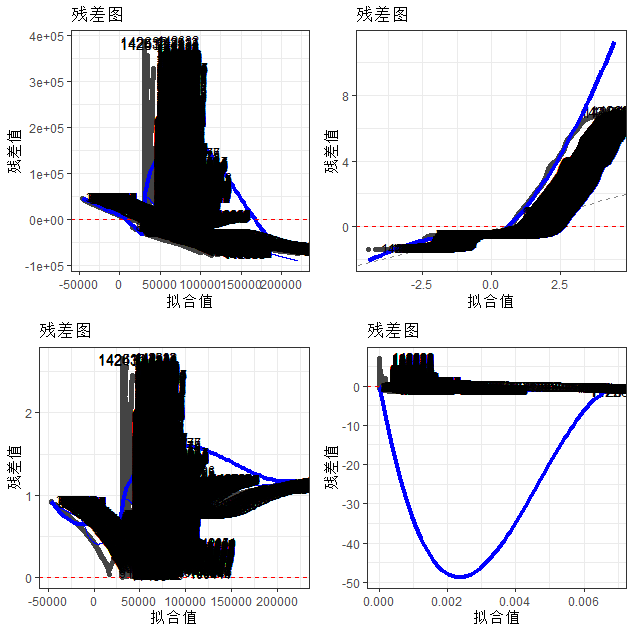 人口数量和死亡人数两个变量与确诊病例数呈负相关,而康复人数对病例数的影响不显著;
人口数量和死亡人数两个变量与确诊病例数呈负相关,而康复人数对病例数的影响不显著;
3.2 偏最小二乘回归(PLSR)模型
如何使用偏最小二乘回归(PLSR)的其他统计模型,并使用交叉验证来评估模型的拟合程度?
install.packages("pls")
library(pls)
# 使用偏最小二乘回归(PLSR)模型
model_pls <- plsr(confirmed_per_million ~ population + deaths + recovered, data = combined_data, ncomp = 3)
# 查看模型摘要
summary(model_pls)
cv_pcr <- pcr(confirmed_per_million ~ Population + deaths + recovered, data = combined_data,
scale = TRUE, validation = "CV")结果展示
Data: X dimension: 121410 3
Y dimension: 121410 1
Fit method: kernelpls
Number of components considered: 3
TRAINING: % variance explained
1 comps 2 comps 3 comps
X 99.9907 100.000 100.000
confirmed_per_million 0.5092 1.624 5.404
从医学的角度来看,这个结果表明,在考虑与COVID-19相关的人口、死亡和康复指标时,研究人员需要考虑多个预测变量,而单独考虑其中任何一个变量的预测能力都很低。这反映了COVID-19作为复杂疾病的本质,其病情和传播受到多个因素的影响。
另外,该模型的结果也显示,无论使用多少个主成分,这个模型都具有很高的预测能力,可以有效地解释大部分输入数据中的方差。这与COVID-19研究的目标非常吻合,即通过对多个指标进行建模,以预测患者的风险和预后,并为疾病管理和应对提供指导。
3.3 画图寻找特征和规律
#install.packages("tidyverse")
library(tidyverse)
# install.packages("viridis")
library(viridis)
# 对中国、意大利和美国的每日感染病例数进行可视化
china_df <- combined_data %>%
filter(Country.Region == "China") %>%
mutate(new_cases = confirmed_cases - lag(confirmed_cases, default = 0))
italy_df <- combined_data %>%
filter(Country.Region == "Italy") %>%
mutate(new_cases = confirmed_cases - lag(confirmed_cases, default = 0))
us_df <- combined_data %>%
filter(Country.Region == "US") %>%
mutate(new_cases = confirmed_cases - lag(confirmed_cases, default = 0))
national_df <- rbind(china_df, italy_df, us_df)
ggplot(national_df, aes(x = date, y = new_cases, colour = Country.Region)) +
geom_line(size = 1) +
theme_minimal() +
labs(title = "每日新增病例数对比", x = "日期", y = "新增病例数")
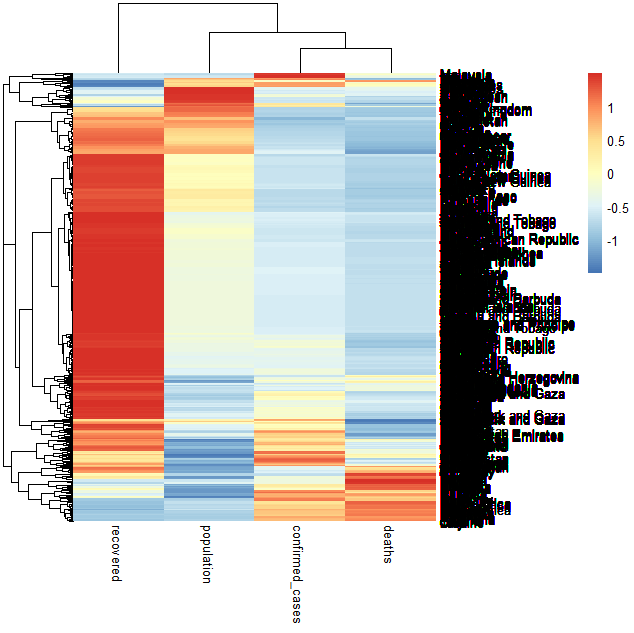
# 使用热力图分析全球各国/地区之间的COVID-19传播情况
# 由于数据量过大,电脑会内存溢出,所以先进行一次过滤
heatmap_df <- combined_data %>% filter(date >= "2021-08-08")
library(pheatmap)
heatmap_df_d <- heatmap_df[,c("confirmed_cases","deaths","recovered","population")]
heatmap_df_scale <- scale(heatmap_df_d)
heatmap_df_scale[is.na(heatmap_df_scale)] <- 0
row.names(heatmap_df_scale) <- heatmap_df$Country.Region
pheatmap(heatmap_df_scale,scale='row')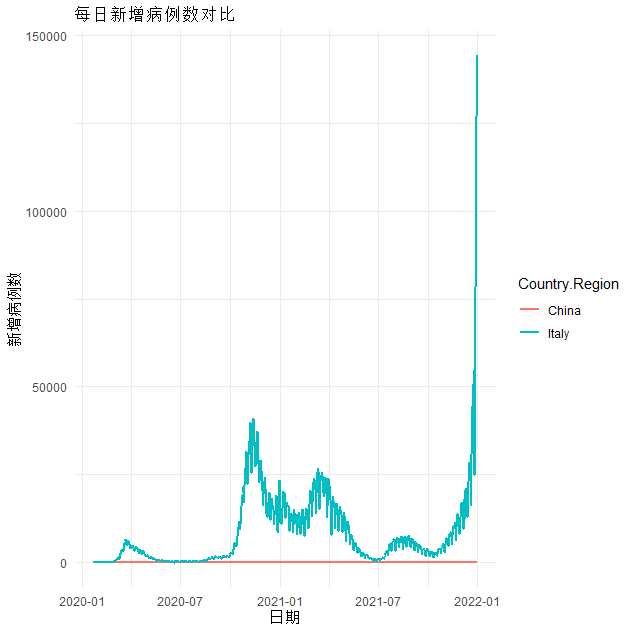
 从图中可以看出,中国每日新增病例趋近于0,意大利前期趋于平稳,后期出现集中爆发的情况,每日新增出现指数级递增的情况。
从图中可以看出,中国每日新增病例趋近于0,意大利前期趋于平稳,后期出现集中爆发的情况,每日新增出现指数级递增的情况。
四、COVID-19预测
4.1 预测未来数据点
在R语言中使用时间序列分析方法来预测未来数据点
install.packages("forecast")
library(forecast)
# 读取预测所需的数据,这里以印度为例
india_df <- combined_data %>%
filter(Country.Region == "India") %>%
select(date, confirmed_cases)
# 将数据设置为时间序列对象
india_ts <- ts(india_df$confirmed_cases, frequency = 7)
# 运用Holt-Winters方法进行预测
hw_model <- hw(india_ts, seasonal = "additive", h = 14)
hw_forecast <- forecast(hw_model, h = 14)
# 绘制预测结果对比图
autoplot(hw_forecast) +
autolayer(india_ts) +
labs(title = "印度COVID-19确诊病例数预测", x = "日期", y = "确诊病例数")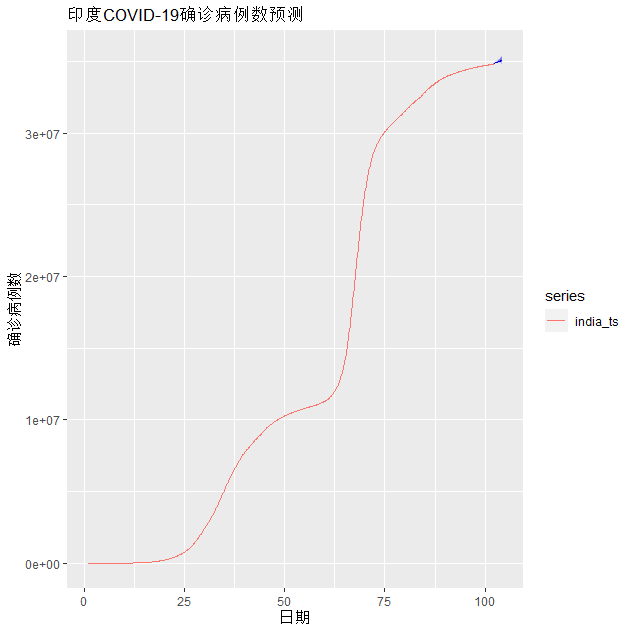 蓝色部分为预测部分,预测出确证病例数会走高,上升会加快。
蓝色部分为预测部分,预测出确证病例数会走高,上升会加快。
4.2 可视化方法
# 对巴西、英国和俄罗斯每日死亡病例数进行可视化
brazil_df <- combined_data %>%
filter(Country.Region == "Brazil") %>%
mutate(new_deaths = deaths - lag(deaths, default = 0))
uk_df <- combined_data %>%
filter(Country.Region == "United Kingdom") %>%
mutate(new_deaths = deaths - lag(deaths, default = 0))
russia_df <- combined_data %>%
filter(Country.Region == "Russia") %>%
mutate(new_deaths = deaths - lag(deaths, default = 0))
national_df <- rbind(brazil_df, uk_df, russia_df)
ggplot(national_df, aes(x = date, y = new_deaths, colour = Country.Region)) +
geom_line(size = 1) +
theme_minimal() +
labs(title = "每日新增死亡病例数对比", x = "日期", y = "新增死亡病例数")
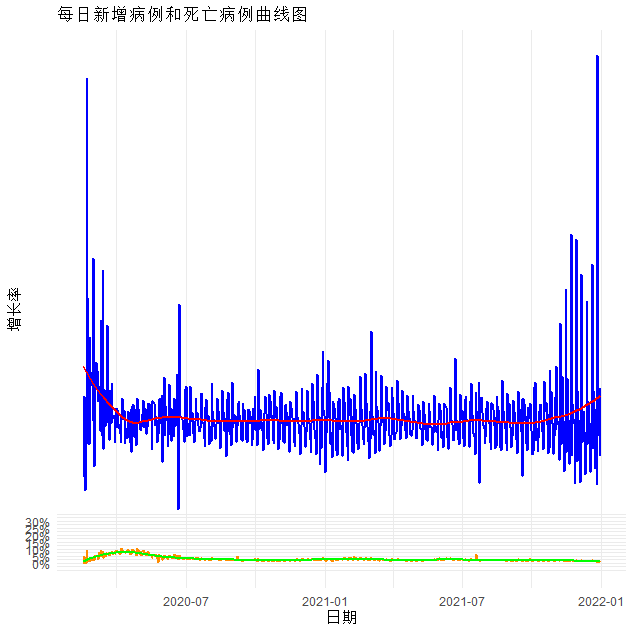
# 绘制每日新增病例数和新增死亡病例数曲线图,并进行拟合
daily_growth_df <- global_new_cases_df %>%
mutate(growth_rate = new_cases / lag(new_cases, default = 1),
death_rate = new_deaths / lag(new_cases, default = 1)) %>%
filter(growth_rate < quantile(growth_rate, 0.999)) %>%
filter(death_rate < quantile(death_rate, 0.999))
ggplot(daily_growth_df, aes(x = date)) +
geom_line(aes(y = growth_rate), colour = "blue", size = 1) +
geom_smooth(aes(y = growth_rate), method = "loess", se = FALSE, span = 0.2, colour = "red") +
geom_line(aes(y = death_rate), colour = "darkorange", size = 1) +
geom_smooth(aes(y = death_rate), method = "loess", se = FALSE, span = 0.2, colour = "green") +
labs(title = "每日新增病例和死亡病例曲线图", x = "日期", y = "增长率") +
theme_minimal() +
scale_y_continuous(breaks = seq(0, 0.3, by = 0.05), labels = scales::percent) +
theme(legend.position = c(0.2, 0.85))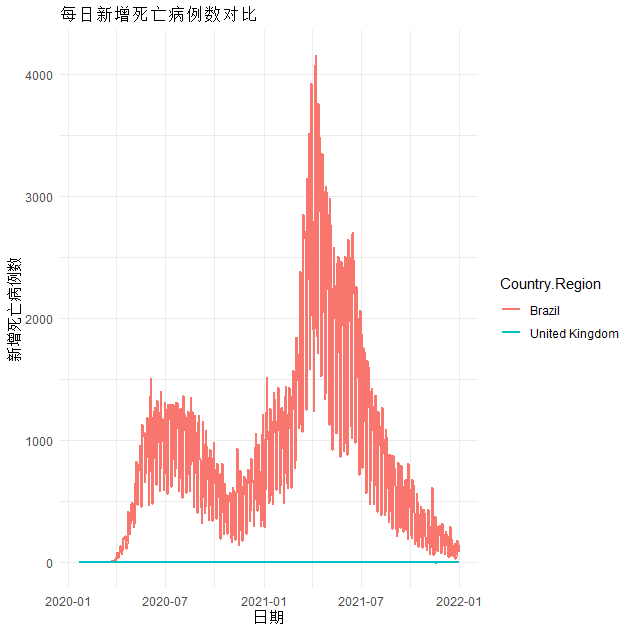
在这篇文章中,我们讨论了如何在R语言中分析COVID-19相关数据,从数据清洗和准备、统计分析到预测建模展开了一系列的步骤。
通过这些步骤,我们可以了解COVID-19感染率、死亡率以及人口特征的相关性,以及了解各因素对COVID-19感染率和死亡率的影响。同时,我们还介绍了使用统计绘图工具,如散点图和热力图,在数据中找出隐藏的特征和规律的方法。
最后,我们还学习了如何使用时间序列分析方法来预测未来数据点,并介绍了如何使用可视化方法更好地展示数据的变化趋势。这些方法可以帮助我们更好地了解COVID-19的传播和流行情况,从而帮助人们应对和控制COVID-19的传播。 ✔