一. sub-subXXX文件夹 + sub-subXXX.html
二. sub-subXXX文件夹
sub-sub097 /
anat /
figures /
func /
log /
anat /
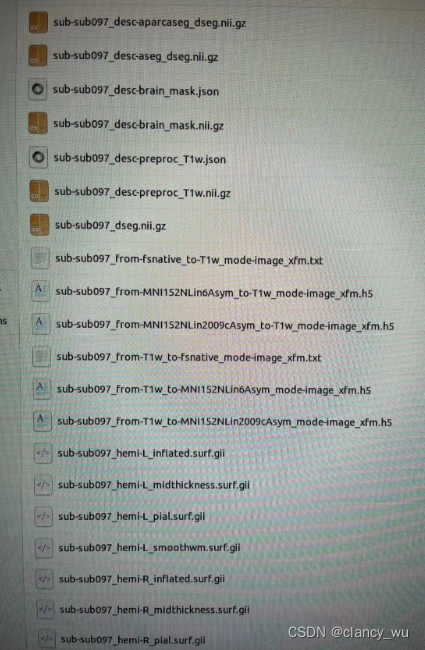
anat文件夹内文件比较多,文件命名规则遵守BIDS要求( https://bids-specification.readthedocs.io/en/stable/05-derivatives/01-introduction.html).
- desc-aparcaseg_dseg.nii.gz 皮层+皮层下分割(以皮层为主)
- desc-aseg_dseg.nii.gz 皮层下分割
- desc-brain_mask.nii.gz 脑罩 / 掩码
- desc-preproc_T1w.nii.gz 没有去头骨的T1加权像
- desc.nii.gz
- label-CFS_probseg.nii.gz 分割的脑脊液
- label-GM_probseg.nii.gz 分割的灰质
- label-WM_probseg.nii.gz 分割的白质
这些都是预处理的过程文件,是在被试原始空间。 - from-fsnative_to-T1w_mode-image_xfm.txt
- from-MNI152NLin6Asym_to-T1w_mode-image_xfm.h5
- from-MNI152NLin2009cAsym_to-T1w_mode-image_xfm.h5
- from-T1w_to-fsnative_mode-image_xfm.txt
- from-T1w_to-MNI152NLin6Asym_mode-image_xfm.h5
- from-T1w_to-MNI152NLin2009cAsym_mode-image_xfm.h5
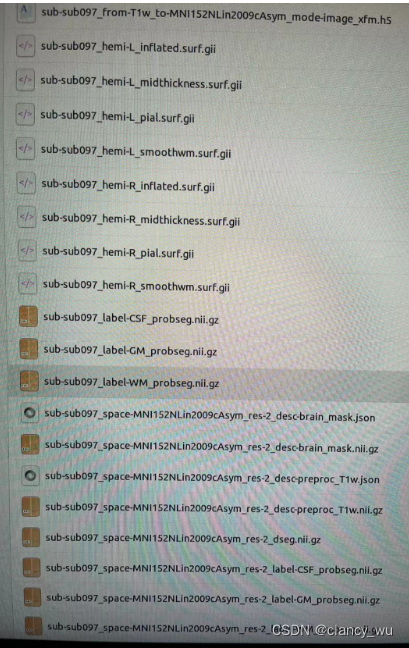
这些是被试空间到原始空间,原始空间到被试空间的转换。2009c是freesurfer的模板,6是FSL的模板。 - hemi-L_inflated.surf.gii
- hemi-L_midthickness.surf.gii
- hemi-L_pial.surf.gii
- hemi-L_smoothwm.surf.gii
这些是被试原始空间的皮层数据。(from-fsnative_to-T1w_mode-image_xfm.txt等是转换矩阵)。 - space-MNI152NLin2009cAsym_res-2_desc-brain_mask.nii.gz
- space-MNI152NLin2009cAsym_res-2_desc-preproc_T1w.nii,gz
- space-MNI152NLin2009cAsym_res-2_desc.nii.gz
- space-MNI152NLin2009cAsym_res-2_label-CSF_probseg.nii.gz
- space-MNI152NLin2009cAsym_res-2_label-GM_probseg.nii.gz
- space-MNI152NLin2009cAsym_res-2_label-WM_probseg.nii.gz
这里sapce就是MNI152NLin2009cAsym空间,而res-2是resolution 2mm分辨率的意思。
func/ 文件夹
- space-MNI152NLin2009cAsym_res-2_boldref.nii.gz 标准空间目标分辨率bold参考像
- space-MNI152NLin2009cAsym_res-2_desc-aparcaseg_dseg.nii.gz
- space-MNI152NLin2009cAsym_res-2_desc-aseg_dseg.nii.gz
- space-MNI152NLin2009cAsym_res-2_desc-preproc_bold.nii.gz
这些都是标准空间MNI152NLin2009cAsym和目标分辨率2mm的预处理数据
Freesurfer文件夹输出
Recon-all 从三维体积重建二维皮质表面。从 MRI 扫描仪收集的图像是三维块,这些图像通过 recon-all 转换为光滑、连续的二维表面。
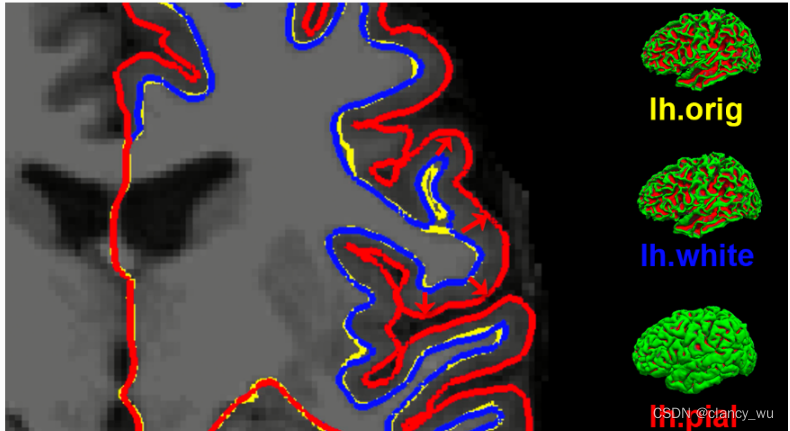
Recon-all 首先从解剖图像中剥离头骨以生成名为brainmask.mgz,估计两个半球的白质和灰质之间的界面存储在名为lh.orig和rh.orig的文件中。这个初始估计经过改进,然后保存到名为lh.white和rh.white的文件中。然后将此边界用作基础,生成软脑膜表面数据lh.pial和rh.pial。

为了更容易地看到激活图位于脑沟岸和脑回脊的位置,可以进一步生成膨胀球体lh.inflated和rh.inflated数据集。
freesurfer文件夹输出包括:label, mri, scripts, stats, surf, temp, touch, trash。
最后三个基本不用管:temp, touch, trash。
label文件夹放的是atlas地图集,可以简单理解为ROI。
mri文件夹放的是volume级别的数据。例如mask脑罩,去除头骨的T1像,皮层下分割等。
scripts文件夹放的是运行recon-all过程中的所有log,可以在这里查看recon-all是否存在异常。
stats文件夹放的是结构分割的统计,例如皮层分割(parcellation)的厚度或体积以及皮层下分割(segmentation)的厚度或体积。
值得注意的是,aseg里面的丘脑、海马等体积是错误的,建议不要使用。如果要分析这类,应该单独进行体积生成
surf文件夹放的是皮层文件夹。
所有输出文件data都是在被试原始空间进行的,即ref fsaverage。如果要cross subjects对比,可以使用较低一点的fsaverage模板,如fsaverage5,可以使用 mri_surf2surf / mri_label2label,或者R中也可以实现。
DPABISurf 7.0 中的处理过程:
fastfreesurfer: %!docker run -ti --rm -v /mnt/Data45/RfMRILab/yan/YAN_Work/DPABIUpdating/Program/DPABI_V6.1_220101/DPABISurf/FreeSurferLicense/license.txt:/opt/freesurfer/license.txt -v /mnt/Data45/RfMRILab/yan/YAN_Work/DPABIUpdating/DPABISurf/FastSurferDev/Work/datatest2:/data cgyan/dpabidev /opt/fastsurfer/run_fastsurfer.sh --fs_license /opt/freesurfer/license.txt --t1 /data/BIDS/sub-Sub001/anat/sub-Sub001_T1w.nii --sid sub-Sub001 --sd /data/freesurfer --threads 1
fmriprep:
皮层下分割,mgz2niigz:‘%s parallel -j %g mri_convert %s/freesurfer/{1}/mri/rh.hippoAmygLabels.HBT.FSvoxelSpace.mgz %s/Results/AnatVolu/T1wSpace/{1}/{1}_Subregions_rh.hippoAmygLabels.HBT.FSvoxelSpace.nii.gz ::: %s’, …
DirFile=dir(fullfile(Cfg.WorkingDir,‘Results’,‘AnatVolu’,‘MNISpace’,Cfg.SubjectID{i},[Cfg.SubjectID{i},‘*space-MNI152NLin2009cAsymlabel-WM_probseg.nii’]));
File=fullfile(Cfg.WorkingDir,‘Results’,‘AnatVolu’,‘MNISpace’,Cfg.SubjectID{i},DirFile(1).name);
结构像:(详见y_Organize_fmriprep.asv)
标准空间:
‘%s parallel -j %g mri_surf2surf
–srcsubject {1}
–trgsubject fsaverage
–hemi lh
–sval %s/freesurfer/{1}/surf/lh.thickness
–tval %s/Results/AnatSurfLH/fsaverage/Thickness/{1}_space-fsaverage_hemi-L.thickness.gii ::: %s’,
CommandInit, Cfg.ParallelWorkersNumber, WorkingDir, WorkingDir, SubjectIDString
原始空间 native space:
Thicnkness:
‘%s parallel -j %g mris_convert
-c %s/freesurfer/{1}/surf/lh.thickness
%s/freesurfer/{1}/surf/lh.white
%s/Results/AnatSurfLH/fsnative/Thickness/{1}_space-fsnative_hemi-L.thickness.gii ::: %s’,
CommandInit, Cfg.ParallelWorkersNumber, WorkingDir, WorkingDir, WorkingDir, SubjectIDString
Area:
‘%s parallel -j %g mris_convert
-c %s/freesurfer/{1}/surf/lh.area
%s/freesurfer/{1}/surf/lh.white
%s/Results/AnatSurfLH/fsnative/Area/{1}_space-fsnative_hemi-L.area.gii ::: %s’,
CommandInit, Cfg.ParallelWorkersNumber, WorkingDir, WorkingDir, WorkingDir, SubjectIDString
Curv:
‘%s parallel -j %g mris_convert
-c %s/freesurfer/{1}/surf/lh.curv
%s/freesurfer/{1}/surf/lh.white
%s/Results/AnatSurfLH/fsnative/Curv/{1}_space-fsnative_hemi-L.curv.gii ::: %s’,
CommandInit, Cfg.ParallelWorkersNumber, WorkingDir, WorkingDir, WorkingDir, SubjectIDString
**解释:mris_convert是freesurfer的函数,其用法为 ** mris_convert [-c curvinfilepath] [-o origfilepath] [-p] [-t] infilepath outfilepath。正如我们之前讲的,lh.orig(黄色部分)是最开始的粗略估计,计算半球白质和灰质之间的界限在哪里,然后在此基础上,进行再一次改善的估计,就是lh.white(蓝色部分),这就是完成的白质边界,然后最后根据这个搜索灰质边缘,生成第三个,即lh.pial(红色部分)。用这些就能计算各种皮层曲率指标了。
总结:如果是要native space的指标,用mris_convert可以在多个曲率指标间转换。把Area等resample到被试native space的white就是native space的area指标。把Area等resample到fsaverage5等的white就是standard space的area指标。
转换至原始空间:
文件:RefFile=[Cfg.WorkingDir,filesep,‘Masks’,filesep,‘AutoMasks’,filesep,Cfg.SubjectID{i},‘_task-rest_space-T1w_desc-brain_mask.nii.gz’];
转换矩阵:TransformFile=[Cfg.WorkingDir,filesep,‘fmriprep’,filesep,Cfg.SubjectID{i},filesep,‘anat’,filesep,Cfg.SubjectID{i},‘_from-MNI152NLin2009cAsym_to-T1w_mode-image_xfm.h5’];
转换命令(y_WarpByANTs(SourceFile,MaskName,RefFile,TransformFile,Interpolation,Dimensionality,InputImageType,DefaultValue,IsFloat0,DockerName:Fiber-subfunctions)
antsApplyTransforms
–default-value 0
–dimensionality 3
–float 0
–input /DPABI/Templates/HarvardOxford-sub-maxprob-thr25-2mm_YCG.nii
–interpolation MultiLabel
–output /data/Masks/MasksForDwi/T1Space_sub-Sub001_HarvardOxford-sub-maxprob-thr25-2mm_YCG.nii
–reference-image /data/qsiprep/sub-Sub001/dwi/sub-Sub001_space-T1w_desc-preproc_dwi.nii.gz
–transform /data/qsiprep/sub-Sub001/anat/sub-Sub001_from-MNI152NLin2009cAsym_to-T1w_mode-image_xfm.h5
即有了转换矩阵,desc-preproc.T1w.nii.gz,就能把标准图像转换为原始空间图像,用的是antsApplyTransforms命令。
平滑:
DirName=dir(fullfile(Cfg.WorkingDir,[FunSessionPrefixSet{iFunSession},Cfg.StartingDirName],Cfg.SubjectID{1},‘_hemi-L.func.gii’));
命令:mri_surf2surf --s %s --hemi lh --sval %s/%s%s/{1}/{1}%s --fwhm %g --cortex --tval %s/%s%sS/{1}/s{1}%s ::: %s
这个hemi-L*.func.gii都是fsaverage5的标准空间。
计算alff:
mkdir([Cfg.WorkingDir,filesep,FunSessionPrefixSet{iFunSession},‘Results’,filesep,‘FunSurfLH’,filesep,‘ALFF_’,Cfg.StartingDirName]);
DirName=dir(fullfile(Cfg.WorkingDir,[FunSessionPrefixSet{iFunSession},Cfg.StartingDirName],Cfg.SubjectID{i},‘_hemi-L.func.gii’));
这个hemi-L*.func.gii都是fsaverage5的标准空间,计算完成之后,进行改名。
原始空间计算ROI:
mri_surf2surf --srcsubject fsaverage5 --trgsubject {1} --mapmethod nnf --hemi rh --sval %s/Masks/MasksForFun/Masks_fsaverage5Space/%s%s --tval %s/Masks/MasksForFun/Masks_fsnativeSpace/%sMaskFile_%g_{1}_%s.gii ::: %s’, CommandInit, Cfg.ParallelWorkersNumber, WorkingDir,name, ‘.gii’,
结果整理:
mkdir([Cfg.WorkingDir,filesep,Cfg.StartingDirName,‘W’,filesep,‘AnatSurfLH’]);
movefile([Cfg.WorkingDir,filesep,Cfg.StartingDirName,filesep,‘AnatSurfLH’,filesep,‘fsaverage’],[Cfg.WorkingDir,filesep,Cfg.StartingDirName,‘W’,filesep,‘AnatSurfLH’]);
movefile([Cfg.WorkingDir,filesep,Cfg.StartingDirName,filesep,‘AnatSurfLH’,filesep,‘fsaverage5’],[Cfg.WorkingDir,filesep,Cfg.StartingDirName,‘W’,filesep,‘AnatSurfLH’]);
mkdir([Cfg.WorkingDir,filesep,Cfg.StartingDirName,‘W’,filesep,‘AnatVolu’]);
movefile([Cfg.WorkingDir,filesep,Cfg.StartingDirName,filesep,‘AnatVolu’,filesep,‘MNISpace’],[Cfg.WorkingDir,filesep,Cfg.StartingDirName,‘W’,filesep,‘AnatVolu’]);
case {‘FunSurfLH’,‘FunSurfRH’}:mri_surf2surf --srcsubject %s --trgsubject %s --hemi lh --sval %s/%s/FunSurfLH/%s/{1} --tval %s/%sW/FunSurfLH/%s/w{1} ::: %s’。
总结:
fmriprep处理以后,会有anat和func文件夹。
在被试anat文件夹里,大致分为4类文件。第一类是native space的预处理文件。包括aparc+aseg的分割文件,brainmask文件,预处理以后的T1w文件,dseg分割文件,CSF, GM, WM文件。第二类是各种native space到standard space的转换矩阵。第三类是native space的皮层gii文件,如inflated, midthickness, pial等。第四类是标准空间的预处理文件。包括brainmask文件,预处理以后的T1w文件,CSF, GM, WM文件。
在被试func文件夹里,大致分为3类文件。第一类是噪音文件。如AROMAnoiseICs.csv等。第二类是转换文件,即被试的bold空间到T1夹权空间。第三类是fsaverage5的皮层gii文件。第四类是标准空间MNI152的预处理文件,如smoothAROMAnonaggr_bold, boldref, aparcaseg, aseg, brainmask, preproc_bold。
在严老师的DPABI软件中,
- 先根据segmentation的结果,生成mask,存储在Masks/SegmentationMasks/FunSpace_ThrdMask_subXX_WM.nii当中(如果之前没有这个,可以通过fmriprep的desc-brain_mask.nii.gz生成)。或者是根据segmentation的结果,生成标准空间mask,存储在Masks/SegmentationMasks/MNIFunSpace_ThrdMask_subXX_WM.nii当中(如果之前没有这个,可以通过fmriprep的space-MNI152-brain_mask.nii.gz生成)。
除此以外,还单独制作了warp功能,能从T1到MNI的转换,只需要有转换矩阵放进antsApplyTransforms当中。 - 当Masks准备好以后,首先把mask进行重采样resample,生成ALLResampled_XX.nii文件。
- 移除协变量、头动等混淆变量
- 将*_hemi-L*.func.gii使用mri_surf2surf进行平滑(在平滑中,space是根据上一阶段制定的,要么是原始空间——应该是fsaverage native space,我猜的——平滑,要么是fsaverage5空间平滑),然后单独提取到s的文件夹中。
- 计算功能性指标ALFF等指标时,使用第1-2步的皮层空间和体空间的Masks,用*_hemi-L*.func.gii文件进行计算。
- 计算结构像等指标时,使用mri_surf2surf功能,将freesurfer文件夹下的sub001的surf文件夹的lh.thickness作为INPUT,trgsubject为fsaverage或fsaverage5,把被试native surf space对齐到fsaverage当中去,然后移动到Results/AnatSurfRH/fsaverage/Thickness/sub001_space-fsaverage_hemi-R.thickness.gii,或者是Results/AnatSurfRH/fsaverage5/Thickness/sub001_space-fsaverage_hemi-R.thickness.gii
声明,以上开源代码仅供个人学习使用,不用于任何商业行为