1 引言
普通遗传算法(Sample Genetic Algorithm, SGA)存在着严重的缺点,它的Pc和Pm的值是固定的,本文采用自适应遗传算法进行求解TSP问题。不管是优良个体还是劣质个体都经过了相同概率的交叉和变异操作。这会引起两个很严重的问题:
- 相同的概率,这可以说是不公平,因为对于优良个体,我们应该减小交叉变异概率,使之能够尽量保存 ; 而对于劣质个体,我们应该增大交叉变异概率,使之能够尽可能的改变劣质的状况 。所以,一成不变的交叉变异概率影响了算法的效率。
- 相同的概率总不能很好的满足种群进化过程中的需要,比如在迭代初期,种群需要较高的交叉和变异概率,已达到快速寻找最优解的目的,而在收敛后期,种群需要较小的交叉和变异概率,以帮助种群在寻找完最优解后快速收敛。所以,一成不变的交叉变异概率影响了算法的效率。
2 问题描述
TSP问题共有3种数学模型,如下。另有https://blog.csdn.net/Zhang_0702_China/article/details/106983492给出了TSP几种建模方式详细介绍。
- 消除子回路约束模型(本文采用,下图)
- MTZ约束消除子回路模型,Miller-Tucker-Zemlin formulation
- 1-tree模型
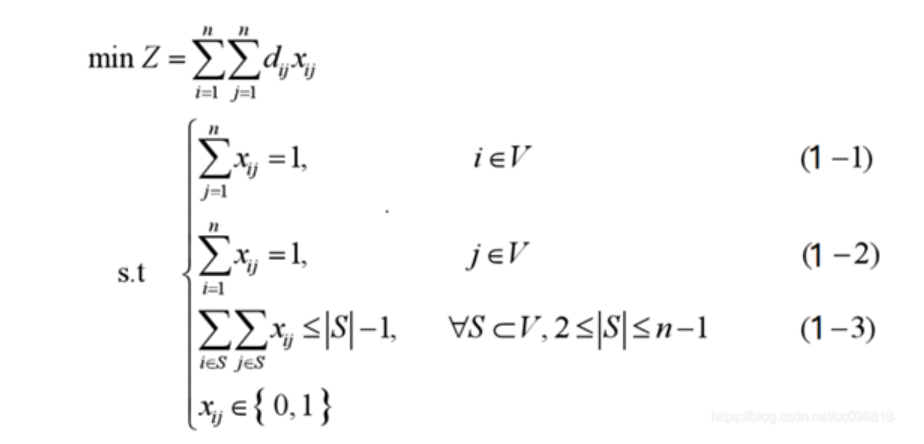
3 遗传算法
3.1初始种群生成
种群规模设置为100,随机城市顺序,采用自然数编码方式构造染色体,用0-19表示20个城市,染色体的编码如图所示,每条染色体表示城市访问顺序。如下图表示为:该旅行商从城市2出发,走到城市8,最后回到城市2。
3.2适应度计算
适应度函数是非负的,任何情况下总希望越大越好。TSP问题的目标函数是总距离越小越好,所以设计适应度函数为
f
i
t
n
e
s
s
=
1
z
fitness=\frac{1}{z}
fitness=z1
3.3 选择操作(精英策略+轮盘赌策略)
精英策略:即选择父代中适应度最大的个体遗传到子代。
轮盘赌选择:计算产生新个体的适应值,根据适应值大小分配个体的概率,根据概率方式选择个体作为下一代种群的父代。基本思想:各个个体被选中的概率与其适应度大小成正比,适应度值越好的个体,被选择的概率就越大。轮盘赌选择步骤如下:
- 计算适应度:适应度为该条染色体(一条可行路径)的总距离的倒数
- 计算每个个体的选择概率:选择概率是个体被遗传到下一代的概率,显然适应度愈大,该个体愈能适应环境,遗传到下一代的概率更高。染色体 i i i的选择概率计算公式为 p i ( s e l e c t ) = f i t n e s s i ∑ i = 1 N f i t n e s s i p_i(select)=\frac{fitness_i}{\sum_{i=1}^N{fitness_i}} pi(select)=∑i=1Nfitnessifitnessi ,其中 i i i代表个体, N N N代表种群规模。
- 计算每个个体的累积概率: p i ( c u l ) = ∑ j = 1 i p j ( s e l e c t ) p_i(cul)=\sum_{j=1}^i{p_j(select)} pi(cul)=∑j=1ipj(select)
- 执行精英策略:在当代种群population中把选择概率最大的个体,直接复制到子代种群offspring,即
offspring[1] = population[argmax(p_select)],其中p_select= { p i ( s e l e c t ) } i = 1 N \{p_i(select)\}_{i=1}^N {pi(select)}i=1N - 执行轮盘赌选择:对每个个体 i i i,在[0, 1]区间生成一个随机数 r i r_i ri,若 p i − 1 ( c u l ) ≤ r i ≤ p i ( c u l ) p_{i-1}(cul) \leq r_i \leq p_i(cul) pi−1(cul)≤ri≤pi(cul),则个体i被选择至下一代。
- 重复步骤5,N-1次。
3.4 交叉算法
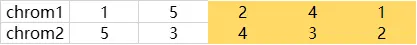
随机选择两条染色体 chrom1和chrom2,确定两个切点cut1和cut2,如cut1=2,cut2=4,则交换chrom1和chrom2的黄色部分。
交换完成后如下图:
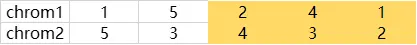
此时,chrom1和chrom2都含有重复的基因,chrom1的基因0(城市1)重复,chrom2的基因1(城市3)重复(即绿色部分)

将绿色部分调整,一个思路是采用映射表。调整过程如下:
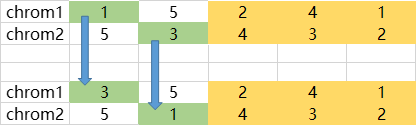
黄色部分(交叉片段)即为映射表(如下),即检查为参与交叉的基因,若该基因在映射表中,则按映射表进行互换。如上如chrom1中,城市1和城市5为非交叉基因,城市1在映射表中需替换为城市2,又城市2需替换为城市4,又城市4需替换为城市3。最终城市1替换为城市3。
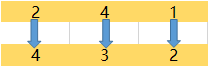
3.5 变异算法
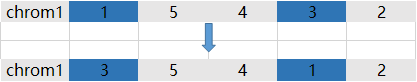
采用2-opt方法,即选择染色体,随机选择2个位置,将这两个位置的基因交换,如下图。
3.6 停止准则
停止准则有3种,本文选择第三种。
- 当最优个体的适应度达到给定的阈值
- 最优个体的适应度和群体适应度不再上升
- 迭代次数达到预设的代数时,算法终止(本文采用)
4 运行结果
运行一把迭代10000次结果如下,800多就逼近最优解:
Iteration: 0 path: [11, 17, 4, 2, 15, 10, 14, 18, 7, 12, 19, 8, 9, 5, 3, 1, 6, 16, 13, 0] distance: 1729.0
Iteration: 200 path: [15, 18, 17, 14, 11, 8, 4, 2, 9, 12, 13, 10, 7, 5, 3, 1, 6, 16, 19, 0] distance: 1122.0
Iteration: 400 path: [15, 18, 17, 14, 11, 8, 4, 2, 9, 12, 19, 16, 13, 10, 6, 1, 3, 7, 5, 0] distance: 893.0
Iteration: 600 path: [15, 18, 17, 14, 11, 8, 4, 5, 9, 12, 19, 16, 13, 10, 6, 1, 3, 7, 2, 0] distance: 887.0
Iteration: 800 path: [15, 18, 17, 14, 11, 8, 4, 5, 9, 12, 19, 16, 13, 10, 6, 1, 3, 7, 2, 0] distance: 887.0
Iteration: 1000 path: [15, 18, 17, 14, 11, 8, 4, 9, 7, 12, 19, 16, 13, 10, 6, 1, 3, 5, 2, 0] distance: 879.0
Iteration: 1200 path: [15, 18, 17, 14, 11, 8, 4, 9, 7, 12, 19, 16, 13, 10, 6, 1, 3, 5, 2, 0] distance: 879.0
Iteration: 1400 path: [15, 18, 17, 14, 11, 8, 4, 9, 7, 12, 19, 16, 13, 10, 6, 1, 3, 5, 2, 0] distance: 879.0
Iteration: 1600 path: [15, 18, 17, 14, 11, 8, 4, 9, 7, 12, 19, 16, 13, 10, 6, 1, 3, 5, 2, 0] distance: 879.0
Iteration: 1800 path: [15, 18, 17, 14, 11, 8, 4, 9, 7, 12, 19, 16, 13, 10, 6, 1, 3, 5, 2, 0] distance: 879.0
Iteration: 2000 path: [15, 18, 17, 14, 11, 8, 4, 9, 7, 12, 19, 16, 13, 10, 6, 1, 3, 5, 2, 0] distance: 879.0
Iteration: 2200 path: [15, 18, 17, 14, 11, 8, 4, 9, 7, 12, 19, 16, 13, 10, 6, 1, 3, 5, 2, 0] distance: 879.0
Iteration: 2400 path: [15, 18, 17, 14, 11, 8, 4, 9, 7, 12, 19, 16, 13, 10, 6, 1, 3, 5, 2, 0] distance: 879.0
Iteration: 2600 path: [15, 18, 17, 14, 11, 8, 4, 9, 7, 12, 19, 16, 13, 10, 6, 1, 3, 5, 2, 0] distance: 879.0
Iteration: 2800 path: [15, 18, 17, 14, 11, 8, 4, 9, 7, 12, 19, 16, 13, 10, 6, 1, 3, 5, 2, 0] distance: 879.0
Iteration: 3000 path: [15, 18, 17, 14, 11, 8, 4, 9, 7, 12, 19, 16, 13, 10, 6, 1, 3, 5, 2, 0] distance: 879.0
Iteration: 3200 path: [15, 18, 17, 14, 11, 8, 4, 9, 7, 12, 19, 16, 13, 10, 6, 1, 3, 5, 2, 0] distance: 879.0
Iteration: 3400 path: [15, 18, 17, 14, 11, 8, 4, 9, 7, 12, 19, 16, 13, 10, 6, 1, 3, 5, 2, 0] distance: 879.0
Iteration: 3600 path: [15, 18, 17, 14, 11, 8, 4, 9, 7, 12, 19, 16, 13, 10, 6, 1, 3, 5, 2, 0] distance: 879.0
Iteration: 3800 path: [15, 18, 17, 14, 11, 8, 4, 9, 7, 12, 19, 16, 13, 10, 6, 1, 3, 5, 2, 0] distance: 879.0
Iteration: 4000 path: [15, 18, 17, 14, 11, 8, 4, 9, 7, 12, 19, 16, 13, 10, 6, 1, 3, 5, 2, 0] distance: 879.0
Iteration: 4200 path: [15, 18, 17, 14, 11, 8, 4, 9, 7, 12, 19, 16, 13, 10, 6, 1, 3, 5, 2, 0] distance: 879.0
Iteration: 4400 path: [15, 18, 17, 14, 11, 8, 4, 9, 7, 12, 19, 16, 13, 10, 6, 1, 3, 5, 2, 0] distance: 879.0
Iteration: 4600 path: [15, 18, 17, 14, 11, 8, 4, 9, 7, 12, 19, 16, 13, 10, 6, 1, 3, 5, 2, 0] distance: 879.0
Iteration: 4800 path: [15, 18, 17, 14, 11, 8, 4, 9, 7, 12, 19, 16, 13, 10, 6, 1, 3, 5, 2, 0] distance: 879.0
Iteration: 5000 path: [15, 18, 17, 14, 11, 8, 4, 9, 7, 12, 19, 16, 13, 10, 6, 1, 3, 5, 2, 0] distance: 879.0
Iteration: 5200 path: [15, 18, 17, 14, 11, 8, 4, 9, 7, 12, 19, 16, 13, 10, 6, 1, 3, 5, 2, 0] distance: 879.0
Iteration: 5400 path: [15, 18, 17, 14, 11, 8, 4, 9, 7, 12, 19, 16, 13, 10, 6, 1, 3, 5, 2, 0] distance: 879.0
Iteration: 5600 path: [15, 18, 17, 14, 11, 8, 4, 9, 7, 12, 19, 16, 13, 10, 6, 1, 3, 5, 2, 0] distance: 879.0
Iteration: 5800 path: [15, 18, 17, 14, 11, 8, 4, 9, 7, 12, 19, 16, 13, 10, 6, 1, 3, 5, 2, 0] distance: 879.0
Iteration: 6000 path: [15, 18, 17, 14, 11, 8, 4, 9, 7, 12, 19, 16, 13, 10, 6, 1, 3, 5, 2, 0] distance: 879.0
Iteration: 6200 path: [15, 18, 17, 14, 11, 8, 4, 9, 7, 12, 19, 16, 13, 10, 6, 1, 3, 5, 2, 0] distance: 879.0
Iteration: 6400 path: [15, 18, 17, 14, 11, 8, 4, 9, 7, 12, 19, 16, 13, 10, 6, 1, 3, 5, 2, 0] distance: 879.0
Iteration: 6600 path: [15, 18, 17, 14, 11, 8, 4, 9, 7, 12, 19, 16, 13, 10, 6, 1, 3, 5, 2, 0] distance: 879.0
Iteration: 6800 path: [15, 18, 17, 14, 11, 8, 4, 9, 7, 12, 19, 16, 13, 10, 6, 1, 3, 5, 2, 0] distance: 879.0
Iteration: 7000 path: [15, 18, 17, 14, 11, 8, 4, 9, 7, 12, 19, 16, 13, 10, 6, 1, 3, 5, 2, 0] distance: 879.0
Iteration: 7200 path: [15, 18, 17, 14, 11, 8, 4, 9, 7, 12, 19, 16, 13, 10, 6, 1, 3, 5, 2, 0] distance: 879.0
Iteration: 7400 path: [15, 18, 17, 14, 11, 8, 4, 9, 7, 12, 19, 16, 13, 10, 6, 1, 3, 5, 2, 0] distance: 879.0
Iteration: 7600 path: [15, 18, 17, 14, 11, 8, 4, 9, 7, 12, 19, 16, 13, 10, 6, 1, 3, 5, 2, 0] distance: 879.0
Iteration: 7800 path: [15, 18, 17, 14, 11, 8, 4, 9, 7, 12, 19, 16, 13, 10, 6, 1, 3, 5, 2, 0] distance: 879.0
Iteration: 8000 path: [15, 18, 17, 14, 11, 8, 4, 9, 7, 12, 19, 16, 13, 10, 6, 1, 3, 5, 2, 0] distance: 879.0
Iteration: 8200 path: [15, 18, 17, 14, 11, 8, 4, 9, 7, 12, 19, 16, 13, 10, 6, 1, 3, 5, 2, 0] distance: 879.0
Iteration: 8400 path: [15, 18, 17, 14, 11, 8, 4, 9, 7, 12, 19, 16, 13, 10, 6, 1, 3, 5, 2, 0] distance: 879.0
Iteration: 8600 path: [15, 18, 17, 14, 11, 8, 4, 9, 7, 12, 19, 16, 13, 10, 6, 1, 3, 5, 2, 0] distance: 879.0
Iteration: 8800 path: [15, 18, 17, 14, 11, 8, 4, 9, 7, 12, 19, 16, 13, 10, 6, 1, 3, 5, 2, 0] distance: 879.0
Iteration: 9000 path: [15, 18, 17, 14, 11, 8, 4, 9, 7, 12, 19, 16, 13, 10, 6, 1, 3, 5, 2, 0] distance: 879.0
Iteration: 9200 path: [15, 18, 17, 14, 11, 8, 4, 9, 7, 12, 19, 16, 13, 10, 6, 1, 3, 5, 2, 0] distance: 879.0
Iteration: 9400 path: [15, 18, 17, 14, 11, 8, 4, 9, 7, 12, 19, 16, 13, 10, 6, 1, 3, 5, 2, 0] distance: 879.0
Iteration: 9600 path: [15, 18, 17, 14, 11, 8, 4, 9, 7, 12, 19, 16, 13, 10, 6, 1, 3, 5, 2, 0] distance: 879.0
Iteration: 9800 path: [15, 18, 17, 14, 11, 8, 4, 9, 7, 12, 19, 16, 13, 10, 6, 1, 3, 5, 2, 0] distance: 879.0
Iteration: 10000 path: [15, 18, 17, 14, 11, 8, 4, 9, 7, 12, 19, 16, 13, 10, 6, 1, 3, 5, 2, 0] distance: 879.0
time used: 1s
程序
- GeneticAlgorithm遗传算法
- Main运行
- Instance读取文件(20个城市的坐标,)
- Util类:一些工具方法
package geneticalgorithm.algorithm;
import geneticalgorithm.util.Util;
import geneticalgorithm.tsp.TSP;
import java.util.*;
public class GeneticAlgorithm {
TSP tsp;
//距离矩阵
double[][] distance;
//种群规模
int size;
//基因数量
int gene;
//父代种群
int[][] parent;
//适应度
double[] fitness;
//子代种群
int[][] child;
//最大迭代次数
private final int GEN;
//交叉概率
private double pcrossover;
//变异概率
private double pmutation;
public GeneticAlgorithm(String fileName, int GEN) {
tsp = new TSP(fileName);
distance = tsp.distance;
pcrossover = 0.;
pmutation = 0.;
this.GEN = GEN;
}
public void initialize(int size) {
this.size = size;
gene = distance.length;
parent = new int[size][gene];
fitness = new double[size];
child = new int[size][gene];
}
private void initializePopulation() {
int size = parent.length;
for (int i = 0; i < size; i++) {
parent[i] = Util.randArr(gene);
}
}
//适应度函数
private void fitness() {
for (int i = 0; i < parent.length; i++)
fitness[i] = 1000000.0 / getLen(parent[i]);
updateProbality();
}
//更新交叉概率、变异概率
private void updateProbality() {
//最大适应度
double fmax = Util.max(fitness);
//平均适应度
double favg = Util.sum(fitness) / size;
pcrossover = 100 / (fmax - favg);
pmutation = 100 / (fmax - favg);
}
/**
* @param chrom 染色体
* @return 一条染色体的总距离
*/
private double getLen(int[] chrom) {
double len = 0;
for (int k = 0; k < chrom.length - 1; k++) {
int i = chrom[k];
int j = chrom[k + 1];
len += distance[i][j];
}
len = len + distance[chrom[chrom.length - 1]][0];
return len;
}
//选择操作
private void select() {
eliteSelect();
routtleSelect();
}
//精英选择1条染色体
private void eliteSelect() {
int elite = Util.getMaxIndex(fitness);
updatePopulation(parent, child, elite, 0);
}
//轮盘赌选择99条染色体
private void routtleSelect() {
double[] cumul = cumulativep();
// System.out.println(Arrays.toString(cumulativep()));
for (int i = 1; i < parent.length; i++) {
double randDouble = Math.random();
int selected = 0;
for (int j = 0; j < cumul.length; j++) {
if (randDouble < cumul[j]) {
selected = j;//选择第j条染色体
break;
}
}
updatePopulation(parent, child, selected, i);
}
}
//更新种群
private void updatePopulation(int[][] parent, int[][] child, int selected, int i) {
System.arraycopy(parent[selected], 0, child[i], 0, gene);
}
//累积概率
private double[] cumulativep() {
double[] cumulativep = new double[size];
double[] slectionp = slectionp();
for (int i = 0; i < size; i++) {
double res = 0.;
for (int j = 0; j <= i; j++) {
res += slectionp[j];
}
cumulativep[i] = res;
}
return cumulativep;
}
//选择概率
private double[] slectionp() {
double[] selectionp = new double[size];
for (int i = 0; i < size; i++)
selectionp[i] = fitness[i] / Util.sum(fitness);
return selectionp;
}
public void cross() {
int ch1 = Util.randInt(size - 1) + 1;
int ch2 = Util.randInt(size - 1) + 1;//不选择精英个体
int[] chrom1 = child[ch1];
int[] chrom2 = child[ch2];
cross(chrom1, chrom2);
}
private void cross(int[] chrom1, int[] chrom2) {
int cut1 = Util.randInt(gene);
int cut2 = Util.randInt(cut1, gene - 1);
int[][] map = new int[cut2 - cut1 + 1][2];
swap(chrom1, chrom2, map, cut1, cut2);
adjust(chrom1, chrom2, map, cut1, cut2);
}
private void swap(int[] chrom1, int[] chrom2, int[][] map, int cut1, int cut2) {
for (int i = cut1; i <= cut2; i++) {
map[i - cut1][0] = chrom1[i];
map[i - cut1][1] = chrom2[i];
chrom1[i] = map[i - cut1][1];
chrom2[i] = map[i - cut1][0];
}
for (int i = 0; i < map.length; i++) {
for (int j = 0; j < map.length; j++) {
if (map[i][0] == map[j][1]) {
map[i][0] = map[j][0];
map[j][1] = map[i][1];
map[i][0] = map[i][1] = -1;
}
}
}
}
private static void adjust(int[] chrom1, int[] chrom2, int[][] map, int cut1, int cut2) {
for (int i = 0; i < chrom1.length; i++) {
if (i < cut1 || i > cut2)
for (int[] ints : map) {
if (chrom1[i] == ints[1]) chrom1[i] = ints[0];
if (chrom2[i] == ints[0]) chrom2[i] = ints[1];
}
}
}
public void mutate() {
//i从1开始 保留精英
for (int i = 1; i < child.length; i++) {
int r1 = Util.randInt(20);//产生[0,20)的随机整数
int r2 = Util.randInt(20);
int gene1 = child[i][r1];
int gene2 = child[i][r2];
child[i][r1] = gene2;
child[i][r2] = gene1;
}
}
public void runGeneticAlgorithm() {
initializePopulation();
fitness();
long start = System.currentTimeMillis();
for (int gen = 0; gen <= GEN; gen++) {
select();
if (Math.random() < pcrossover) cross();
if (Math.random() < pmutation) mutate();
for (int i = 0; i < child.length; i++) parent[i] = child[i].clone();
for (int i = 0; i < child.length; i++) {
if (Util.isRepetition(child[i])) {
System.out.println(Arrays.toString(child[i]));
throw new IllegalArgumentException("repetition!");
}
}
fitness();
displayInfo(gen);
}
long end = System.currentTimeMillis();
System.out.printf("time used: %ss", (end - start) / 1000);
}
private void displayInfo(int counter) {
if (counter % 200 == 0) {
int ch = Util.getMaxIndex(fitness);
System.out.println();
System.out.printf("Iteration: %s\t", counter);
System.out.printf(" path: %s\t", Arrays.toString(child[ch]));
System.out.printf(" distance: %s", getLen(child[ch]));
System.out.println();
}
}
}
package geneticalgorithm.tsp;
import java.io.BufferedReader;
import java.io.FileReader;
import java.io.IOException;
public class Instance {
public double[][] readFile(String filename) {
double[] x = new double[20];
double[] y = new double[20];
double[][] distance = new double[x.length][y.length];
try (BufferedReader bf = new BufferedReader(new FileReader(filename))) {
int counter = 0;
String line;
while ((line = bf.readLine()) != null) {
String[] coordinate = line.split(",");
x[counter] = Double.parseDouble(coordinate[0]);
y[counter] = Double.parseDouble(coordinate[1]);
counter += 1;
}
} catch (IOException e) {
System.out.println("File not found!");
System.exit(-1);
}
for (int i = 0; i < x.length; i++) {
distance[i][i] = 0;
for (int j = 0; j < y.length; j++) {
double e1 = Math.abs((x[i] - x[j]));
double e2 = Math.abs((y[i] - y[j]));
distance[i][j] = distance[j][i] = (int) Math.sqrt(e1 * e1 + e2 * e2);
}
}
return distance;
}
}
package geneticalgorithm.tsp;
import java.io.BufferedReader;
import java.io.FileReader;
import java.io.IOException;
public class Instance {
public double[][] readFile(String filename) {
double[] x = new double[20];
double[] y = new double[20];
double[][] distance = new double[x.length][y.length];
try (BufferedReader bf = new BufferedReader(new FileReader(filename))) {
int counter = 0;
String line;
while ((line = bf.readLine()) != null) {
String[] coordinate = line.split(",");
x[counter] = Double.parseDouble(coordinate[0]);
y[counter] = Double.parseDouble(coordinate[1]);
counter += 1;
}
} catch (IOException e) {
System.out.println("File not found!");
System.exit(-1);
}
for (int i = 0; i < x.length; i++) {
distance[i][i] = 0;
for (int j = 0; j < y.length; j++) {
double e1 = Math.abs((x[i] - x[j]));
double e2 = Math.abs((y[i] - y[j]));
distance[i][j] = distance[j][i] = (int) Math.sqrt(e1 * e1 + e2 * e2);
}
}
return distance;
}
}
package geneticalgorithm.Main;
import geneticalgorithm.algorithm.GeneticAlgorithm;
public class Main {
public static void main(String[] args) {
String fileName = "D:\\Users\\36297\\JavaWorkspace\\algori\\src\\tsp\\geneticalgori\\data.txt";
GeneticAlgorithm ga = new GeneticAlgorithm(fileName,10000);
ga.initialize(500);
ga.runGeneticAlgorithm();
}
}
package geneticalgorithm.util;
import java.util.Arrays;
import java.util.HashSet;
import java.util.Random;
import java.util.Set;
import java.util.concurrent.ThreadLocalRandom;
public class Util {
/**
* @param n [0-n]长度
* @return [0-n]范围不重复的随机数组
*/
public static int[] randArr(int n) {
return ThreadLocalRandom.current().ints(0, n).distinct().limit(n).toArray();
}
/**
* @param array 一维数组
* @return maximum element of an integer array
*/
public static int max(int[] array) {
return Arrays.stream(array).max().getAsInt();
}
/**
* @param array integer array
* @return index of the max
*/
public static int getMaxIndex(int[] array) {
double max = max(array);
for (int i = 0; i < array.length; i++)
if (max == array[i]) return i;
return -1;
}
/**
* @param array 一维数组
* @return maximum element of an double array
*/
public static double max(double[] array) {
return Arrays.stream(array).max().getAsDouble();
}
/**
* @param array double array
* @return index of the max
*/
public static int getMaxIndex(double[] array) {
double max = max(array);
for (int i = 0; i < array.length; i++)
if (max == array[i]) return i;
return -1;
}
/**
* @param array double array
* @return 数组求和
*/
public static double sum(double[] array) {
return Arrays.stream(array).sum();
}
/**
* @param index [0,index)
* @return 返回[0, index)的一个随机整数
*/
public static int randInt(int index) {
return new Random().nextInt(index);
}
/**
* @param min [min,max)
* @param max [min,max)
* @return [min, max)间的一个随机数
*/
public static int randInt(int min, int max) {
return (int) (Math.random() * (max + 1 - min) + min);
}
public static boolean isRepetition(int[] args) {
Set<Object> set = new HashSet<>();
for (int arg : args) set.add(arg);
return set.size() != args.length;
}
}
数据 城市坐标
60,200
180,200
80,180
140,180
20,160
100,160
200,160
140,140
40,120
100,120
180,100
60,80
120,80
180,60
20,40
100,40
200,40
20,20
60,20
160,20