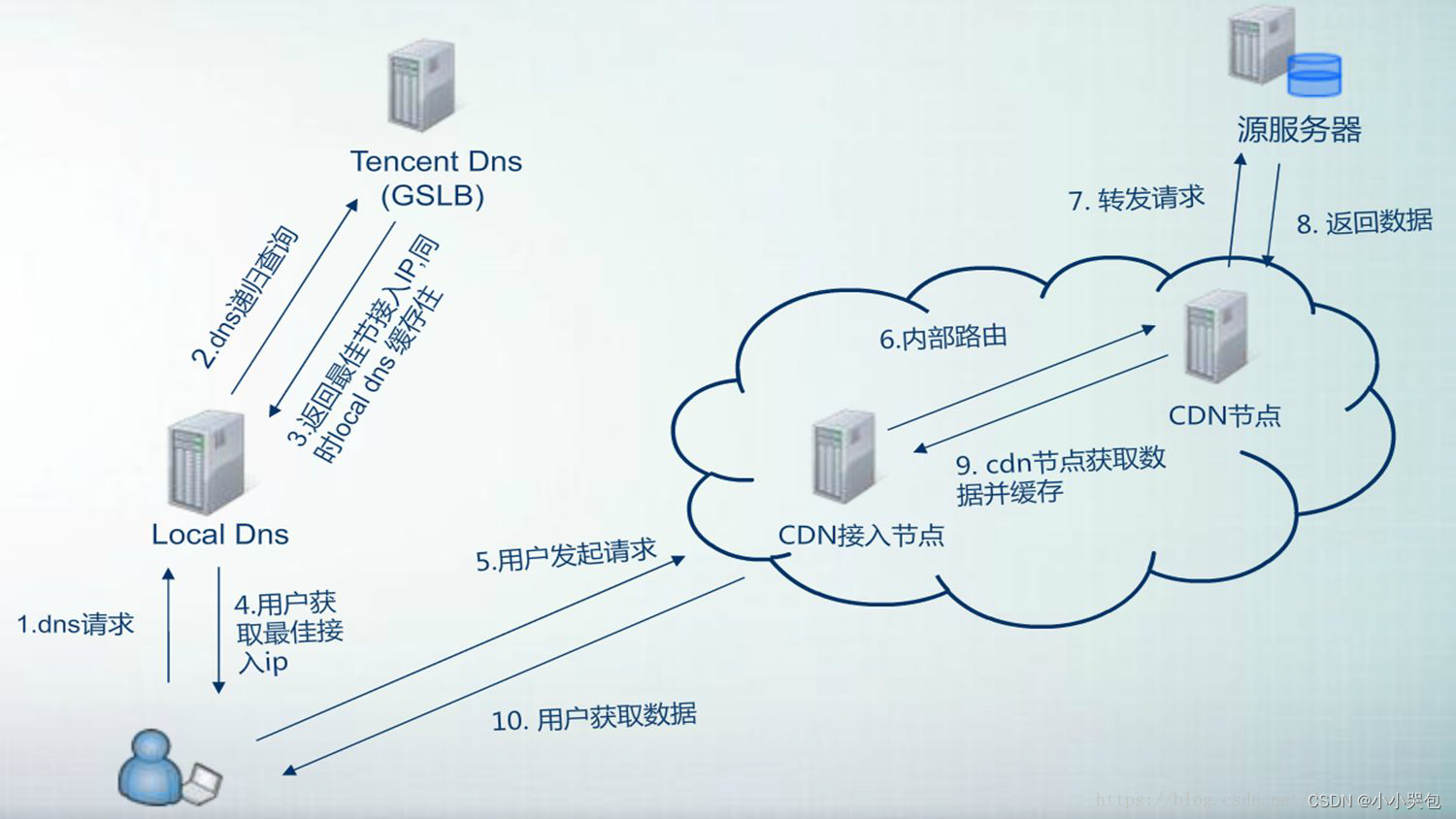
前言
前文近红外数据的预处理和平均(上)提到fNIRS是一种评估氧和脱氧血红蛋白浓度变化的方法,可与fMRI相媲美。fNIRS的不足是它的空间分辨率比fMRI差,但其优点是有更高的时间分辨率,并允许测量无法通过fMRI扫描仪测试的人群的大脑活动。使用多通道近红外记录可以让研究人员考察所发现的与事件相关的大脑活动是局部的(主要在一个通道中发现),还是多个通道中都能观察到。虽然受限于测量的空间分辨率,但是还是可以确定效应的位置。
通常,探测器可以检测来自多个源光电二极管的光。NIRS系统的不同之处在于如何实现这一点:例如,有些系统对每个源光电二极管使用略有不同的源波长,而另一些系统则改变每个源光电二极管的脉冲频率。系统的灵活性也各不相同:有些只允许特定的探测器“观察”特定的光源,另一些则允许更多不同的光源和光电极的组合。有些灵活的系统允许这样的放置,例如源光电极位于x,y位置,而在x+1,y位置放置探测器以及在x+2,y位置放置另一个探测器。这样的排列方式能够使研究人员发现近端、远端效应,即在邻近的源端和探测器之间观察到的效应,以及在较深处的大脑活动,即在距离较远的源端和探测器之间观察到的效应。将短通道分离与正常或较长的通道分离相结合(通道分离是指源和探测器之间的距离)是一种消除皮肤和运动等其他因素伪影的有效方法。在进行这些更复杂的分析类型之前,首先需要熟悉分析的多个通道,这是本文的目标。
多通道近红外数据的预处理和平均
本文将处理由多个通道组成的功能性近红外光谱(fNIRS)数据集。包括读取原始数据,查看设置和数据,结合多通道设置的特定程序对数据进行预处理,并探索使用不同的方法来可视化响应时间和地形图。注意,其他NIRS系统的文件格式与这里使用的不同,本文可能不会详细的讲解不同格式数据的转换,因为这不是本教程的目标,但是这种总体分析结构同样适用于其他近红外系统。
本教程所使用的数据集
本文使用的数据可以从https://download.fieldtriptoolbox.org/tutorial/nirs_multichannel/上获得。对于XML文件,需要右键单击并使用另存为选项。
下载完成之后,你可以在文件夹中看到以下文件:
LR-01-2015-06-01-0002.oxy3LR-02-2015-06-08-0001.oxy3LR-03-2015-06-15-0001.oxy3LR-04-2015-06-17-0001.oxy3LR-05-2015-06-23-0001.oxy3nirs_48ch_layout.matoptodetemplates.xml
Oddball任务
参与者在主动(目标探测)倾听情境下完成事件相关的听觉Oddball范式任务,同时记录左、右颞叶皮层内氧合(HbO2)和脱氧血红蛋白(HbR)的变化。只要有声音出现,就向数据采集系统发送一个TTL脉冲。这种脉冲能够精确地对刺激进行计时,并与fNIRS的反应同步。刺激由标准的1000Hz的音调或偏差的1500Hz的音调组成。刺激间隔为150ms,偏差为连续一列刺激中的第3~12个刺激。
fNIRS测量
fNIRS数据的记录使用了Artinis公司的4个Oxymon系统,这些系统连接起来可以实现48通道的记录。光电纤维的一半(n=16)放置在左颞叶皮层,另一半放置在右颞叶皮层。源极和探测器光电极的距离分别为3cm和1.5cm的深通道和浅通道。采样率降至250Hz。触发器事件记录在ADC通道1(标准)和通道2(偏差)中。

图1.数据记录设置示例图。
分析步骤
根据数据和实验设计的不同,分析可以有许多不同的方式和顺序。本教程的步骤顺序如下(图2):
1、读取连续数据;
2、优先降采样,以减少内存需求;
3、删除不良通道;
4、定义epochs;
5、将光密度转化为含氧血红蛋白(oxyHb)和脱氧血红蛋白(deoxyHb)浓度的变化;
6、将功能响应与系统反应分开,可以使用以下方法其中之一,或者组合使用;
①滤波
②减去参考通道
③每个通道都有反相关的oxyHb/deoxyHb
7、将每种条件下的试次数据进行平均;
8、结果可视化。
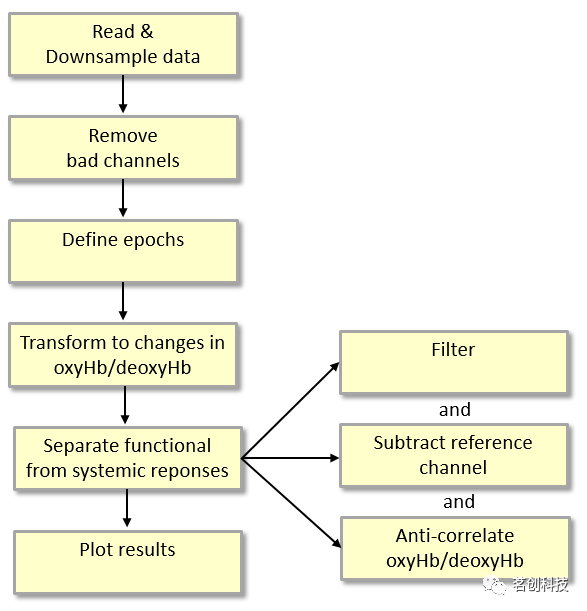
图2.fNIRS分析流程概览。
读取数据并降采样
首先需要将数据读入到MATLAB工作区中,然后执行ft_preprocessing:
cfg = [];cfg.dataset = 'LR-01-2015-06-01-0002.oxy3';data_raw = ft_preprocessing(cfg);
你会在命令窗口中看到如下内容:
processing channel { 'Rx1a-Tx1 [844nm]' ... 'ADC007' 'ADC008' }reading and preprocessingreading and preprocessing trial 1 from 1the call to "ft_preprocessing" took 10 seconds and required the additional allocation of an estimated 732 MB
结构data_raw包含关于实验的所有数据和信息,都存储在单独的字段中。要从如上所示的数据文件中查看通道布局,可以使用:
cfg = [];cfg.opto = 'LR-01-2015-06-01-0002.oxy3';ft_layoutplot(cfg);

图3.通道布局。
检测触发器
对于标准音调刺激的时间在通道ADC001中表示,对于偏差刺激,则在通道ADC002中表示。这些通道可以通过查看label字段找到:
find(strcmp(data_raw.label,'ADC001'))find(strcmp(data_raw.label,'ADC002'))
将ADC001和ADC002的数据绘制成下图,显示模拟电压的TTL脉冲。接下来,当对数据进行分段时,可以使用一个自动化程序来查找这些标记的更改。
figure; hold on% plot the voltage of ADC001 and ADC002% increase the scale of ADC002 a little bit to make it more clear in the figureplot(data_raw.time{1}, data_raw.trial{1}(97,:)*1.0, 'b-')plot(data_raw.time{1}, data_raw.trial{1}(98,:)*1.1, 'r:')
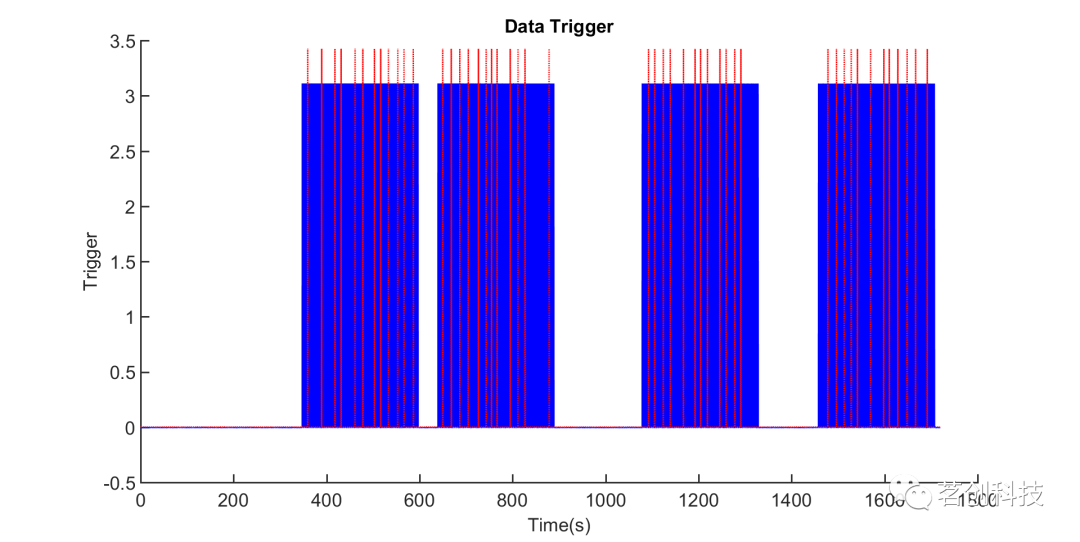
图4.Oddball范式触发器。所有刺激都用蓝线表示。红色虚线表示偏差刺激的开始。
FieldTrip自动检测ADC通道中的起始点,并将ADC通道中向上的一侧表示为事件。
event = ft_read_event('LR-01-2015-06-01-0002.oxy3')接下来,将使用这些事件来定义感兴趣的部分,并从连续数据中剪切出标准试次和偏差试次。如前所述,当前存储的fNIRS数据为250Hz。你可以用以下代码进行查看:
data_raw.fsample为了节省内存并使后续的处理更快,可以使用ft_resampledata将数据降采样至10Hz。
cfg = [];cfg.resamplefs = 10;data_down = ft_resampledata(cfg, data_raw);
在EEG或MEG中,每个试次的片段通常在一秒左右,试次不重叠。但在fNIRS中,需要非常长的片段来进行分析,因为HRF比较缓慢。由于该实验中的听觉刺激相互间的快速跟随,导致实验的数据段重叠。这些重叠的段会影响内存运行效率,因此需要使用降采样。另一种选择是跳过对标准音调的反应的处理,只处理偏差音调(因为目前分析只对偏差数据感兴趣)。
接下来,绘制数据,让我们来看看数据是什么样子。在cfg.preproc中,可以为动态预处理指定一些选项。这里,将对数据进行去均值处理,即减去平均值。在cfg.preproc中指定的选项与ft_preprocessing中的选项基本相同,不同的是,在当前指令ft_databrowser中,去均值化仅用于绘图,数据本身保持不变。
cfg = [];cfg.preproc.demean = 'yes';cfg.viewmode = 'vertical';cfg.continuous = 'no';cfg.ylim = [ -0.003 0.003 ];cfg.channel = 'Rx*'; % only show channels starting with Rxft_databrowser(cfg, data_down);

图5.高通滤波前降采样数据的光密度轨迹。
这...太噪了!但不要放弃,还有拯救的机会。在接下来的步骤中,可以去除大部分噪声。由于我们对血流动力学响应中非常缓慢的变化也不感兴趣,所以可以通过高通滤波安全地去除低频信息。
cfg = [];cfg.hpfilter = 'yes';cfg.hpfreq = 0.01;data_flt = ft_preprocessing(cfg,data_down);
这一步骤消除了通道间血流动力学响应的一些可变性。接下来,绘制过滤后的数据,看看改善情况。
cfg = [];cfg.preproc.demean = 'yes';cfg.viewmode = 'vertical';cfg.continuous = 'no';cfg.ylim = [ -0.003 0.003 ];cfg.channel = 'Rx*'; % only show channels starting with Rxft_databrowser(cfg, data_flt);
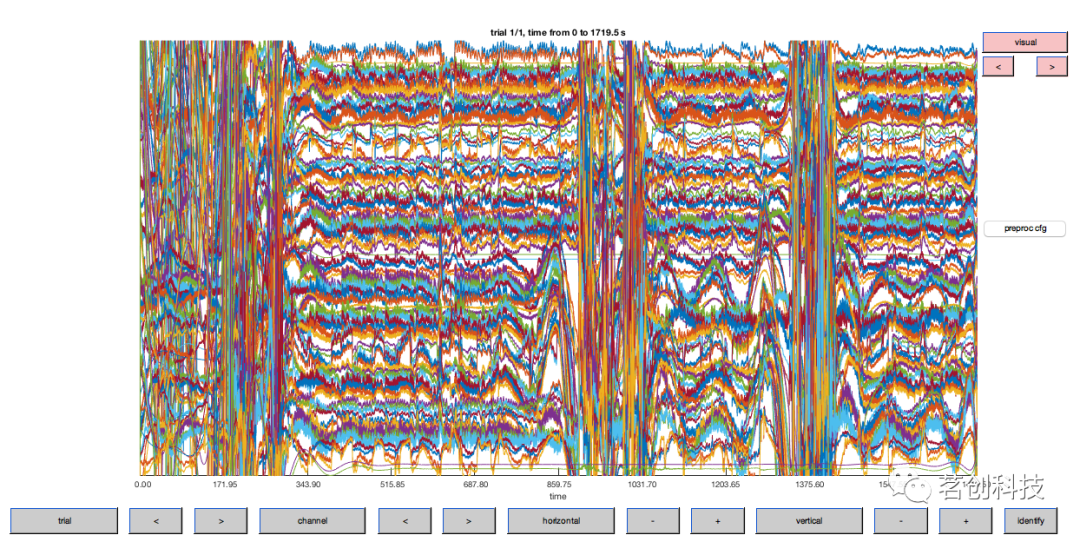
图6.高通滤波后降采样数据的光密度轨迹。对比图5可以看到,DC(偏移量)已经被大大消除了。
分段
对数据进行分段,以获得感兴趣的时间段,然后再进一步处理数据。在该实验中,兴趣段是刺激开始前5s到刺激后20s之间的一段时间。可以使用ft_redefinetrial函数删除数据中的片段,和使用ft_definetrial来确定段。
event = ft_read_event('LR-01-2015-06-01-0002.oxy3');adc001 = find(strcmp({event.type}, 'ADC001'));adc002 = find(strcmp({event.type}, 'ADC002'));% get the sample number in the original data% note that we transpose them to get columnssmp001 = [event(adc001).sample]';smp002 = [event(adc002).sample]';factor = data_raw.fsample / data_down.fsample% get the sample number after downsamplingsmp001 = round((smp001-1)/factor + 1);smp002 = round((smp002-1)/factor + 1);pre = round( 5*data_down.fsample);post = round(20*data_down.fsample);offset = -pre; % see ft_definetrialtrl001 = [smp001-pre smp001+post];trl002 = [smp002-pre smp002+post];% add the offsettrl001(:,3) = offset;trl002(:,3) = offset;trl001(:,4) = 1; % add a column with the condition numbertrl002(:,4) = 2; % add a column with the condition number% concatenate the two conditions and sort themtrl = sortrows([trl001; trl002])% remove trials that stretch beyond the end of the recordingsel = trl(:,2)<size(data_down.trial{1},2);trl = trl(sel,:);cfg = [];cfg.trl = trl;data_epoch = ft_redefinetrial(cfg,data_down);
如果输入data_epoch,应该会在命令窗口中看到这个:
data_epoch =struct with fieldhdr: [1x1 struct]trial: {1x597 cell}time: {1x597 cell}fsample: 10label: {104x1 cell}opto: [1x1 struct]trialinfo: [597x1 double]sampleinfo: [597x2 double]cfg: [1x1 struct]
值得注意的是,trial和time字段现在都有1x597个单元阵列。这与呈现的597种刺激相对应。在data_epoch.trialinfo中:存储有关刺激类型的信息(事件1或事件2)。因此,可以找到哪个单元是第一个偏差刺激:
idx = find(data_epoch.trialinfo==2, 1, 'first')运行以上行代码,将得到:
idx =8
让我们通过绘制平均光密度图来看看第一个偏差附近的情况:
cfg = [];cfg.channel = 'Rx*';cfg.trials = 8;cfg.baseline = 'yes';ft_singleplotER(cfg, data_epoch)
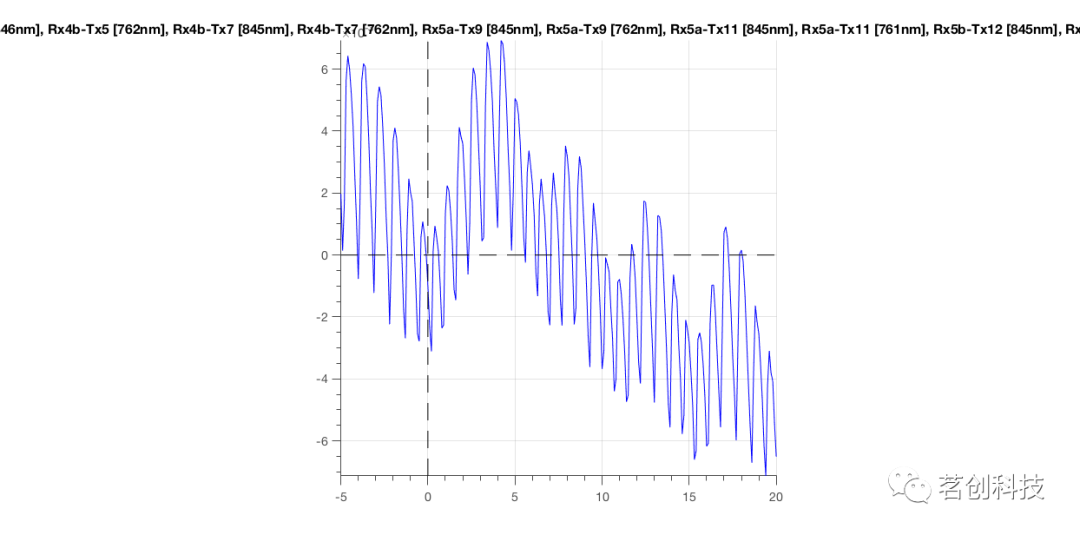
图7.分段后,第一个偏差刺激点周围的光密度数据。
删除不良通道
首先移除与头皮接触不良、因此产生不良信号的通道。从光密度轨迹可以估计光电二极管和头皮之间的耦合是否良好,因为来自每个光电二极管的两个信号(对应于两个波长)应该有一个正相关的心跳。如果相关性很小或为负,那么将在进一步处理中排除该光电极。这一步可以在ft_nirs_scalpcouplingindex中实现。
cfg = [];data_sci = ft_nirs_scalpcouplingindex(cfg, data_epoch);
可以看到在data_sci.label中删除了一些通道,现在只有86个标签,而不是104个:
data_sci =struct with fieldhdr: [1x1 struct]trial: {1x597 cell}time: {1x597 cell}fsample: 10label: {86x1 cell}opto: [1x1 struct]trialinfo: [597x1 double]sampleinfo: [597x2 double]cfg: [1x1 struct]
删除伪影
这里已经通过分段去除了主要的运动伪影,并删除了耦合不良的光电极。因此,对于该数据集,可以忽略此步骤。
将光密度转化为氧和脱氧血红蛋白浓度的变化
通过ft_nirs_transform_ODs将光密度转换为含氧和脱氧血红蛋白的浓度。
cfg = [];cfg.target = {'O2Hb', 'HHb'};cfg.channel = 'nirs'; % e.g., one channel incl. wildcards, you can also use ?all? to select all NIRS channelsdata_conc = ft_nirs_transform_ODs(cfg, data_sci);
使用ft_singleplotER再次检查数据。你可以在信号中看到一个明显的心跳。

图8.血红蛋白浓度随时间的变化。
将功能响应与系统反应分开
低通滤波
心跳并不是该数据集对应的研究中感兴趣的信号,所以将使用低通滤波对数据进行过滤,使其低于心跳频率(约1Hz)。
cfg = [];cfg.lpfilter = 'yes';cfg.lpfreq = 0.8;data_lpf = ft_preprocessing(cfg, data_conc);
这里的平均浓度变化是一个完美的血液动力学响应例子。信号在刺激开始时上升,在4s左右达到峰值,然后再次下降。

图9.低通滤波后的血红蛋白浓度。
绘制结果
首先,运行ft_timelockanalysis计算均值。ft_timelockanalysis默认对所有试次进行平均。若想对偏差试次和标准试次分别进行平均,需要告诉代码哪些试次属于标准刺激哪些属于偏差刺激。有关条件的信息存储在trialinfo中,其中1表示标准,2表示偏差。
cfg = [];cfg.trials = find(data_lpf.trialinfo(:,1) == 1);timelockSTD = ft_timelockanalysis(cfg, data_lpf);
然后,使用ft_timelockbaseline进行基线校正。刺激前5s将被用作基准的时间窗。
cfg = [];cfg.baseline = [-5 0];timelockSTD = ft_timelockbaseline(cfg, timelockSTD);
对偏差刺激执行同样的步骤。
cfg = [];cfg.trials = find(data_lpf.trialinfo(:,1) == 2);timelockDEV = ft_timelockanalysis(cfg, data_lpf);cfg = [];cfg.baseline = [-5 0];timelockDEV = ft_timelockbaseline(cfg, timelockDEV);
该通道布局可以使用MATLAB函数load读取nirs_48ch_layout.mat文件。该文件包含一个名为lay的结构。
load('nirs_48ch_layout.mat')figure; ft_plot_layout(lay) % note that O2Hb and HHb channels fall on top of each other
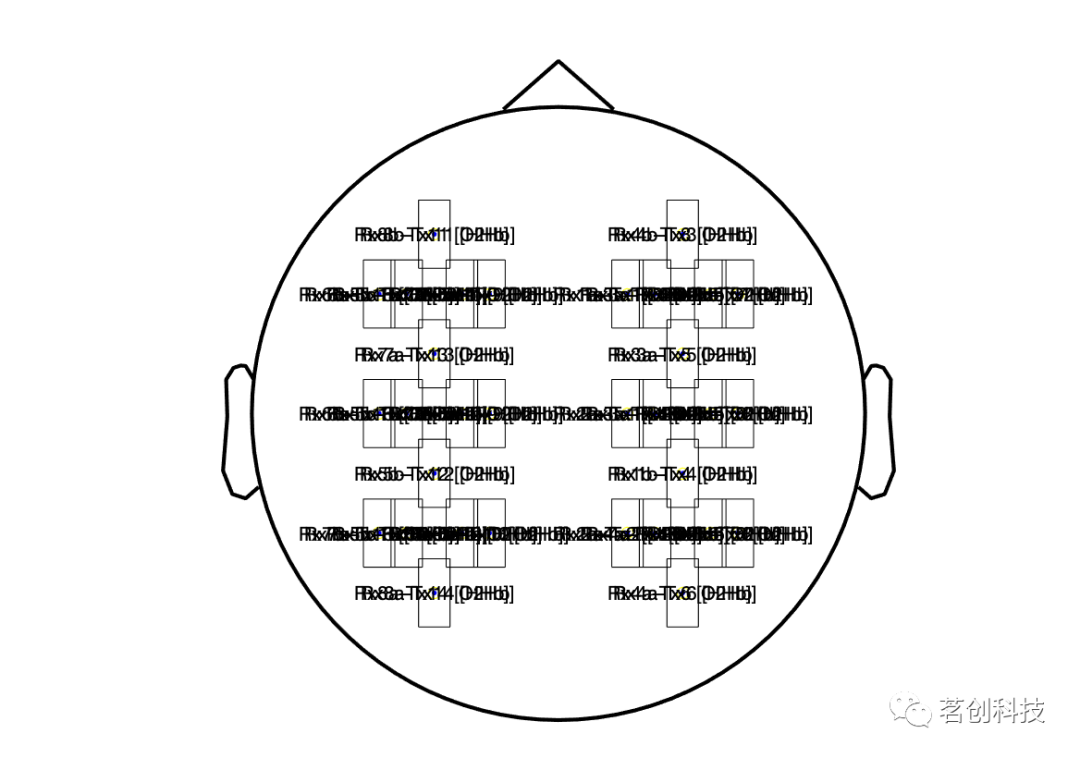
图10.通道布局。
有许多FieldTrip选项可用于结果可视化,例如ft_singleplotER(ER表示事件相关),该函数允许绘制单个通道,以及ft_multiplotER,该函数允许绘制多个通道。ft_multiplotER还可以在交互模式下使用,以选择感兴趣的数据片段(例如选择特定的通道和特定的时间窗)。
但值得注意的是,要运行ft_multiplotER,需要使用cfg.layout = lay将FieldTrip指向布局结构。
cfg = [];cfg.showlabels = 'yes';cfg.layout = lay; % you could also specify the name of the mat filecfg.interactive = 'yes';cfg.linecolor = 'rb';cfg.colorgroups(contains(timelockDEV.label, 'O2Hb')) = 1; % these will be redcfg.colorgroups(contains(timelockDEV.label, 'HHb')) = 2; % these will be blueft_multiplotER(cfg, timelockDEV);
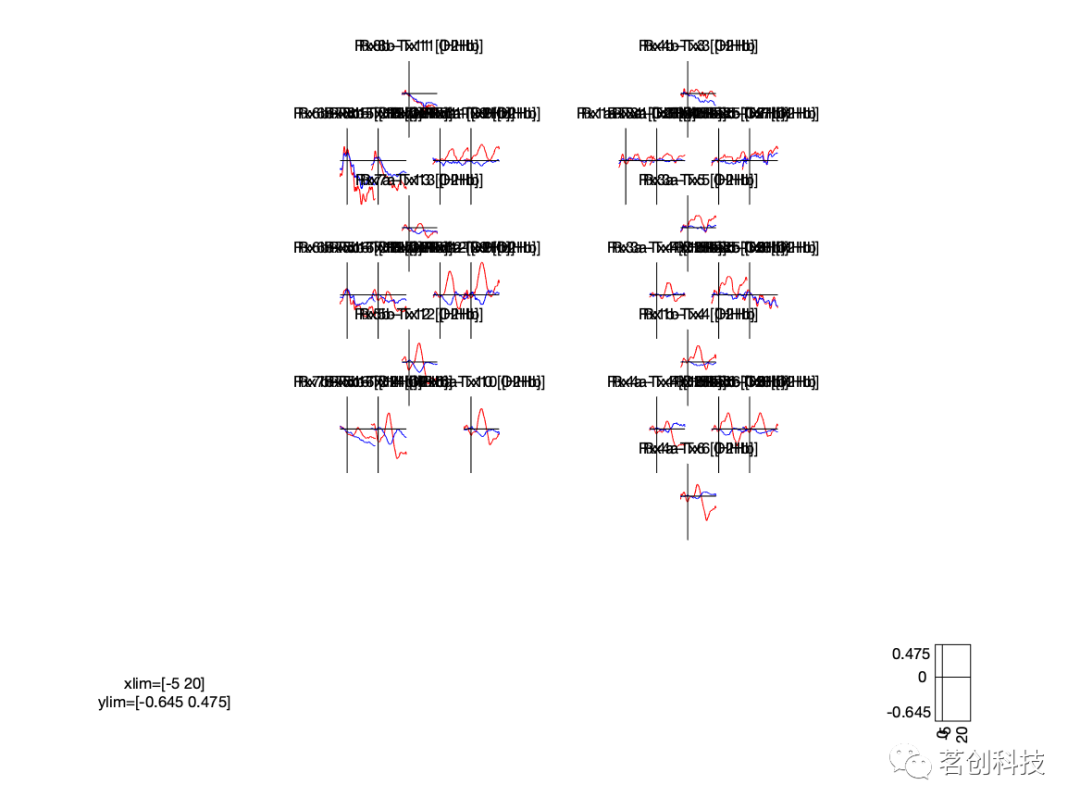
图11.每个通道的平均时间过程。
此外,还可以在某个时间点或时间窗上生成信号的空间地形图。时间窗可以通过cfg.xlim = [5 7];命令进行设置,响应强度的范围可以设置为-0.2到0.2,但这取决于你研究中的数据情况:例如,许多fNIRS研究人员使用block设计,根据block持续时间,响应可能具有更大的振幅。
cfg = [];cfg.layout = lay; % you could also specify the name of the mat filecfg.marker = 'labels';cfg.xlim = [5 7];cfg.zlim = [-0.2 0.2];cfg.channel = '* [O2Hb]';figure; subplot(1,2,1);ft_topoplotER(cfg, timelockDEV);title('[O2Hb]');cfg.channel = '* [HHb]';subplot(1,2,2);ft_topoplotER(cfg, timelockDEV);title('[HHb]');
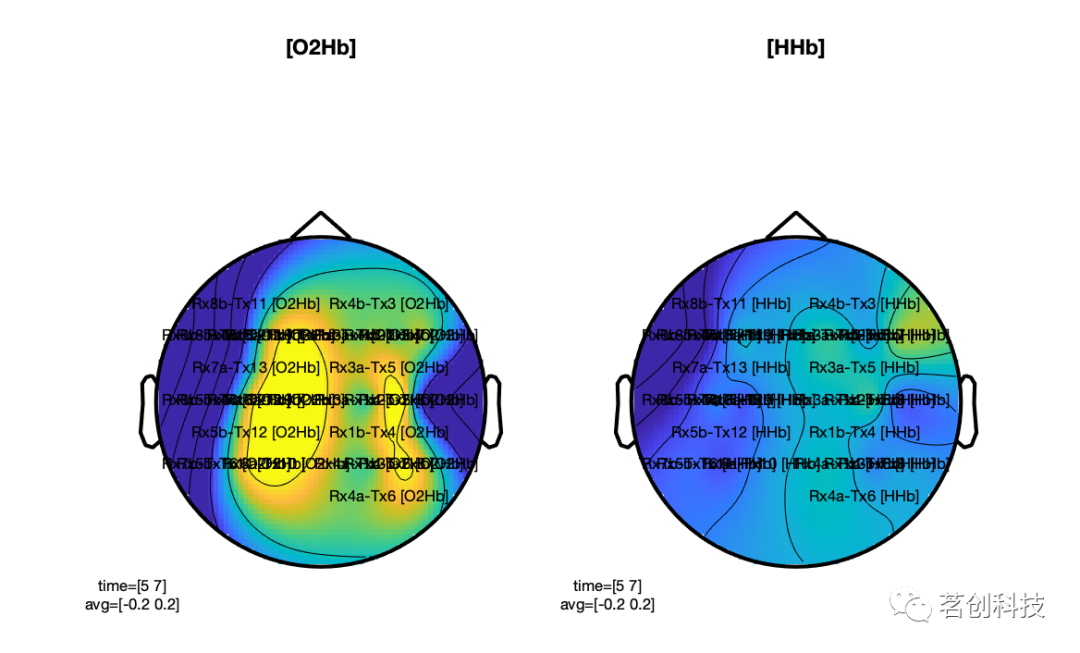
图12.偏差刺激的含氧和脱氧血红蛋白信号变化的地形图。
参考来源:
Preprocessing and averaging of multi-channel NIRS data.
https://www.fieldtriptoolbox.org/tutorial/nirs_multichannel/
https://download.fieldtriptoolbox.org/tutorial/nirs_multichannel/